
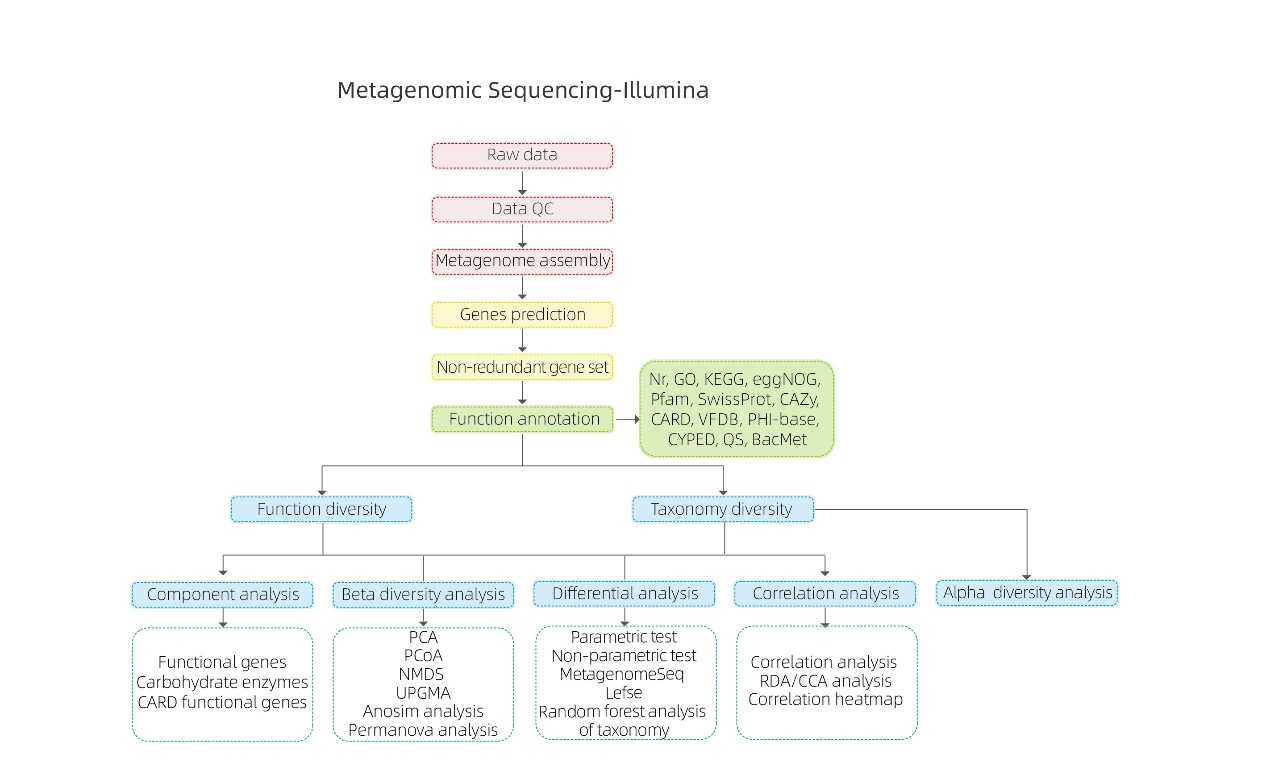
Metagenomische sequencing-NGS
Servicevoordelen
● Isolatie en teeltvrij voor profilering van microbiële gemeenschappen
● Hoge resolutie voor het detecteren van soorten met een lage dichtheid in milieumonsters
● Het idee van “meta-” integreert alle biologische kenmerken op functioneel niveau, soortniveau en genniveau, wat een dynamische visie weerspiegelt die dichter bij de werkelijkheid staat.
● BMK heeft een enorme ervaring opgebouwd met diverse soorten monsters, met meer dan 10.000 verwerkte monsters.
Servicespecificaties
| Platform | Volgorde aanbrengen in | Aanbevolen gegevens | Doorlooptijd |
| Illumina NovaSeq-platform | PE150 | 6G/10G/20G | 45 werkdagen |
Bio-informatica analyses
● Kwaliteitscontrole van ruwe gegevens
● Metagenoomassemblage
● Niet-redundante genenset en annotatie
● Analyse van de soortendiversiteit
● Analyse van genetische functiediversiteit
● Intergroepsanalyse
● Associatieanalyse tegen experimentele factoren
Monstervereisten en levering
Voorbeeldvereisten:
VoorDNA-extracten:
| Voorbeeldtype | Hoeveelheid | Concentratie | Puurheid |
| DNA-extracten | > 30 ng | > 1 ng/μl | OD260/280= 1,6-2,5 |
Voor milieumonsters:
| Voorbeeldtype | Aanbevolen bemonsteringsprocedure |
| Bodem | Monsterhoeveelheid: ca.5 gram;De resterende verdorde substantie moet van het oppervlak worden verwijderd;Maal grote stukken en passeer door een filter van 2 mm;Aliquot-monsters in steriele EP-buis of cyrotube voor reservering. |
| Ontlasting | Monsterhoeveelheid: ca.5 gram;Verzamel en aliquoteer monsters in een steriele EP-buis of cryobuis voor reservering. |
| Darminhoud | Monsters moeten onder aseptische omstandigheden worden verwerkt.Was het verzamelde weefsel met PBS;Centrifugeer de PBS en verzamel het neerslagmiddel in EP-buizen. |
| Slib | Monsterhoeveelheid: ca.5 gram;Verzamel het slibmonster en verdeel het in een steriele EP-buis of cryobuis ter reservering |
| Waterlichaam | Voor monsters met een beperkte hoeveelheid microbiële stoffen, zoals kraanwater, bronwater enz., verzamel ten minste 1 liter water en voer dit door een filter van 0,22 μm om de microbiële stoffen op het membraan te verrijken.Bewaar het membraan in een steriele buis. |
| Huid | Schraap het huidoppervlak voorzichtig af met een steriel wattenstaafje of chirurgisch mesje en plaats het in een steriele buis. |
Aanbevolen monsterlevering
Bevries de monsters in vloeibare stikstof gedurende 3-4 uur en bewaar ze in vloeibare stikstof of -80 graden tot langetermijnreservering.Verzending van monsters met droogijs is vereist.
Servicewerkstroom

Levering van monsters

Bouw van bibliotheek

Volgorde aanbrengen in

Gegevensanalyse

After-sales diensten
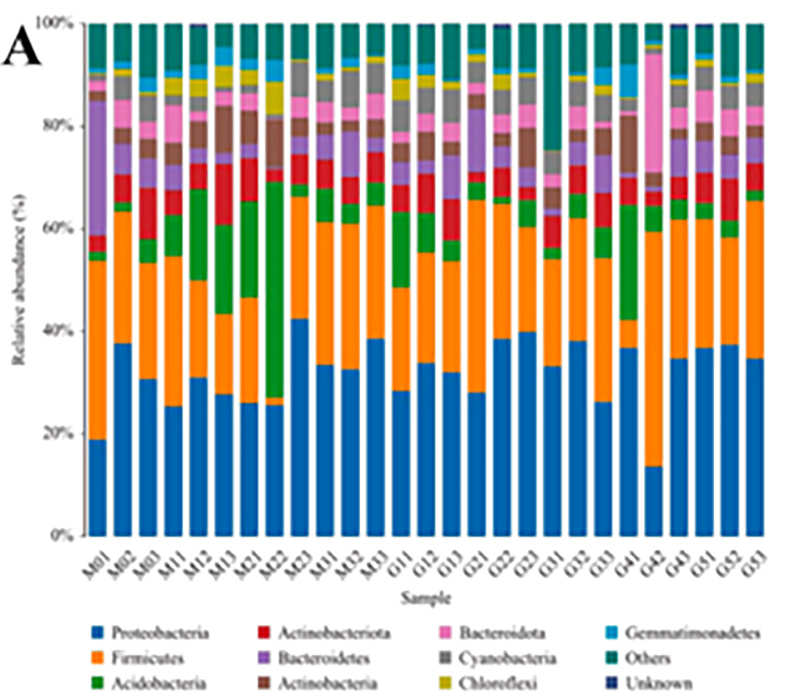
1.Histogram: verspreiding van soorten
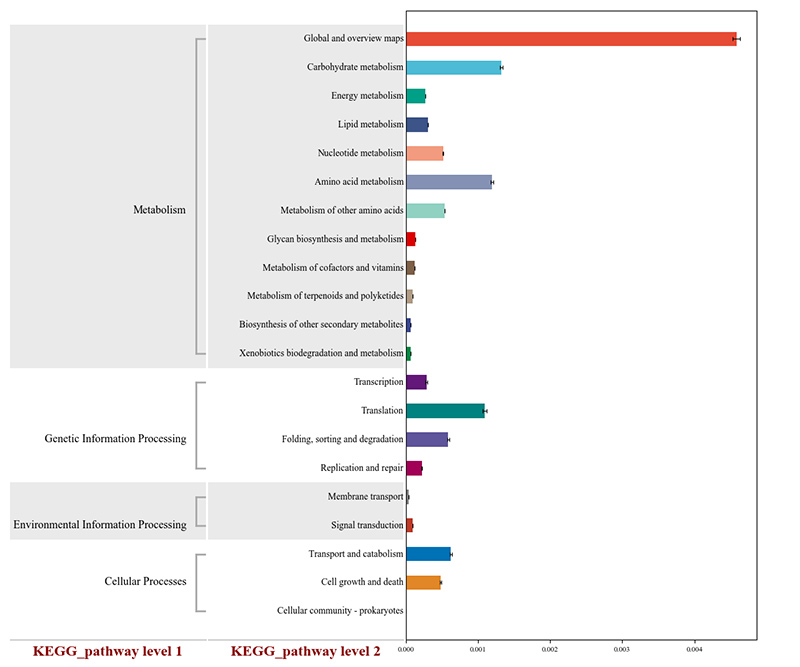
2. Functionele genen geannoteerd voor KEGG-metabolische routes
3. Warmtekaart: differentiële functies gebaseerd op relatieve genenabundantie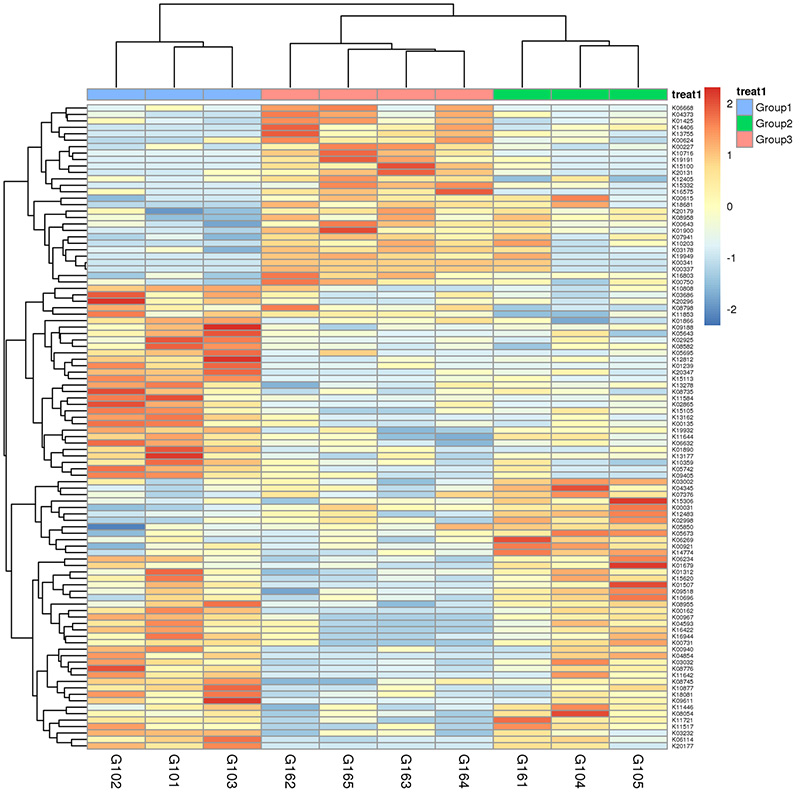 4. Circos van CARD-antibioticaresistentiegenen
4. Circos van CARD-antibioticaresistentiegenen
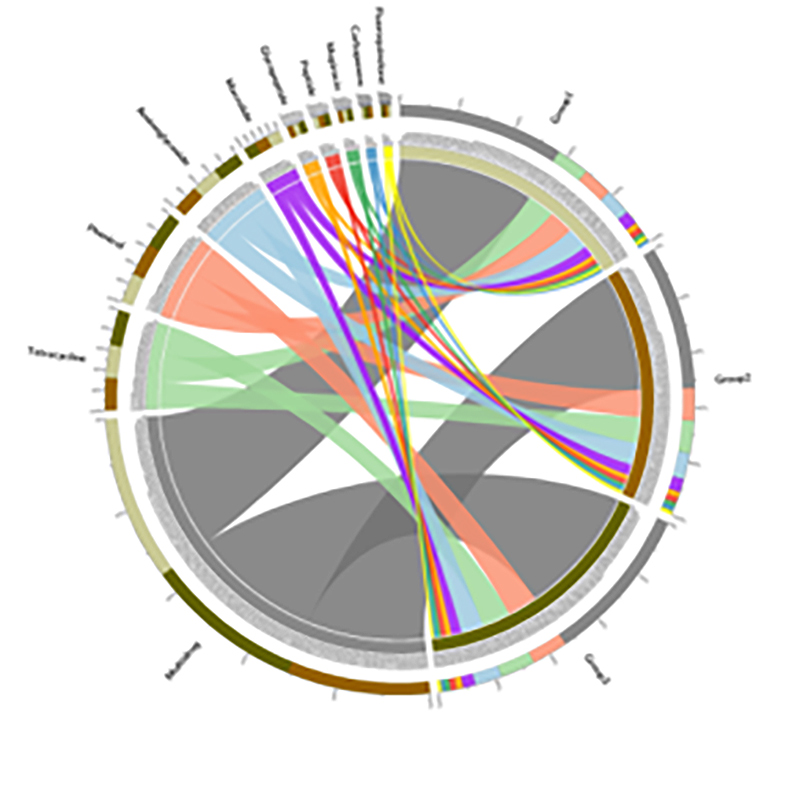
BMK-zaak
Prevalentie van antibioticaresistentiegenen en bacteriële pathogenen langs het bodem-mangrovewortelcontinuüm
Gepubliceerd:Tijdschrift voor gevaarlijke materialen, 2021
Sequentiestrategie:
Materialen: DNA-extracten van vier fragmenten van met mangrovewortel geassocieerde monsters: ongeplante grond, rhizosfeer-, episfeer- en endosfeercompartimenten
Platform: Illumina HiSeq 2500
Doelstellingen: Metagenoom
16S rRNA-gen V3-V4-regio
Belangrijkste resultaten
Metagenomische sequencing en metabarcoding-profilering op het bodem-wortelcontinuüm van mangroveboompjes werden verwerkt om de verspreiding van antibioticaresistentiegenen (ARG's) vanuit de bodem naar planten te bestuderen.Metagenomische gegevens onthulden dat 91,4% van de antibioticaresistentiegenen algemeen werden geïdentificeerd in alle vier bovengenoemde bodemcompartimenten, wat een continu patroon vertoonde.16S rRNA-ampliconsequencing genereerde 29.285 sequenties, die 346 soorten vertegenwoordigen.In combinatie met soortprofilering door middel van ampliconsequencing bleek deze verspreiding onafhankelijk te zijn van wortelgeassocieerde microbiota, maar deze zou kunnen worden vergemakkelijkt door mobiele genetische elementen.Deze studie identificeerde de stroom van ARG's en ziekteverwekkers van de bodem naar de planten via een onderling verbonden bodem-wortelcontinuüm.
Referentie
Wang, C., Hu, R., Strong, PJ, Zhuang, W., en Shu, L..(2020).Prevalentie van antibioticaresistentiegenen en bacteriële pathogenen langs het bodem-mangrovewortelcontinuüm.Tijdschrift voor gevaarlijke materialen, 408, 124985.











