
Metagenomiskā sekvencēšana -NGS
Pakalpojuma priekšrocības
● Bez izolācijas un kultivēšanas mikrobu kopienas profilēšanai
● Augsta izšķirtspēja, nosakot sugas ar zemu sastopamību vides paraugos
● “Meta-” ideja integrē visas bioloģiskās pazīmes funkcionālā līmenī, sugas un gēnu līmenī, kas atspoguļo dinamisku skatījumu, kas ir tuvāks realitātei.
● BMK uzkrāj milzīgu pieredzi dažādu paraugu veidošanā ar vairāk nekā 10 000 apstrādātu paraugu.
Pakalpojuma specifikācijas
| Platforma | Secība | Ieteicamie dati | Apgrozījuma laiks |
| Illumina NovaSeq platforma | PE150 | 6 G/10 G/20 G | 45 darba dienas |
Bioinformātikas analīzes
● Neapstrādātu datu kvalitātes kontrole
● Metagenomu montāža
● Nelieka gēnu kopa un anotācija
● Sugu daudzveidības analīze
● Ģenētisko funkciju daudzveidības analīze
● Starpgrupu analīze
● Asociāciju analīze pret eksperimentāliem faktoriem
Prasību paraugs un piegāde
Prasību paraugs:
PriekšDNS ekstrakti:
| Parauga veids | Summa | Koncentrēšanās | Tīrība |
| DNS ekstrakti | > 30 ng | > 1 ng/μl | OD260/280= 1,6-2,5 |
Vides paraugiem:
| Parauga veids | Ieteicamā paraugu ņemšanas procedūra |
| Augsne | Paraugu ņemšanas apjoms: apm.5 g;Atlikušo nokaltušo vielu nepieciešams noņemt no virsmas;Sasmalcina lielus gabalus un izlaiž cauri 2 mm filtram;Paraugu alikvotā daļa rezervēšanai sterilā EP mēģenē vai cirotūbiņā. |
| Izkārnījumi | Paraugu ņemšanas apjoms: apm.5 g;Savākt un alikvot paraugus sterilā EP mēģenē vai kriomēģenē rezervēšanai. |
| Zarnu saturs | Paraugi jāapstrādā aseptiskos apstākļos.Nomazgājiet savāktos audus ar PBS;Centrifugējiet PBS un savāciet nogulsnes EP mēģenēs. |
| Dūņas | Paraugu ņemšanas apjoms: apm.5 g;Savāc un alikvotā dūņu paraugu sadala sterilā EP mēģenē vai kriomēģenē rezervēšanai |
| Ūdenstilpne | Paraugam ar ierobežotu mikrobu daudzumu, piemēram, krāna ūdeni, akas ūdeni utt., Savākt vismaz 1 l ūdens un izlaist cauri 0,22 μm filtram, lai bagātinātu membrānas mikroorganismus.Uzglabājiet membrānu sterilā mēģenē. |
| Āda | Uzmanīgi nokasiet ādas virsmu ar sterilu vates tamponu vai ķirurģisko asmeni un ievietojiet to sterilā mēģenē. |
Ieteicamā paraugu piegāde
Sasaldējiet paraugus šķidrā slāpeklī 3-4 stundas un uzglabājiet šķidrā slāpeklī vai -80 grādos līdz ilgstošai rezervēšanai.Nepieciešama paraugu piegāde ar sauso ledu.
Pakalpojuma darba plūsma

Paraugu piegāde

Bibliotēkas celtniecība

Secība

Datu analīze

Pēcpārdošanas pakalpojumi
1.Histogramma: sugu izplatība
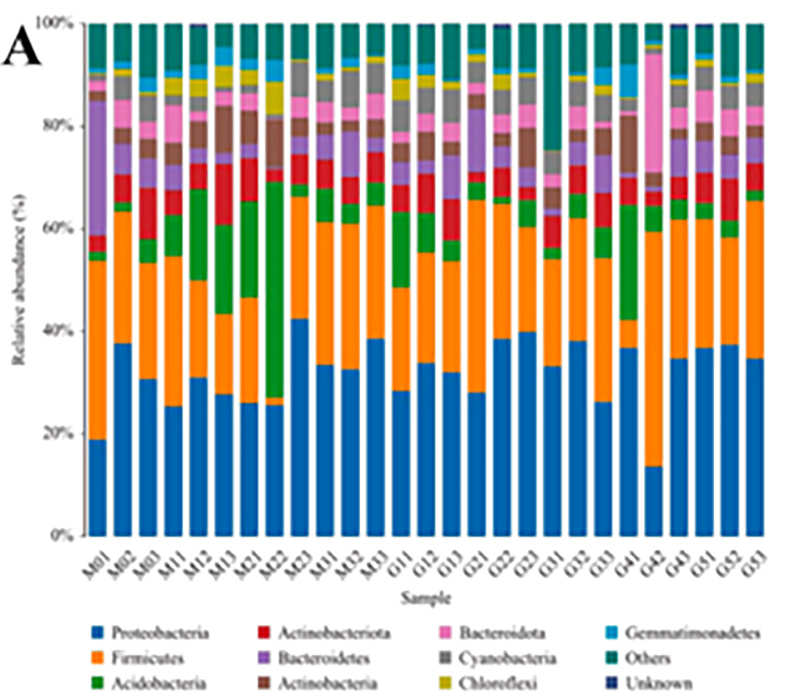
2. Funkcionālie gēni, kas anotēti uz KEGG vielmaiņas ceļiem
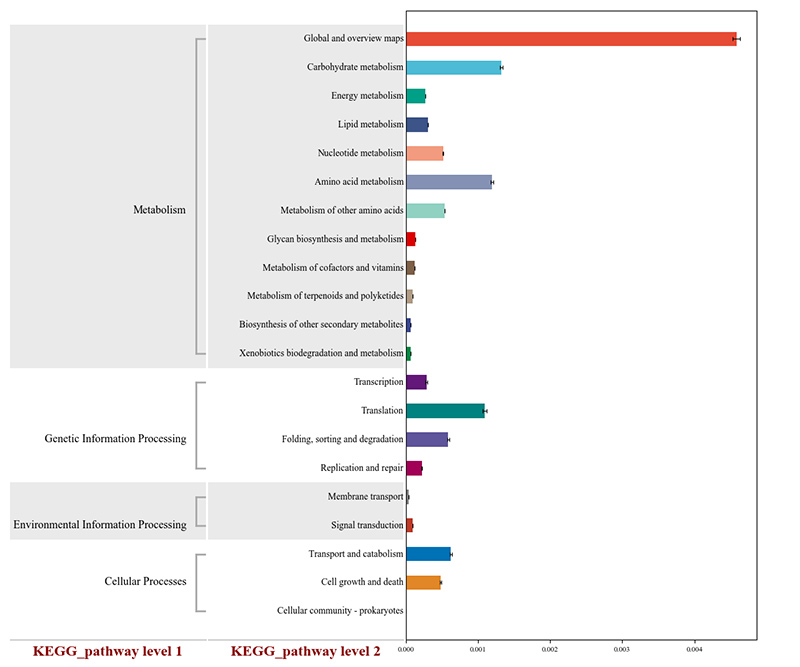
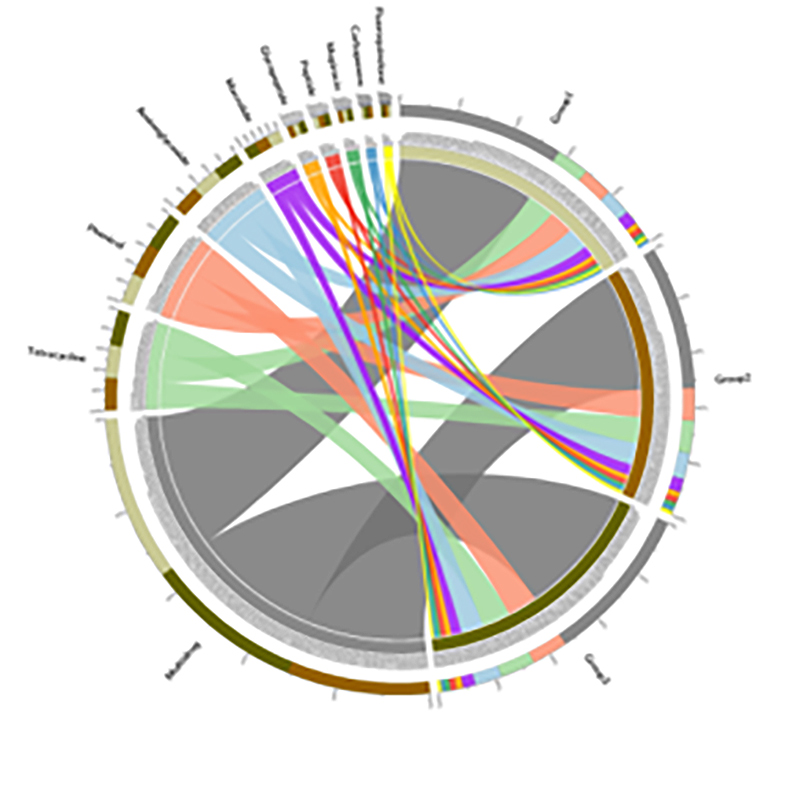
3. Siltuma karte: Diferenciālās funkcijas, kuru pamatā ir relatīvais gēnu daudzums 4. CARD antibiotiku rezistences gēnu cirki
4. CARD antibiotiku rezistences gēnu cirki
BMK lieta
Antibiotiku rezistences gēnu un baktēriju patogēnu izplatība augsnes-mangrovju sakņu kontinuumā
Publicēts:Bīstamo materiālu žurnāls, 2021. gads
Secības noteikšanas stratēģija:
Materiāli: DNS ekstrakti no četriem ar mangrovju saknēm saistīto paraugu fragmentiem: neapstādīta augsne, rizosfēra, episfēras un endosfēras nodalījumi
Platforma: Illumina HiSeq 2500
Mērķi: metagenoms
16S rRNS gēna V3-V4 reģions
Galvenie rezultāti
Lai izpētītu antibiotiku rezistences gēnu (ARG) izplatīšanos no augsnes augos, tika apstrādāta metagenomiskā sekvencēšana un metabarkodēšanas profilēšana mangrovju stādu augsnes-sakņu kontinuumā.Metagenomiskie dati atklāja, ka 91, 4% antibiotiku rezistences gēnu parasti tika identificēti visos četros iepriekšminētajos augsnes nodalījumos, kas liecināja par nepārtrauktu modi.16S rRNS amplikonu sekvencēšana radīja 29 285 sekvences, kas pārstāv 346 sugas.Apvienojumā ar sugu profilēšanu, izmantojot amplikonu sekvencēšanu, tika konstatēts, ka šī izplatīšana nav atkarīga no ar saknēm saistītās mikrobiotas, tomēr to varētu veicināt ģenētisko elementu mobilie.Šis pētījums identificēja ARG un patogēnu plūsmu no augsnes augos, izmantojot savstarpēji saistītu augsnes-sakņu kontinuumu.
Atsauce
Wang, C., Hu, R., Strong, PJ, Zhuang, W., & Shu, L..(2020).Antibiotiku rezistences gēnu un baktēriju patogēnu izplatība augsnes un mangrovju sakņu kontinuumā.Bīstamo materiālu žurnāls, 408, 124985.











