MIKROBIOLOGIA

Zgodność bakteryjna i immobilizacja z biowęglem poprawiają degradację tebukonazolu, skład i funkcjonowanie mikrobiomu gleby
Sekwencjonowanie pełnej długości amplikonu 16S |PacBio HiFi |Różnorodność alfa |Różnorodność beta
W tym badaniu pełnej długości sekwencjonowanie amplikonu 16S metodą PacBio i analizę bioinformatyczną wykonała firma Biomarker Technologies.
Przegląd najważniejszych wydarzeń
Bakterie rozkładające tebukonazol unieruchomione na biowęglu Alcaligenes faecalis WZ-2 badano pod kątem wydajności biodegradacji i wpływu na glebę skażoną tebukonazolem w porównaniu z wolno rozkładającym się szczepem WZ-2.
1. Unieruchomiony biowęglem WZ-2 wykazał bardziej efektywną degradację w tebukonazolu w porównaniu z wolnym WZ-2 poprzez skrócenie okresu półtrwania tebukonazolu w glebie z 18,7 dni do 13,3 dni.
2. Unieruchomione na biowęglu WZ-2 były w stanie przywrócić aktywność enzymów natywnych drobnoustrojów glebowych, w tym ureazy, dehydrogenazy i inwertazy itp.
3. Profil mikrobiologiczny w glebie traktowanej WZ-2 unieruchomionym biowęglem, określony za pomocą sekwencjonowania 16S pełnej długości, zdecydowanie potwierdził, że system ten może przywrócić zdrowie gleby poprzez poprawę struktury społeczności bakteryjnych pod wpływem skażenia tebukonazolem.
Eksperyment (związany z sekwencjonowaniem)
Grupowanie: CK: Gleba naturalna;T: Gleba wzbogacona tebukonazolem;S: Tebukonazol zawierał glebę z wolnym szczepem WZ-2;BC: Tebukonazol zawierał glebę z biowęglem;BCS: Tebukonazol zawierał glebę z unieruchomionym biowęglem WZ-2.
Pobieranie próbek: Ekstrakcja całkowitego DNA gleby amplifikowana starterami 16S rDNA
27F (5′-AGAGTTTTGATCCTGGCTCAG-3′) i 1492R (5′-GGTTACCTTGTTACGA),reprezentujący pełnej długości 16S rDNA
Platforma sekwencjonowania: PacBio RS II
Strategia sekwencjonowania: odczyt CCS HIFI
Analiza danych:BMKCloudPlatformy bioinformatyczne czynią ze złotych rybek doskonały system modeli genetycznych dla fizjologii i ewolucji ryb.
Wynik
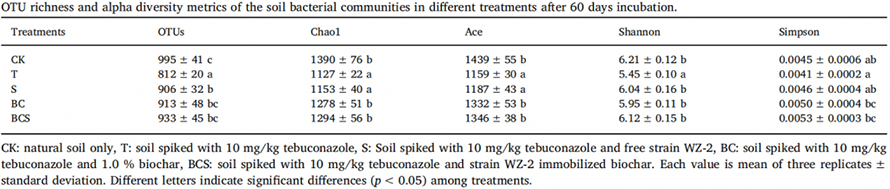
Strukturę zbiorowisk drobnoustrojów w glebie określono za pomocą sekwencjonowania 16S rDNA.Oceniono bogactwo OTU i wskaźnik różnorodności alfa, w tym wskaźniki Chao1, Ace, Shannona i Simpsona, aby ujawnić różnorodność gatunkową w każdym systemie.Po 60 dniach inkubacji wszystkie wskaźniki wykazały podobny trend, tj. tebukonazol mógł powodować zmniejszenie bogactwa gatunkowego i różnorodności gleby.Jednakże dodanie szczepu WZ-2 umożliwiło częściowe odzyskanie zbiorowiska bakterii glebowych zarówno pod względem bogactwa, jak i różnorodności.Zaobserwowano ograniczone różnice między BC, BCS i CK, co wskazuje, że biowęgiel i WZ-2 unieruchomiony biowęglem mogą skutecznie przywracać zdrowie biologiczne gleby.
W tym badaniu zastosowano metodę nieważonych grup par ze średnimi arytmetycznymi (UPGMA), aby wykazać zróżnicowanie beta między grupami.Jak pokazano na poniższym rysunku, BC, BSC i CK mają bardziej podobny wzór składu mikrobiologicznego w porównaniu z grupami T i S, co dodatkowo wskazuje, że wprowadzenie biowęgla do bioremediacji gleby skażonej tebukonazolem może w dużym stopniu ułatwić odbudowę społeczności drobnoustrojów w glebie.
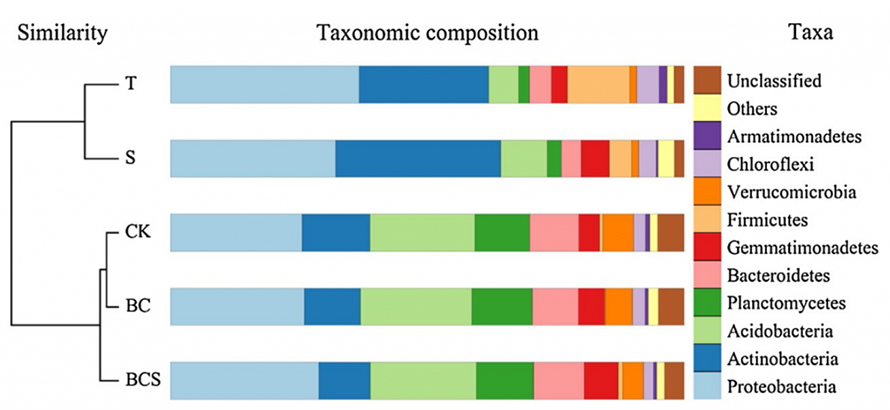
Postać.Grupowanie UPGMA społeczności bakteryjnej na poziomie gromady pod różnym leczeniem
Odniesienie
Sun, Tong i in.„Zgodność bakteryjna i immobilizacja z biowęglem poprawiają degradację tebukonazolu, skład i funkcjonowanie mikrobiomu gleby”.Dziennik materiałów niebezpiecznych398 (2020): 122941.
Wiadomości i najważniejsze informacje ma na celu podzielenie się najnowszymi pomyślnymi przypadkami z firmą Biomarker Technologies, uchwycenie nowatorskich osiągnięć naukowych, a także wybitnych technik zastosowanych podczas badania.
Czas publikacji: 08 stycznia 2022 r

