성적증명서
자연
연락
만성 림프구성 백혈병에서 SF3B1 돌연변이의 전체 길이 전사체 특성 분석은 보유 인트론의 하향 조절을 나타냅니다.
전체 길이 성적표|나노포어 시퀀싱|대체 이소형 분석
배경
S스플라이싱 인자 SF3B1의 omatic 돌연변이는 만성 림프구성 백혈병(CLL), 포도막 흑색종, 유방암 등을 비롯한 다양한 암과 관련이 있는 것으로 널리 보고되었습니다. 또한, 단기 전사체 연구에서는 SF3B1 돌연변이에 의해 유도된 비정상적인 스플라이싱 패턴이 밝혀졌습니다.그러나 이러한 대체 접합 패턴에 대한 연구는 짧은 읽기 조립 사본의 한계로 인해 오랫동안 이벤트 수준과 이소형 수준에 대한 지식 부족으로 제한되어 왔습니다.여기서는 AS isoform에 대한 조사를 강화하는 전체 길이의 사본을 생성하기 위해 nanopore 시퀀싱 플랫폼이 도입되었습니다.
실험적 설계
실험
그룹화:1. CLL-SF3B1(WT) 2. CLL-SF3B1(K700E 돌연변이);3. 일반 B세포
시퀀싱 전략:MinION 2D 라이브러리 시퀀싱, PromethION 1D 라이브러리 시퀀싱;동일한 샘플의 짧은 읽기 데이터
시퀀싱 플랫폼:ONT 미니언;ONT 프로메션;
생물정보학 분석
결과
ㅏ6개의 CLL 샘플과 3개의 B 세포에서 총 2억 5,700만 개의 판독이 생성되었습니다.이러한 읽기 중 평균 30.5%가 전체 길이의 사본으로 식별되었습니다.
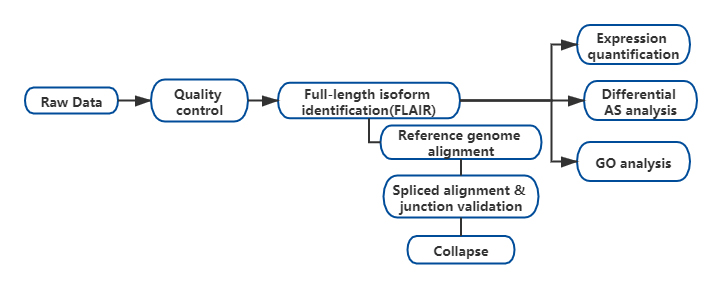
FRNA(FLAIR)의 전체 길이 대체 이소형 분석은 신뢰도가 높은 이소형 세트를 생성하기 위해 개발되었습니다.FLAIR는 다음과 같이 요약될 수 있습니다.
Nanopore 읽기 정렬: 참조 게놈을 기반으로 일반 전사체 구조를 식별합니다.
S플라이스 접합 수정: 주석이 달린 인트론, 짧은 판독 데이터의 인트론 또는 둘 다의 스플라이스 사이트에서 시퀀스 오류(빨간색)를 수정합니다.
Collapse: 스플라이스 접합 체인(첫 번째 통과 세트)을 기반으로 대표적인 isoform을 요약합니다.지원되는 읽기 수(임계값: 3)를 기준으로 높은 신뢰도의 isofrom을 선택합니다.
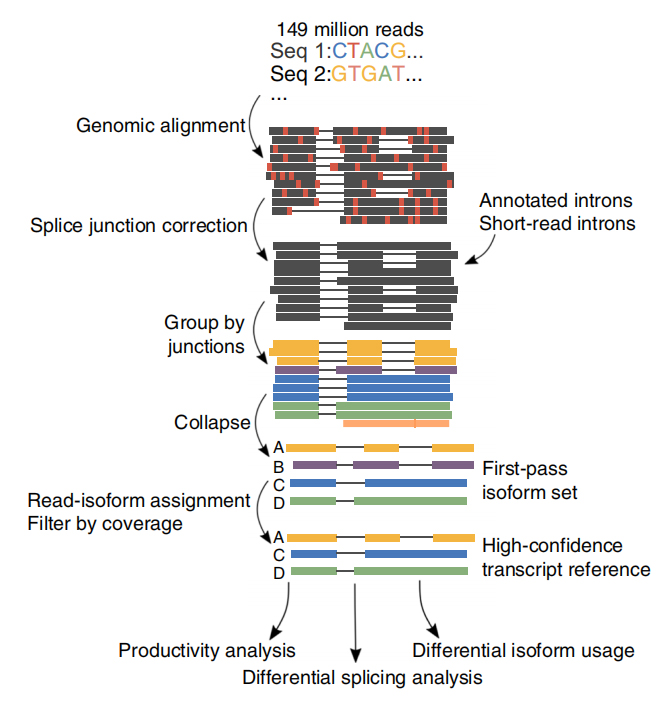
그림 1. CLL의 SF3B1 돌연변이와 관련된 전체 길이의 이소형을 식별하기 위한 FLAIR 분석
FLAIR는 326,699개의 신뢰도가 높은 접합된 이소형을 식별했으며, 그 중 90%는 새로운 이소형입니다.이러한 주석이 없는 이소형의 대부분은 알려진 스플라이스 접합(142,971)의 새로운 조합인 것으로 밝혀졌고, 나머지 소설 이소형은 유지된 인트론(21,700) 또는 신규 엑손(3594)을 포함했습니다.
Long-read 서열은 isoform 수준에서 돌연변이 SF3B1-K700E 변경된 스플라이스 사이트를 식별하는 데 도움을 줍니다.35개의 대체 3'SS와 10개의 대체 5'SS가 SF3B1-K700E와 SF3B1-WT 사이에 상당히 차등적으로 접합되는 것으로 밝혀졌습니다.35개의 변경 중 33개가 긴 판독 시퀀스에 의해 새로 발견되었습니다.Nanopore 데이터에서 SF3B1-K700E 변경 3'SS와 표준 사이트 피크 사이의 거리 분포는 약 -20bp이며 이는 CLL 짧은 읽기 시퀀스에서 보고된 것과 유사하게 제어 분포와 크게 다릅니다.ERGIC3 유전자의 이소형을 분석했는데, 근위 스플라이스 부위를 포함하는 새로운 이소형이 SF3B1-K700E에서 더 많이 발견되었습니다.근위 및 원위 3'SS는 모두 여러 isoform을 생성하는 고유한 AS 패턴과 연관되어 있습니다.
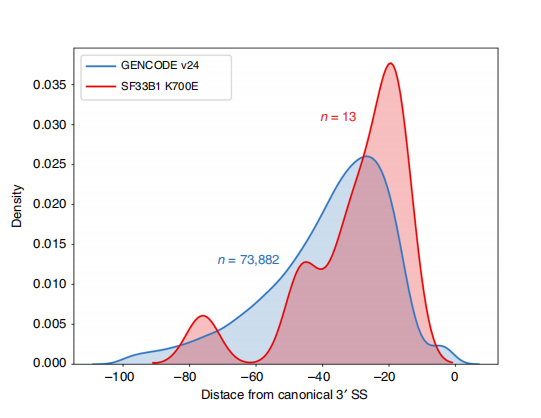
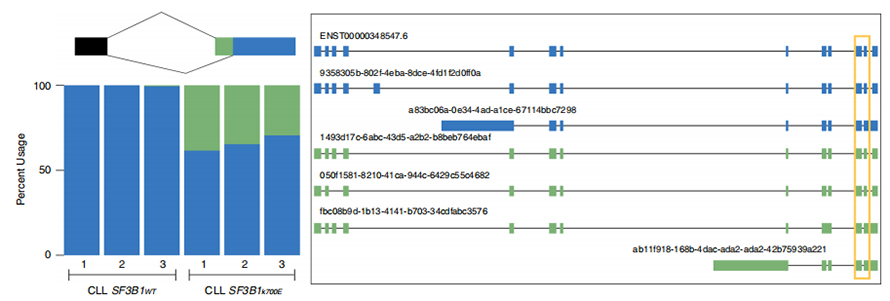
그림 2. 나노기공 시퀀싱 데이터로 확인된 대체 3' 스플라이싱 패턴
IR 이벤트 사용 분석은 IR 식별 및 정량화에 대한 신뢰로 인해 짧은 읽기 기반 분석에서 오랫동안 제한되어 왔습니다.SF3B1-K700E 및 SF3B1-WT에서 IR 이소형의 발현은 나노기공 서열을 기반으로 정량화되었으며, 이는 SF3B1-K700E에서 IR 이소형의 전반적인 하향 조절을 나타냅니다.
그림 4. 세 가지 농업 시스템(A 및 B)에 걸친 농업 강도 및 네트워크 연결성랜덤포레스트 분석(C) 및 농업집약도와 AMF 집락화의 관계(D)
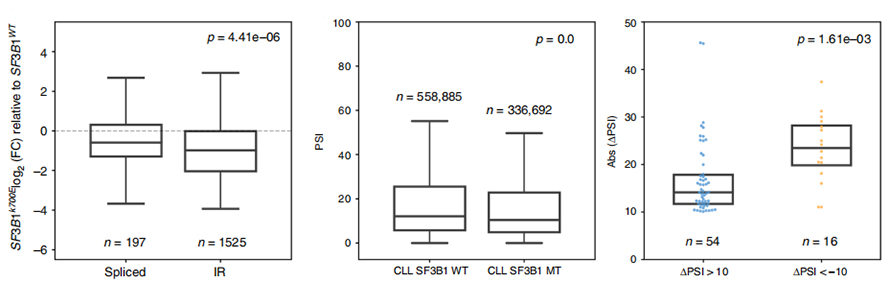
그림 3. 인트론 보유 이벤트는 CLL SF3B1-K700E에서 더욱 강력하게 하향 조절됩니다.
기술
Nanopore 긴 읽기 시퀀싱
N아노포어 시퀀싱(anopore sequencing)은 단일 분자 실시간 전기 신호 시퀀싱 기술입니다.
D이중 가닥 DNA 또는 RNA는 생물막에 내장된 나노다공성 단백질에 결합하고 운동 단백질의 주도 하에 풀립니다.
DNA/RNA 가닥은 전압차의 작용에 따라 특정 속도로 나노기공 채널 단백질을 통과합니다.
M분자는 화학 구조에 따라 서로 다른 전기 신호를 생성합니다.
R시퀀스의 실시간 감지는 베이스 콜링을 통해 이루어집니다.
전장 전사체 시퀀싱 성능
√ 데이터 포화
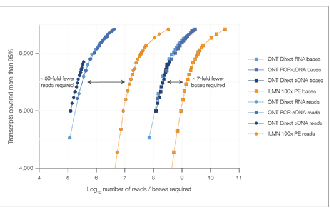
유사한 데이터 포화도에 도달하는 데 필요한 읽기 횟수가 7배 더 적습니다.
√ 성적 증명서 구조 식별
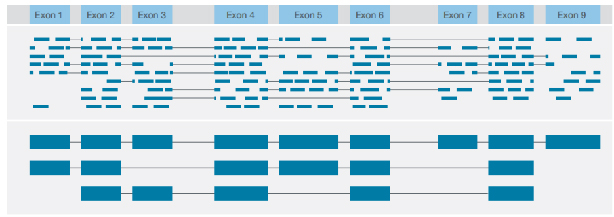
각 성적표의 합의된 전체 길이 판독을 통해 다양한 구조 변형 식별
√ 성적표 수준 차등 분석 - 단문 읽기로 숨겨진 변경 사항 공개
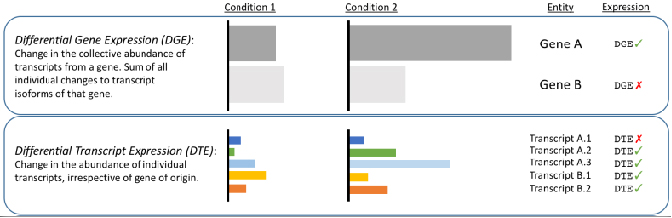
참조
Tang AD, Soulette CM, Baren MJV 등만성 림프구성 백혈병에서 SF3B1 돌연변이의 전체 길이 전사체 특성 분석은 보유된 인트론의 하향 조절을 보여줍니다[J].네이처커뮤니케이션즈.
기술 및 하이라이트 다양한 연구 분야에서 다양한 고처리량 시퀀싱 기술의 최신 성공적인 적용과 실험 설계 및 데이터 마이닝의 뛰어난 아이디어를 공유하는 것을 목표로 합니다.
게시 시간: 2022년 1월 8일

