NGS-mRNA (riferimentu)
U transcriptome hè u ligame trà l'infurmazione genetica genomica è u proteoma di a funzione biologica.A regulazione di u livellu trascrizionale hè u modu di regulazione più impurtante è più studiatu di l'organisimi.A sequenza di u transcriptome pò sequenzià u transcriptome in ogni puntu in u tempu o in ogni cundizione, cù una risoluzione precisa à un unicu nucleotide. Pò riflette dinamicamente u livellu di trascrizzione genica, identificà è quantificà simultaneamente trascrizioni rari è normali, è furnisce l'infurmazioni strutturali di trascrizioni specifiche di mostra.
Attualmente, a tecnulugia di sequencing transcriptome hè stata largamente usata in l'agronomia, a medicina è altri campi di ricerca, cumprese a regulazione di u sviluppu di l'animali è di e piante, l'adattazione ambientale, l'interazione immune, a localizazione di geni, l'evoluzione genetica di e spezie è a rilevazione di tumori è di malatie genetiche.
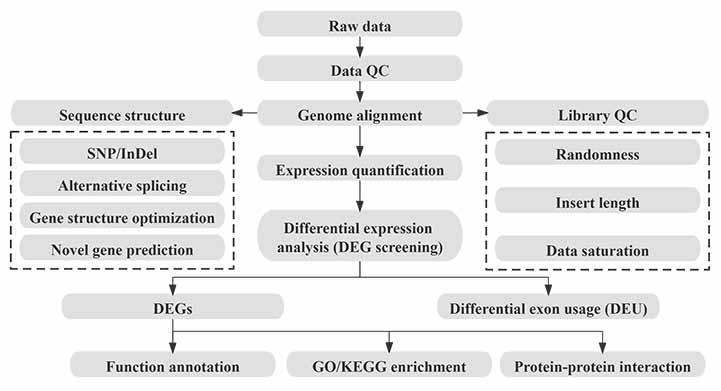
Bioinformatica
Flussu di travagliu di bioinformatica