BMKMANU S1000 Spatial Transcriptome
BMKMANU S1000 Spatial Transcriptome Schema Tecnicu
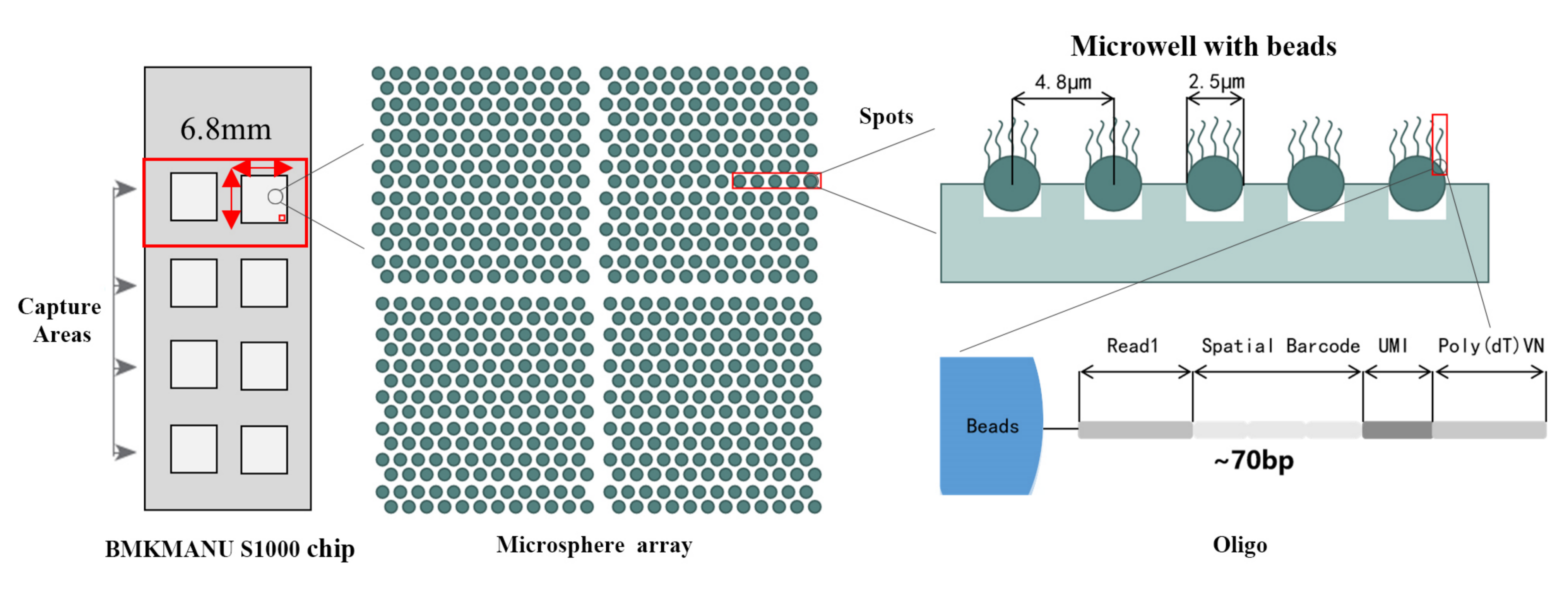
Vantaghji di BMKMANU S1000
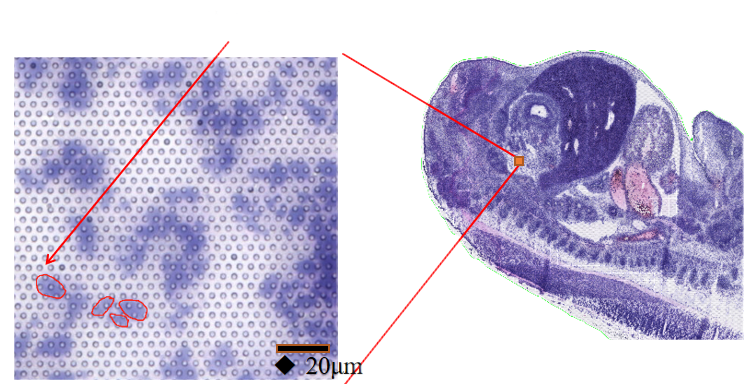
1) Risoluzione sub-cellulare: Ogni area di cattura cuntene > 2 milioni di punti codificati spaziali cù un diametru di 2,5 µm è una spaziatura di 5 µm trà i centri spot, chì permette l'analisi di trascriptoma spaziale cù risoluzione sub-cellulare (5 µm).
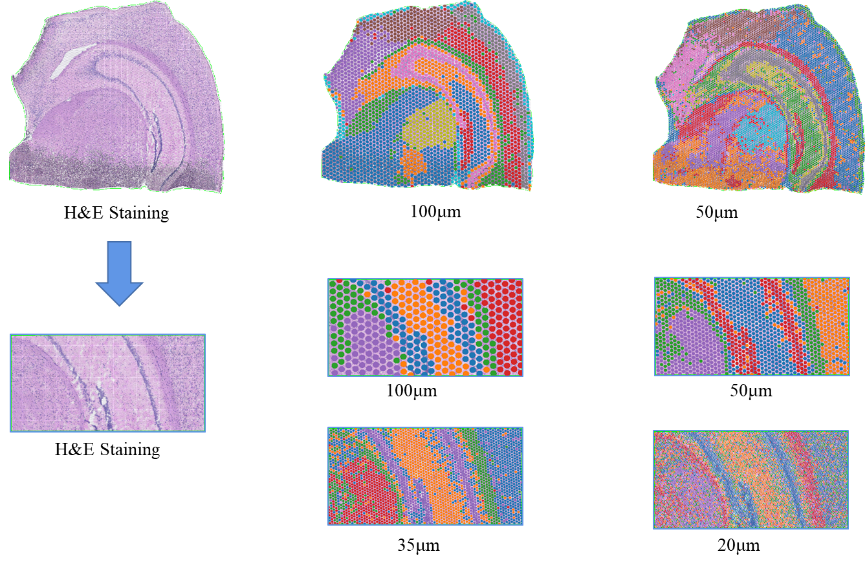
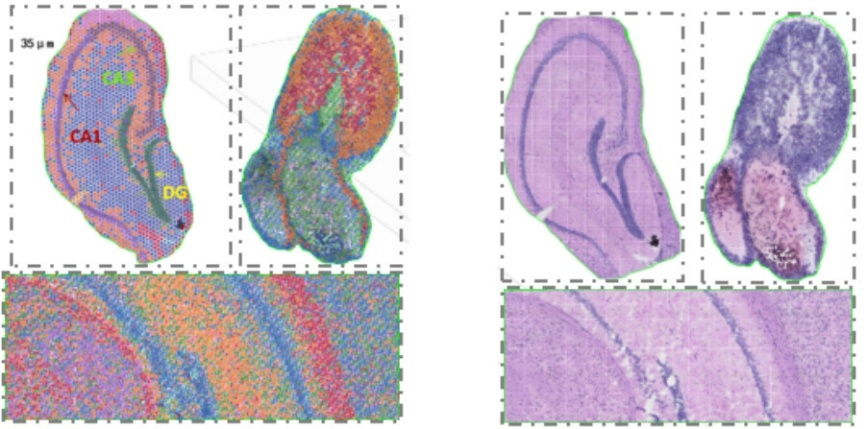
2) Analisi di risoluzione multi-livellu: analisi multi-livellu flessibile chì varieghja da 100 μm à 5 μm per risolve diverse caratteristiche di i tessuti à una risoluzione ottima.
3) Prufessu di transcriptome cumpletu: e trascrizioni catturate da tutta a diapositiva di tissuti ponu esse analizate, senza restrizioni à u numeru di geni di destinazione è di l'area di destinazione.
Specificazioni di serviziu
| Biblioteca | Strategia di sequenza | Data Output cunsigliatu |
| Biblioteca di cDNA S1000 | BMKMANU S1000-Illumina PE150 | 60 Gb/mostra |
Requisiti di mostra
| Campione | numeru | Taglia | Qualità RNA |
| Bloccu di tissutu incrustatu OCT | 2-3 blocchi / campione | Circa.6,8x6,8x6,8 mm3 | RIN≥7 |
Per più dettagli nantu à a guida di preparazione di mostra è u flussu di travagliu di serviziu, sentite liberu di parlà cun aEspertu BMKGENE
Flussu di travagliu di serviziu

Prughjettu di cuncepimentu

Consegna di mostra

Pre-esperimentu
Codici a barre spaziali

Custruzzione di biblioteca

Sequencing

Analisi di dati

Servizi post-vendita
I dati generati da BMKMANU S1000 sò analizati cù u software "BSTMatrix", chì hè cuncepitu indipendentemente da BMKGENE, cumprese:
1) Generazione di matrice di espressione genica
2) Trattamentu di l'imaghjini HE
3) Compatibile cù software di terze parti downstream per l'analisi
4) "BSTViewer" in linea aiuta à ottene risultati di visualizazione in diverse risoluzioni.
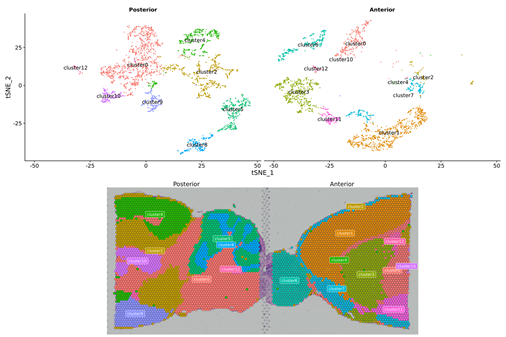
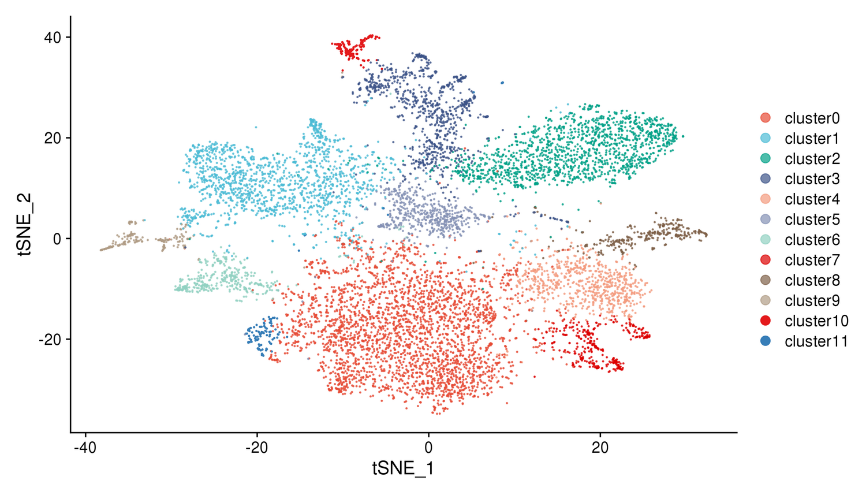
1.Spot clustering
2.Distribuzione spaziale
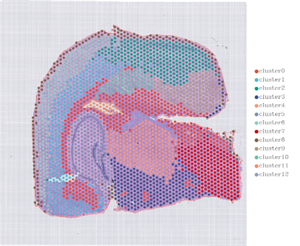
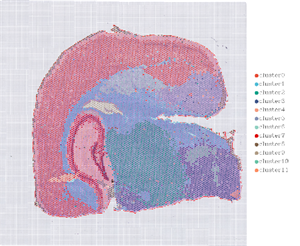
Note: Risoluzionelivellu = 13 (100 µm, manca); 7 (50 µm, diritta)
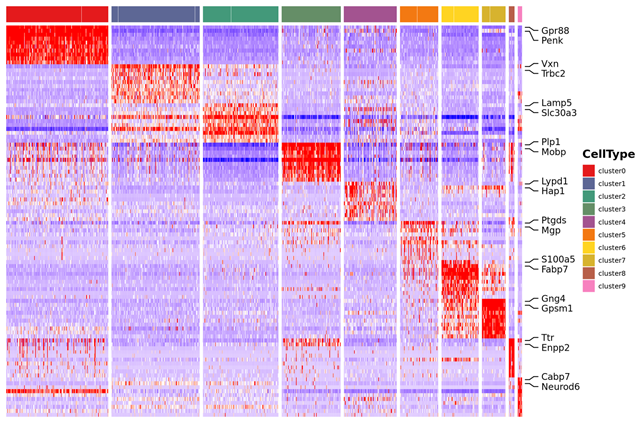
3.Marker expression abbundanza clustering heatmap
4.Analisi di dati Inter-sample