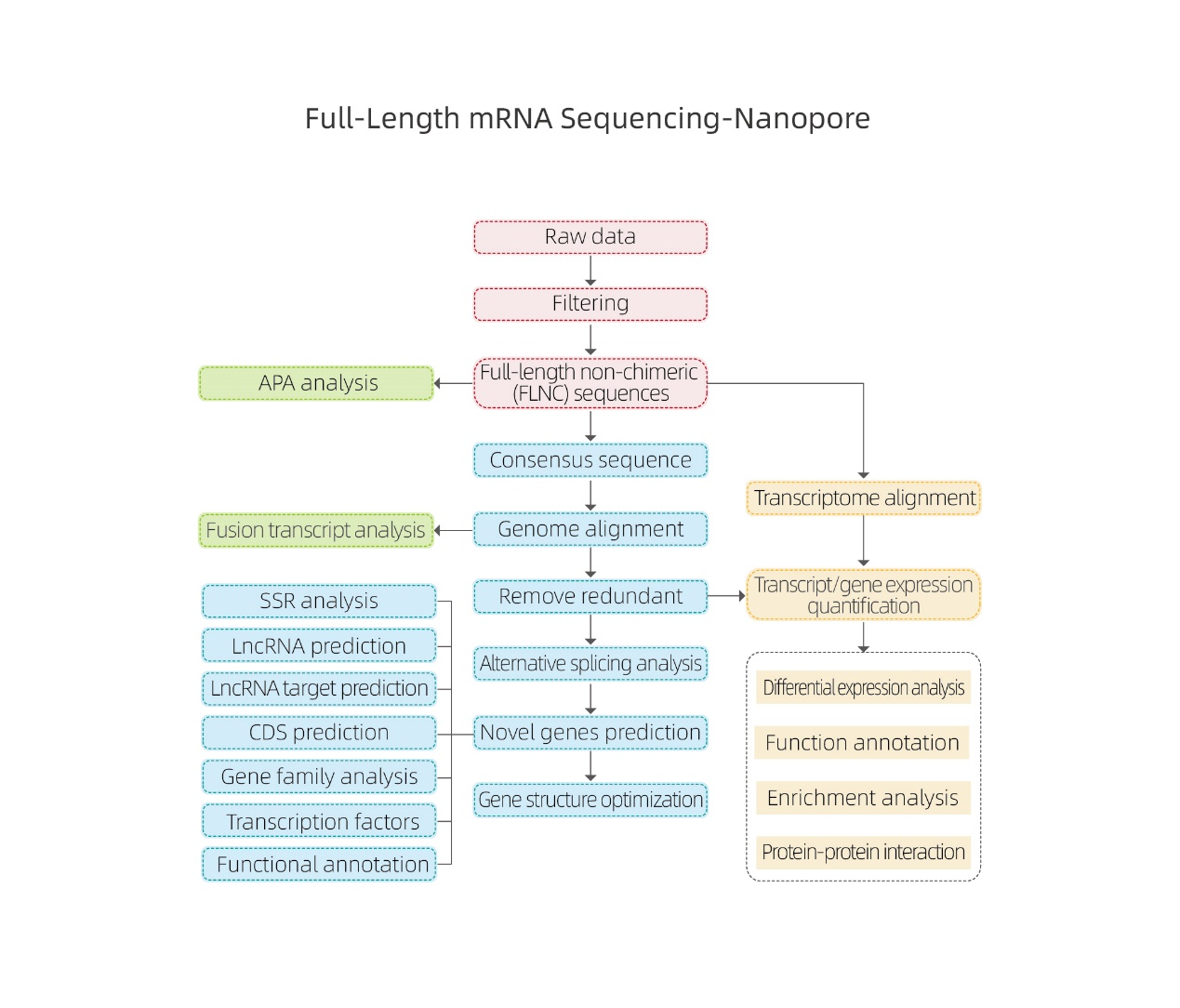
Sequenza di mRNA di lunghezza completa-Nanopore
Vantaghji di serviziu
● Bias di sequenza bassa
● Revealing molécules cDNA full-length
● Meno dati necessariu per copre u listessu numeru di trascrizioni
● Identificazione di parechje isoforme per genu
● Quantificazione di l'espressione in u livellu isoforme
Specificazioni di serviziu
| Biblioteca | Piattaforma | Rendimentu di dati cunsigliatu (Gb) | Controlu di qualità |
| cDNA-PCR (arricchito in Poly-A) | Nanopore PromethION P48 | 6 Gb/campionu (Secondu a spezia) | Rapportu di lunghezza completa> 70% Puntu di qualità mediu: Q10
|
Analisi bioinformatica
●Trattamentu di dati crudi
● Identificazione di a trascrizione
● Splicing alternativu
● Quantificazione di l'espressione in u nivellu di u genu è u livellu di l'isoforma
● Analisi di spressione differenziale
● Annotazione di funzioni è arricchimentu (DEG è DET)
Requisiti di mostra è consegna
Requisiti di mostra:
Nucleotidi:
| Conc. (ng/μl) | Quantità (μg) | Purità | Integrità |
| ≥ 100 | ≥ 0,6 | OD260/280 = 1,7-2,5 OD260/230 = 0,5-2,5 Cuntaminazione limitata o micca di proteine o DNA mostrata nantu à u gel. | Per i pianti: RIN≥7.0; Per l'animali: RIN≥7,5; 5.0≥28S/18S≥1.0; limitazione o senza elevazione di basa |
Tissu: Pesu (secu): ≥1 g
* Per i tissuti più chjuchi di 5 mg, ricumandemu di mandà un campione di tissutu congelatu (in nitrogenu liquidu).
Suspension cellula: Conte di cellula = 3 × 106- 1×107
* Hè cunsigliatu di spedite lisatu di cellule congelate.In casu chì a cellula conta più chjuca di 5 × 105, Flash frozen in nitrogenu liquidu hè cunsigliatu, chì hè preferibile per micro extraction.
Campioni di sangue: Volume≥1 ml
Cunsigliu Cunsigliu di Campione
Contenitore: Tubu da centrifuga da 2 ml (Un fogliu di stagno ùn hè micca cunsigliatu)
Esempiu di etichettatura: Gruppu + replicate per esempiu A1, A2, A3;B1, B2, B3....
Spedizione: 2 、 Ghiaccio seccu: I campioni anu da esse imballati in sacchetti è intarrati in ghiaccio seccu.
- Tubi RNAstable: i campioni di RNA ponu esse secchi in un tubu di stabilizazione di RNA (per esempiu RNAstable®) è spediti à a temperatura di l'ambienti.
Flussu di travagliu di serviziu
Nucleotidi:

Consegna di mostra

Custruzzione di biblioteca

Sequencing

Analisi di dati

Servizi post-vendita
Flussu di travagliu di serviziu
Tissu:

Prughjettu di cuncepimentu

Consegna di mostra

estrazione di RNA

Custruzzione di biblioteca

Sequencing

Analisi di dati

Servizi post-vendita
1.Analisi di l'espressione differenziale - Trama di u vulcanu
L'analisi di l'espressione differenziale pò esse processata in u livellu di u genu per identificà i geni espressi di manera differenziale (DEG) è in u livellu isoforme per identificà in modu differenziale.
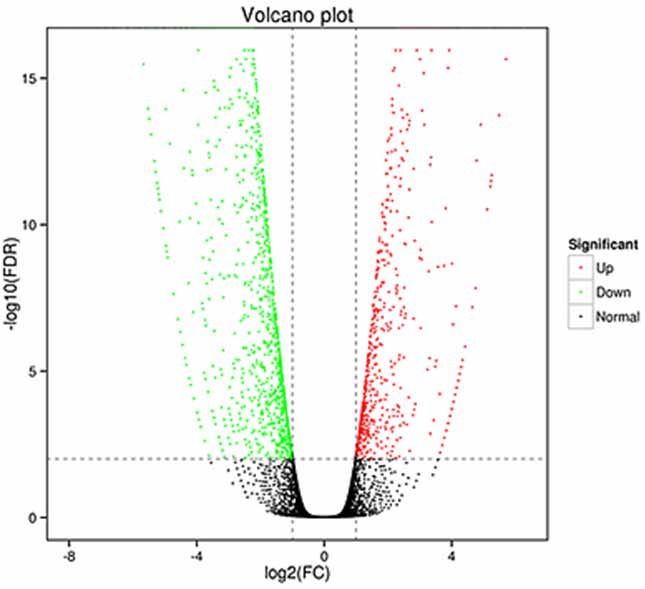
trascrizioni espresse (DET)
2.Mappa di calore di clustering gerarchicu
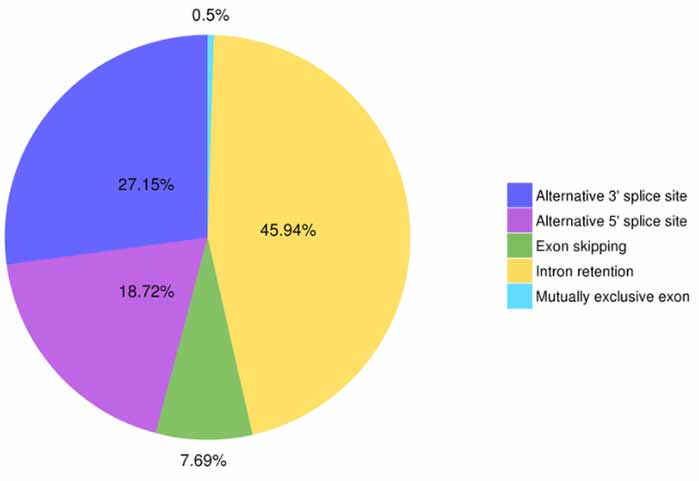
3.Alternative splicing identificazione è classificazione
Cinque tippi di avvenimenti di splicing alternativu ponu esse previsti da Astalavista.
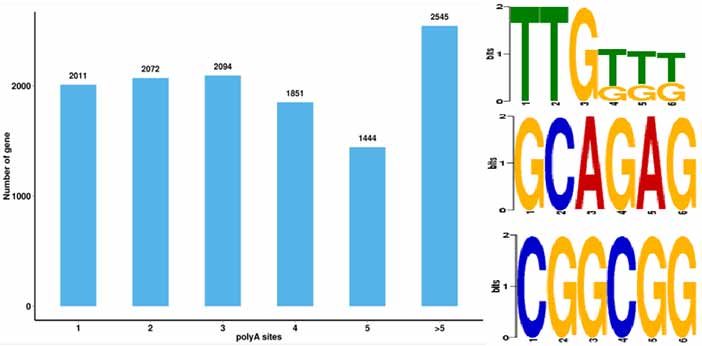
4.Identificazione di eventi di poliadenilazione alternativa (APA) è Motif à 50 bp a monte di poli-A
Casu BMK
Identificazione di splicing alternativu è quantificazione à livellu di isoforme per sequenza di transcriptome full-length di nanopori
Publicatu:Nature Communications, 2020
Strategia di sequenza:
Grouping: 1. CLL-SF3B1 (WT);2. CLL-SF3B1 (mutazione K700E);3. Célule B normale
Strategia di sequenza: sequenza di biblioteca MinION 2D, sequenza di biblioteca PromethION 1D;brevi lettura di dati da i stessi campioni
Piattaforma di sequenza: Nanopore MinION;Nanopore PromethION;
I risultati chjave
1.Isoform-livellu Alternative Splicing Identification
Sequenze di lettura longa permette l'identificazione di SF3B1 mutanteK700E-siti di splice alterati à u livellu di l'isoforma.35 alternative 3′SS è 10 alternative 5′SS sò state trovate esse spliced significativamente differenziali trà SF3B1K700Eè SF3B1WT.33 di e 35 alterazioni sò stati scuperti di novu da sequenze di lettura longa.
2.Isoform-livellu Alternative Splicing quantification
Espressione di isoforme di ritenzione di introni (IR) in SF3B1K700Eè SF3B1WTsò stati quantificati in basa di sequenze di nanopori, chì palesanu una regulazione globale di l'isoforme IR in SF3B1.K700E.
Riferimentu
Tang AD , Soulette CM , Baren MJV , et al.A caratterizzazione di a trascrizione integrale di a mutazione SF3B1 in a leucemia linfocitaria cronica revela una regulazione di l'intruni ritenuti [J].Comunicazioni di a natura.