-
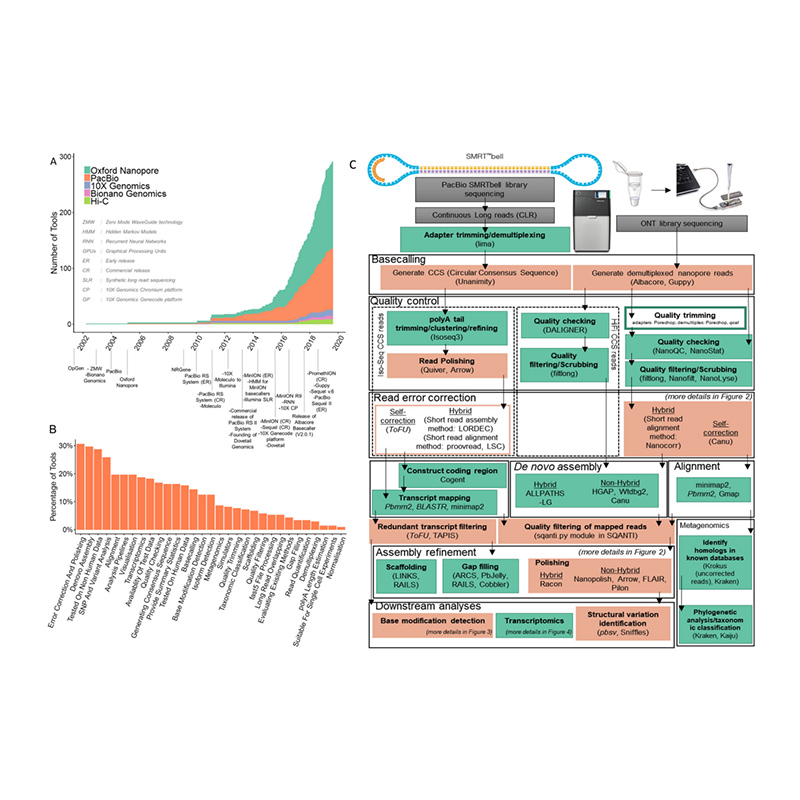
Séquençage du génome végétal/animal de novo
De novole séquençage fait référence à la construction du génome entier d'une espèce à l'aide de technologies de séquençage, par exemple PacBio, Nanopore, NGS, etc., en l'absence d'un génome de référence.L'amélioration remarquable de la longueur de lecture des technologies de séquençage de troisième génération a apporté de nouvelles opportunités dans l'assemblage de génomes complexes, tels que ceux présentant une hétérozygotie élevée, un taux élevé de régions répétitives, des polyploïdes, etc. Avec une longueur de lecture de plusieurs dizaines de kilobases, ces lectures de séquençage permettent résolution des éléments répétitifs, des régions avec des contenus anormaux en GC et d'autres régions très complexes.
Plateforme : PacBio Sequel II /Nanopore PromethION P48/Illumina NovaSeq6000
-

Assemblage du génome basé sur Hi-C
Hi-C est une méthode conçue pour capturer la configuration des chromosomes en combinant le sondage des interactions basées sur la proximité et le séquençage à haut débit.On pense que l'intensité de ces interactions est négativement corrélée à la distance physique sur les chromosomes.Par conséquent, les données Hi-C pourraient guider le regroupement, l'ordre et l'orientation des séquences assemblées dans un projet de génome et les ancrer sur un certain nombre de chromosomes.Cette technologie permet un assemblage du génome au niveau des chromosomes en l'absence de carte génétique basée sur la population.Chaque génome a besoin d'un Hi-C.
Plate-forme:Illumina NovaSeq6000 / DNBSEQ
-

Génétique évolutive
La génétique évolutive est un service de séquençage complet conçu pour fournir une interprétation complète des informations évolutives de matériaux donnés en fonction des variations génétiques, y compris les SNP, InDels, SV et CNV.Il fournit toutes les analyses fondamentales nécessaires pour décrire les changements évolutifs et les caractéristiques génétiques des populations, telles que la structure de la population, la diversité génétique, les relations phylogéniques, etc. Il contient également des études sur le flux génétique, ce qui permet d'estimer la taille effective de la population, le temps de divergence.
-
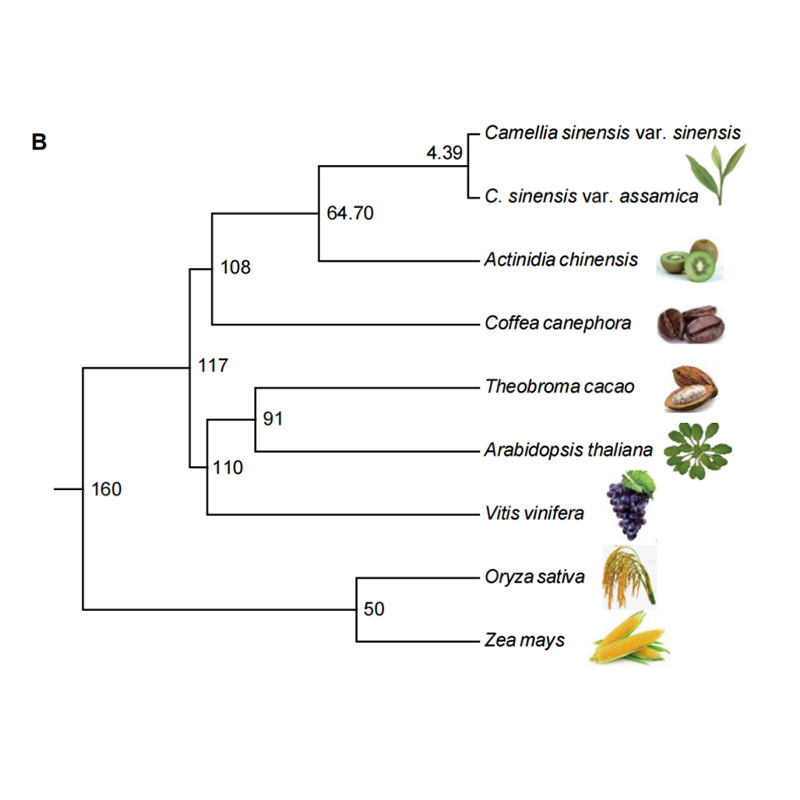
Génomique comparative
La génomique comparative signifie littéralement comparer les séquences et les structures complètes du génome de différentes espèces.Cette discipline vise à révéler l'évolution des espèces, la fonction des gènes, le mécanisme de régulation des gènes au niveau du génome en identifiant les structures de séquence et les éléments qui se sont conservés ou différenciés entre différentes espèces.Une étude de génomique comparative typique comprend des analyses de la famille de gènes, du développement évolutif, de la duplication du génome entier, de la pression sélective, etc.
-

Analyse de Segregant groupée
L'analyse des ségrégants en vrac (BSA) est une technique utilisée pour identifier rapidement les marqueurs génétiques associés au phénotype.Le flux de travail principal de BSA consiste à sélectionner deux groupes d'individus avec des phénotypes extrêmement opposés, à regrouper l'ADN de tous les individus pour former deux volumes d'ADN, à identifier des séquences différentielles entre deux pools.Cette technique a été largement utilisée pour identifier des marqueurs génétiques fortement associés à des gènes ciblés dans des génomes végétaux/animaux.






