-
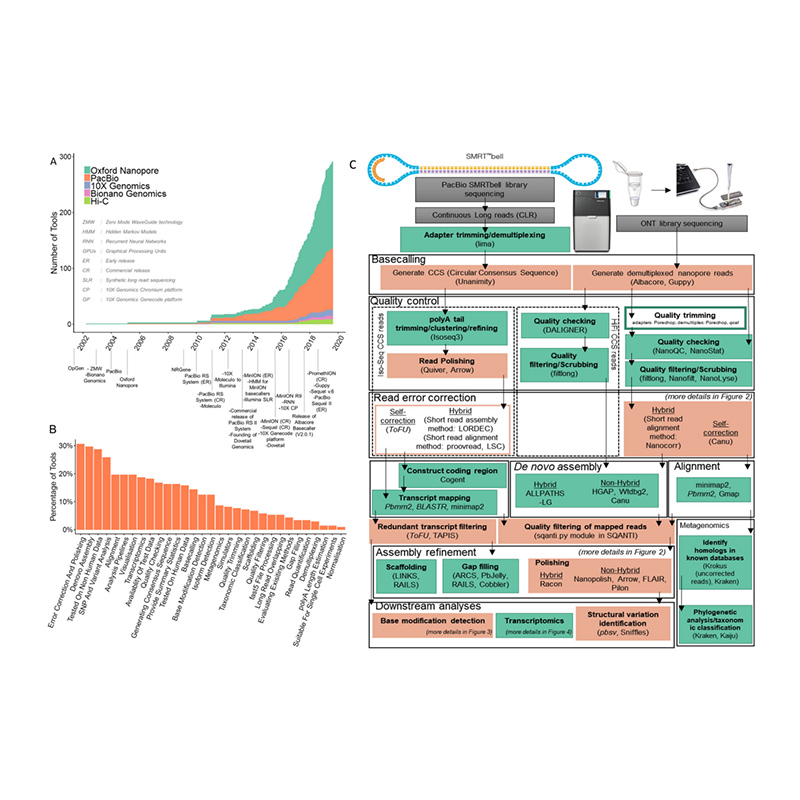
Pflanzen-/Tier-De-novo-Genomsequenzierung
NeuSequenzierung bezieht sich auf die Konstruktion des gesamten Genoms einer Spezies unter Verwendung von Sequenzierungstechnologien, z. B. PacBio, Nanopore, NGS usw., in Abwesenheit eines Referenzgenoms.Die bemerkenswerte Verbesserung der Leselänge von Sequenzierungstechnologien der dritten Generation hat neue Möglichkeiten beim Zusammenbau komplexer Genome eröffnet, z. B. solche mit hoher Heterozygotie, einem hohen Anteil an repetitiven Regionen, Polyploiden usw. Mit einer Leselänge von mehreren zehn Kilobasen ermöglichen diese Sequenzierungslesevorgänge Auflösung von repetitiven Elementen, Regionen mit anormalem GC-Gehalt und anderen hochkomplexen Regionen.
Plattform: PacBio Sequel II / Nanopore PromethION P48 / Illumina NovaSeq6000
-

Hi-C-basierte Genomassemblierung
Hi-C ist eine Methode zur Erfassung der Chromosomenkonfiguration durch die Kombination von proximitätsbasierten Interaktionen und Hochdurchsatz-Sequenzierung.Es wird angenommen, dass die Intensität dieser Wechselwirkungen negativ mit der physischen Entfernung auf Chromosomen korreliert.Daher könnten Hi-C-Daten das Clustern, Ordnen und Ausrichten zusammengesetzter Sequenzen in einem Entwurfsgenom und das Verankern dieser auf einer bestimmten Anzahl von Chromosomen leiten.Diese Technologie ermöglicht einen Genomaufbau auf Chromosomenebene ohne eine populationsbasierte genetische Karte.Jedes einzelne Genom braucht ein Hi-C.
Plattform: Illumina NovaSeq6000 / DNBSEQ
-

Evolutionäre Genetik
Evolutionsgenetik ist ein umfassender Sequenzierungsdienst, der entwickelt wurde, um eine umfassende Interpretation der evolutionären Informationen bestimmter Materialien basierend auf genetischen Variationen, einschließlich SNPs, InDels, SVs und CNVs, bereitzustellen.Es enthält alle grundlegenden Analysen, die zur Beschreibung evolutionärer Veränderungen und genetischer Merkmale von Populationen erforderlich sind, wie z. B. Populationsstruktur, genetische Vielfalt, phylogenetische Beziehungen usw. Es enthält auch Studien zum Genfluss, die die Schätzung der effektiven Populationsgröße und der Divergenzzeit ermöglichen.
-
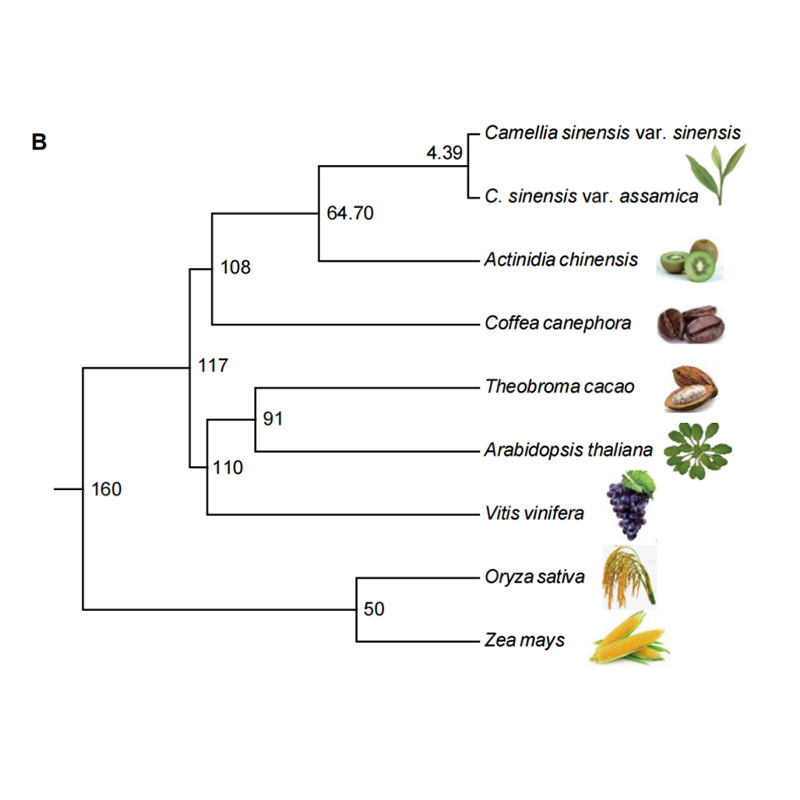
Vergleichende Genomik
Vergleichende Genomik bedeutet wörtlich den Vergleich der vollständigen Genomsequenzen und -strukturen verschiedener Arten.Diese Disziplin zielt darauf ab, die Artenevolution, Genfunktion und Genregulationsmechanismen auf Genomebene aufzudecken, indem die Sequenzstrukturen und Elemente identifiziert werden, die über verschiedene Arten hinweg konserviert oder differenziert wurden.Eine typische vergleichende Genomikstudie umfasst Analysen der Genfamilie, der evolutionären Entwicklung, der Duplikation des gesamten Genoms, des selektiven Drucks usw.
-

Bulked-Segregant-Analyse
Die Bulked-Segregant-Analyse (BSA) ist eine Technik, die eingesetzt wird, um Phänotyp-assoziierte genetische Marker schnell zu identifizieren.Der Hauptarbeitsablauf von BSA umfasst die Auswahl von zwei Gruppen von Individuen mit extrem gegensätzlichen Phänotypen, das Poolen der DNA aller Individuen, um zwei DNA-Massen zu bilden, und das Identifizieren unterschiedlicher Sequenzen zwischen zwei Pools.Diese Technik wurde ausgiebig zur Identifizierung von genetischen Markern eingesetzt, die stark mit Zielgenen in Pflanzen-/Tiergenomen assoziiert sind.






