
Séquençage métagénomique -NGS
Avantages des services
● Sans isolement ni culture pour le profilage de la communauté microbienne
● Haute résolution pour détecter les espèces à faible abondance dans les échantillons environnementaux
● L'idée de « méta- » intègre toutes les caractéristiques biologiques au niveau fonctionnel, au niveau de l'espèce et au niveau des gènes, ce qui reflète une vision dynamique plus proche de la réalité.
● BMK accumule une expérience considérable dans divers types d'échantillons avec plus de 10 000 échantillons traités.
Spécifications des services
| Plate-forme | Séquençage | Données recommandées | Délai d'exécution |
| Plateforme Illumina NovaSeq | PE150 | 6G/10G/20G | 45 jours ouvrables |
Analyses bioinformatiques
● Contrôle qualité des données brutes
● Assemblage du métagénome
● Ensemble de gènes non redondants et annotation
● Analyse de la diversité des espèces
● Analyse de la diversité des fonctions génétiques
● Analyse inter-groupes
● Analyse d'association avec des facteurs expérimentaux
Exigences et livraison des échantillons
Exigences de l'échantillon :
PourExtraits d'ADN:
| Échantillon type | Montant | Concentration | Pureté |
| Extraits d'ADN | > 30ng | > 1 ng/μl | DO260/280 = 1,6-2,5 |
Pour les échantillons environnementaux :
| Échantillon type | Procédure d'échantillonnage recommandée |
| Sol | Quantité d'échantillonnage : env.5g;La substance flétrie restante doit être retirée de la surface ;Broyer les gros morceaux et passer au filtre de 2 mm ;Échantillons aliquotes dans un tube EP ou un cyrotube stérile pour réservation. |
| Excréments | Quantité d'échantillonnage : env.5g;Recueillir et aliquoter les échantillons dans un tube EP stérile ou un cryotube pour réservation. |
| Contenu intestinal | Les échantillons doivent être traités dans des conditions aseptiques.Laver les tissus collectés avec du PBS ;Centrifuger le PBS et recueillir le précipitant dans des tubes EP. |
| Boue | Quantité d'échantillonnage : env.5g;Recueillir et aliquoter l'échantillon de boue dans un tube EP stérile ou un cryotube pour réservation |
| Plan d'eau | Pour les échantillons contenant une quantité limitée de microbes, tels que l'eau du robinet, l'eau de puits, etc., collecter au moins 1 L d'eau et passer à travers un filtre de 0,22 μm pour enrichir les microbes sur la membrane.Conservez la membrane dans un tube stérile. |
| Peau | Grattez soigneusement la surface de la peau avec un coton-tige stérile ou une lame chirurgicale et placez-la dans un tube stérile. |
Livraison d’échantillon recommandée
Congelez les échantillons dans de l'azote liquide pendant 3 à 4 heures et conservez-les dans de l'azote liquide ou à -80 degrés pour une réservation à long terme.L’envoi d’un échantillon avec de la glace carbonique est requis.
Flux de travail des services

Livraison d'échantillon

Construction d'une bibliothèque

Séquençage

L'analyse des données

Services après-vente
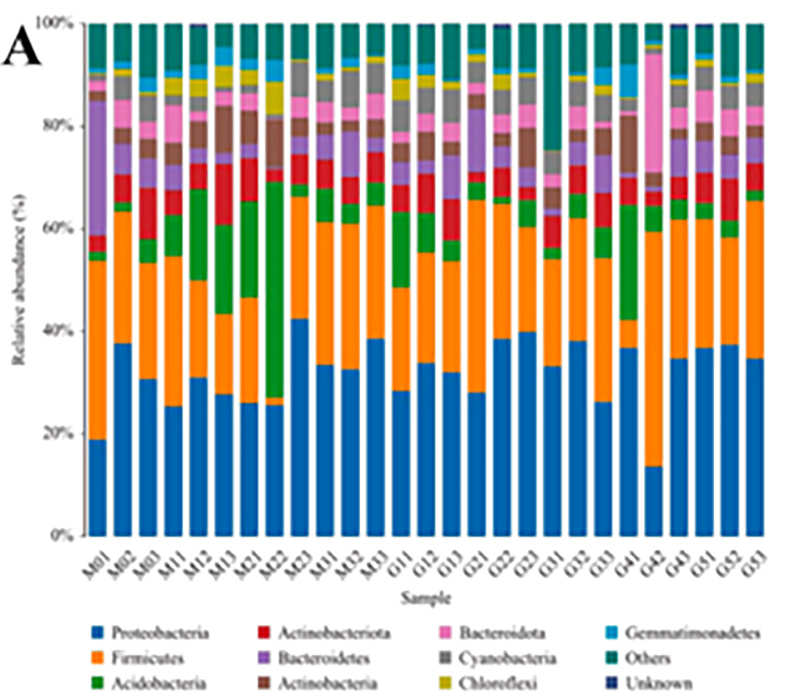
1.Histogramme : répartition des espèces
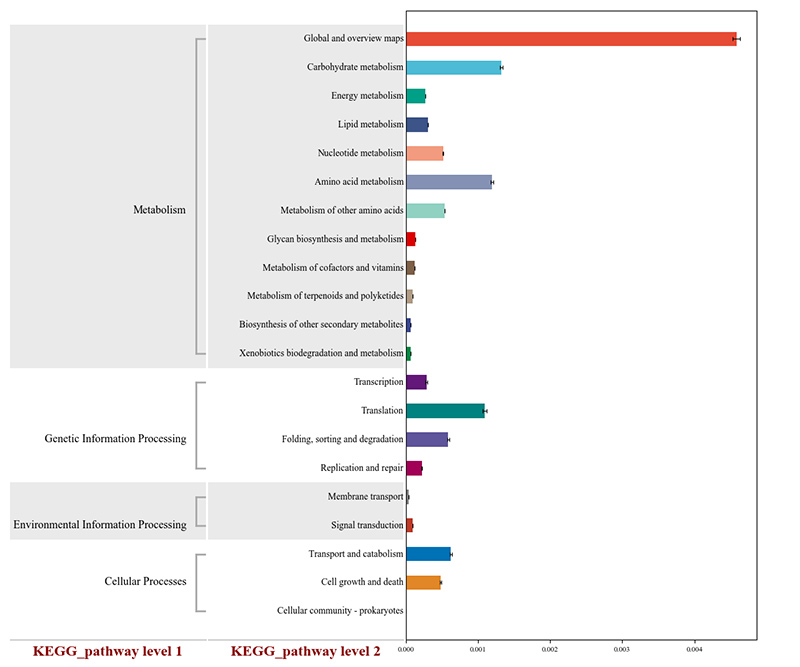
2. Gènes fonctionnels annotés aux voies métaboliques KEGG
3.Carte thermique : fonctions différentielles basées sur l'abondance relative des gènes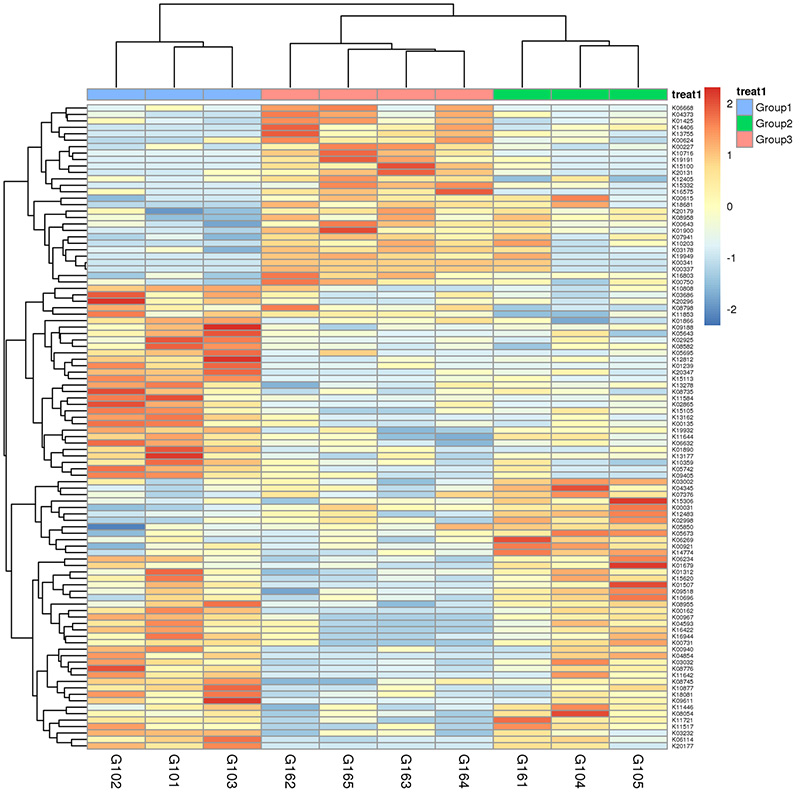 4.Circos des gènes de résistance aux antibiotiques CARD
4.Circos des gènes de résistance aux antibiotiques CARD
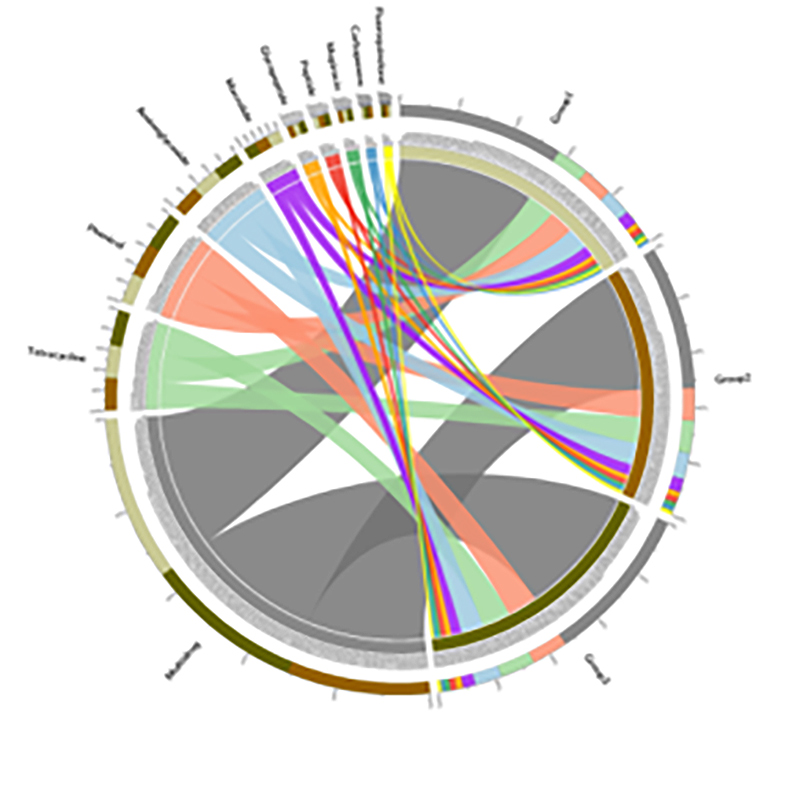
Affaire BMK
Prévalence des gènes de résistance aux antibiotiques et des bactéries pathogènes le long du continuum sol-racine des mangroves
Publié :Journal des matières dangereuses, 2021
Stratégie de séquençage :
Matériel : Extraits d'ADN de quatre fragments d'échantillons associés à des racines de mangrove : compartiments de sol non planté, de rhizosphère, d'épisphère et d'endosphère
Plateforme : Illumina HiSeq 2500
Cibles : métagénome
Région V3-V4 du gène ARNr 16S
Résultats clés
Le séquençage métagénomique et le profilage des métabarcodes sur le continuum sol-racine des jeunes arbres de mangrove ont été traités afin d'étudier la dissémination des gènes de résistance aux antibiotiques (ARG) du sol vers les plantes.Les données métagénomiques ont révélé que 91,4 % des gènes de résistance aux antibiotiques étaient communément identifiés dans les quatre compartiments du sol mentionnés ci-dessus, ce qui montre une tendance continue.Le séquençage des amplicons d’ARNr 16S a généré 29 285 séquences, représentant 346 espèces.En combinaison avec le profilage des espèces par séquençage d'amplicons, cette dissémination s'est avérée indépendante du microbiote associé aux racines, mais elle pourrait être facilitée par la mobilité des éléments génétiques.Cette étude a identifié le flux d'ARG et d'agents pathogènes du sol vers les plantes à travers un continuum sol-racine interconnecté.
Référence
Wang, C. , Hu, R. , Strong, PJ , Zhuang, W. et Shu, L. .(2020).Prévalence des gènes de résistance aux antibiotiques et des bactéries pathogènes le long du continuum sol-racine de mangrove.Journal des Matériaux Dangereux, 408, 124985.











