-

Vollständige mRNA-Sequenzierung – Nanopore
Die RNA-Sequenzierung ist ein unschätzbares Werkzeug für eine umfassende Transkriptomanalyse.Zweifellos hat die traditionelle Short-Read-Sequenzierung hier zahlreiche wichtige Entwicklungen erzielt.Dennoch stößt es häufig auf Einschränkungen bei der Identifizierung von Isoformen in voller Länge, Quantifizierung und PCR-Verzerrung.
Die Nanoporensequenzierung unterscheidet sich von anderen Sequenzierungsplattformen dadurch, dass die Nukleotide direkt ohne DNA-Synthese abgelesen werden und eine lange Ablesung von mehreren zehn Kilobasen erzeugt.Dies ermöglicht das direkte Auslesen von Transkripten in voller Länge und die Bewältigung der Herausforderungen bei Studien auf Isoform-Ebene.
Plattform:Nanopore PromethION
Bücherei:cDNA-PCR
-

De novo Transkriptom-Sequenzierung in voller Länge -PacBio
NeuTranskriptomsequenzierung in voller Länge, auch bekannt alsNeuIso-Seq nutzt die Vorteile des PacBio-Sequenzers in Bezug auf die Leselänge, wodurch die Sequenzierung von cDNA-Molekülen in voller Länge ohne Unterbrechungen ermöglicht wird.Dies vermeidet vollständig alle Fehler, die in Transkript-Assemblierungsschritten erzeugt werden, und konstruiert Unigene-Sets mit Auflösung auf Isoform-Ebene.Diese Unigen-Sets liefern leistungsstarke genetische Informationen als „Referenzgenom“ auf Transkriptom-Ebene.Darüber hinaus ermöglicht dieser Service in Kombination mit Sequenzierungsdaten der nächsten Generation eine genaue Quantifizierung der Expression auf Isoform-Ebene.
Plattform: PacBio Sequel IIBibliothek: SMRT-Glockenbibliothek -

Eukaryotische mRNA-Sequenzierung – Illumina
Die mRNA-Sequenzierung ermöglicht die Profilierung aller mRNAs, die unter bestimmten Bedingungen von Zellen transkribiert wurden.Es ist eine leistungsstarke Technologie zur Aufdeckung von Genexpressionsprofilen, Genstrukturen und molekularen Mechanismen bestimmter biologischer Prozesse.Bisher wurde die mRNA-Sequenzierung in der Grundlagenforschung, der klinischen Diagnostik, der Arzneimittelentwicklung usw. weit verbreitet eingesetzt.
Plattform: Illumina NovaSeq 6000
-

Nicht referenzbasierte mRNA-Sequenzierung – Illumina
Die mRNA-Sequenzierung verwendet die Sequenzierungstechnik der nächsten Generation (NGS), um die Boten-RNA (mRNA) aus Eukaryoten zu einem bestimmten Zeitpunkt zu erfassen, zu dem einige spezielle Funktionen aktiviert werden.Das längste gespleißte Transkript wurde „Unigene“ genannt und als Referenzsequenz für die anschließende Analyse verwendet, was ein wirksames Mittel ist, um den molekularen Mechanismus und das regulatorische Netzwerk der Spezies ohne Referenz zu untersuchen.
Nach Zusammenstellung der Transkriptomdaten und unigener funktioneller Annotation
(1) SNP-Analyse, SSR-Analyse, CDS-Vorhersage und Genstruktur werden durchgeführt.
(2) Es wird eine Quantifizierung der unigenen Expression in jeder Probe durchgeführt.
(3) Unterschiedlich exprimierte Unigene zwischen Proben (oder Gruppen) werden basierend auf der Unigenexpression entdeckt
(4)Clustering, funktionelle Annotation und Anreicherungsanalyse von differentiell exprimierten Unigenen werden durchgeführt
-

Lange nicht codierende Sequenzierung – Illumina
Lange nicht codierende RNAs (lncRNAs) sind eine Art von RNA-Molekülen mit einer Länge von mehr als 200 nt, die sich durch ein extrem niedriges Codierungspotential auszeichnen.LncRNA, als ein Schlüsselelement in nicht-kodierenden RNAs, wird hauptsächlich im Zellkern und Plasma gefunden.Die Entwicklung in der Sequenzierungstechnologie und Bioinformatik ermöglicht die Identifizierung zahlreicher neuer lncRNAs und deren Zuordnung zu biologischen Funktionen.Anhäufungen deuten darauf hin, dass lncRNA in großem Umfang an der epigenetischen Regulation, Transkriptionsregulation und Posttranskriptionsregulation beteiligt ist.
-
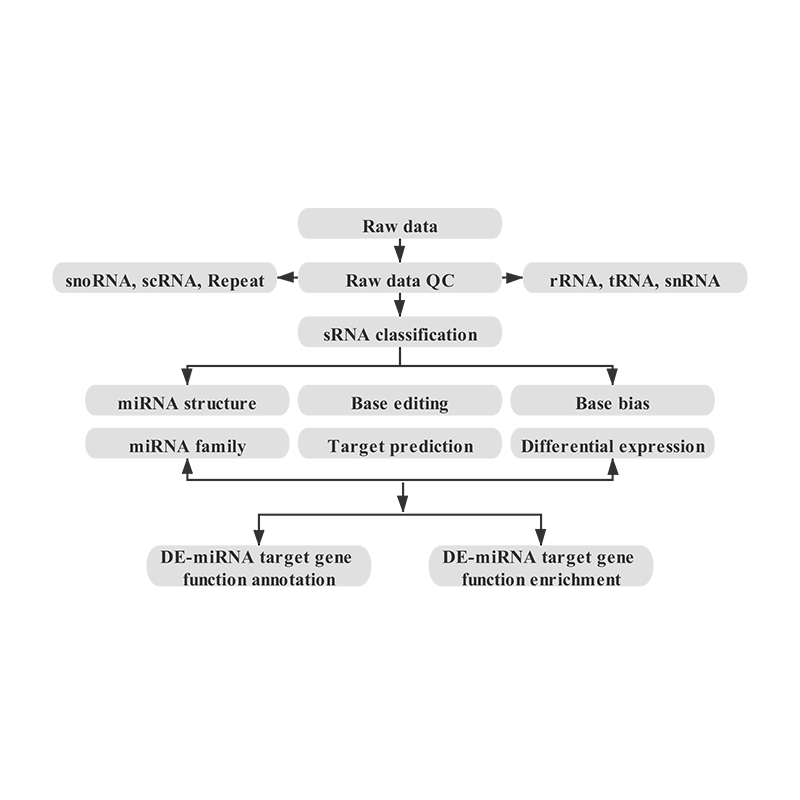
Kleine RNA-Sequenzierung – Illumina
Kleine RNA bezieht sich auf eine Klasse von nicht-codierenden RNA-Molekülen, die normalerweise weniger als 200 nt lang sind, einschließlich Mikro-RNA (miRNA), kleine Interferenz-RNA (siRNA) und Piwi-interagierende RNA (piRNA).
MicroRNA (miRNA) ist eine Klasse endogener kleiner RNA mit einer Länge von etwa 20-24nt, die eine Vielzahl wichtiger regulatorischer Rollen in Zellen spielt.An vielen Lebensprozessen beteiligte miRNA, die eine gewebe- und stadienspezifische Expression aufweist und in verschiedenen Arten hoch konserviert ist.
-
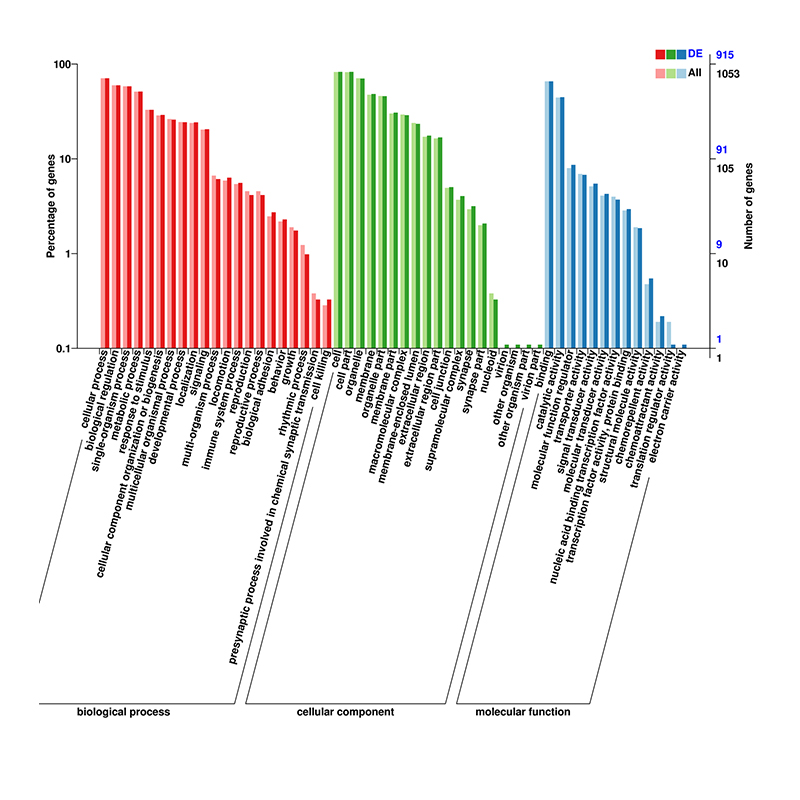
circRNA-Sequenzierung – Illumina
Die Sequenzierung des gesamten Transkriptoms dient dazu, alle Arten von RNA-Molekülen zu profilieren, einschließlich kodierender (mRNA) und nicht-kodierender RNAs (einschließlich lncRNA, circRNA und miRNA), die zu einem bestimmten Zeitpunkt von bestimmten Zellen transkribiert werden.Die Sequenzierung des gesamten Transkriptoms, auch als „Gesamt-RNA-Sequenzierung“ bekannt, zielt darauf ab, umfassende regulatorische Netzwerke auf Transkriptomebene aufzudecken.Unter Ausnutzung der NGS-Technologie stehen Sequenzen ganzer Transkriptomprodukte für die ceRNA-Analyse und gemeinsame RNA-Analyse zur Verfügung, was den ersten Schritt zur funktionellen Charakterisierung darstellt.Aufschlussreiches regulatorisches Netzwerk von circRNA-miRNA-mRNA-basierter ceRNA.
-
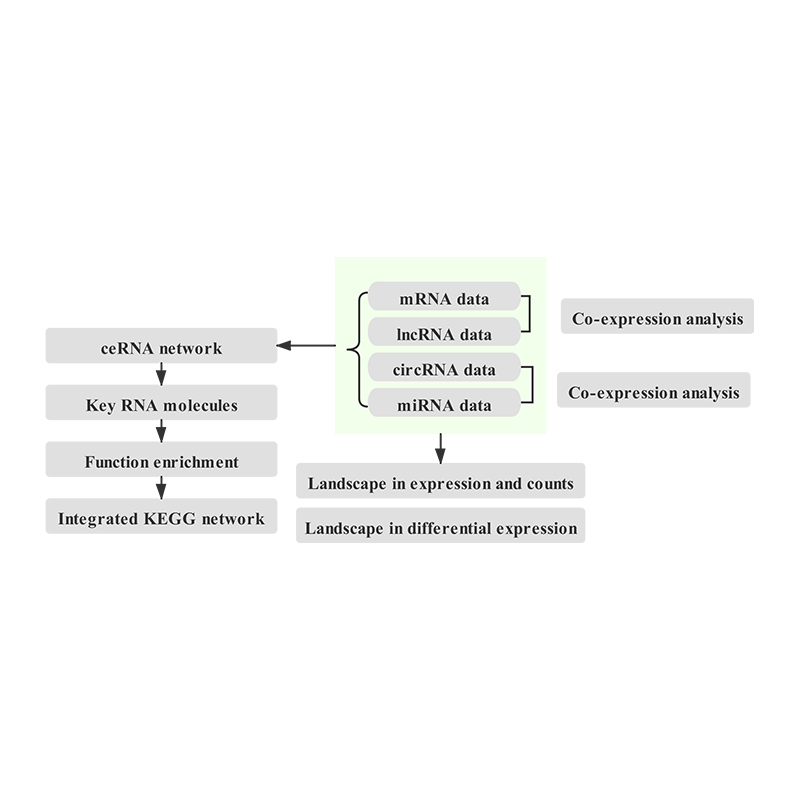
Sequenzierung des gesamten Transkriptoms – Illumina
Die Sequenzierung des gesamten Transkriptoms dient dazu, alle Arten von RNA-Molekülen zu profilieren, einschließlich kodierender (mRNA) und nicht-kodierender RNAs (einschließlich lncRNA, circRNA und miRNA), die zu einem bestimmten Zeitpunkt von bestimmten Zellen transkribiert werden.Die Sequenzierung des gesamten Transkriptoms, auch als „Gesamt-RNA-Sequenzierung“ bekannt, zielt darauf ab, umfassende regulatorische Netzwerke auf Transkriptomebene aufzudecken.Unter Ausnutzung der NGS-Technologie stehen Sequenzen ganzer Transkriptomprodukte für die ceRNA-Analyse und gemeinsame RNA-Analyse zur Verfügung, was den ersten Schritt zur funktionellen Charakterisierung darstellt.Aufschlussreiches regulatorisches Netzwerk von circRNA-miRNA-mRNA-basierter ceRNA.
-

Prokaryotische RNA-Sequenzierung
Die prokaryotische RNA-Sequenzierung verwendet die Sequenzierung der nächsten Generation (NGS), um das Vorhandensein und die Menge von RNA zu einem bestimmten Zeitpunkt aufzudecken, indem das sich ändernde zelluläre Transkriptom analysiert wird.Die prokaryotische RNA-Sequenzierung unseres Unternehmens zielt speziell auf Prokaryoten mit Referenzgenomen ab und bietet Ihnen Transkriptom-Profile, Genstrukturanalysen usw. Sie wurde in großem Umfang in der Grundlagenforschung, Arzneimittelforschung und -entwicklung und mehr angewendet.
Plattform: Illumina NovaSeq 6000
-

Metatranskriptom-Sequenzierung
Die Metatranskriptom-Sequenzierung identifiziert die Genexpression von Mikroben (sowohl Eukaryoten als auch Prokaryoten) in natürlichen Umgebungen (dh Boden, Wasser, Meer, Fäkalien und Darm). Insbesondere ermöglicht Ihnen dieser Service, vollständige Genexpressionsprofile komplexer mikrobieller Gemeinschaften und taxonomische Analysen zu erhalten von Arten, funktionelle Anreicherungsanalyse unterschiedlich exprimierter Gene und mehr.
Plattform: Illumina NovaSeq 6000











