BMKMANU S1000 Romlig transkriptom
BMKMANU S1000 Spatial Transcriptome Technical Scheme
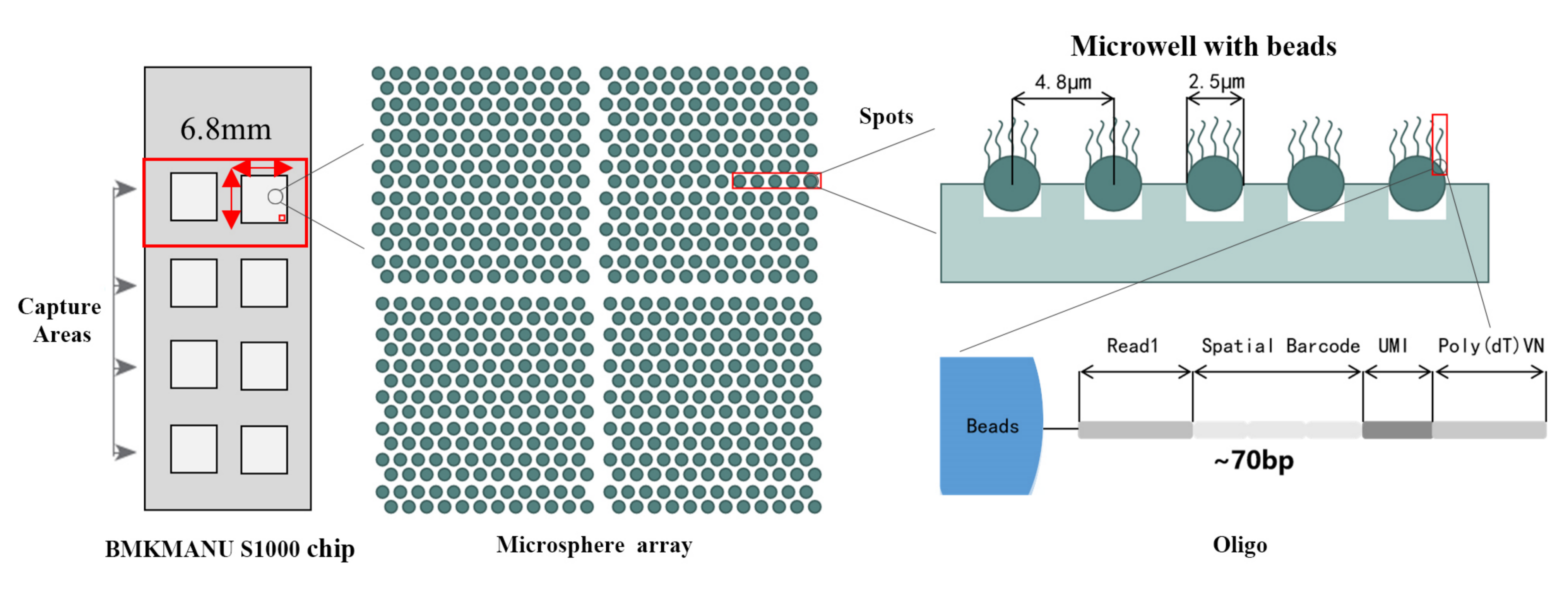
Fordeler med BMKMANU S1000
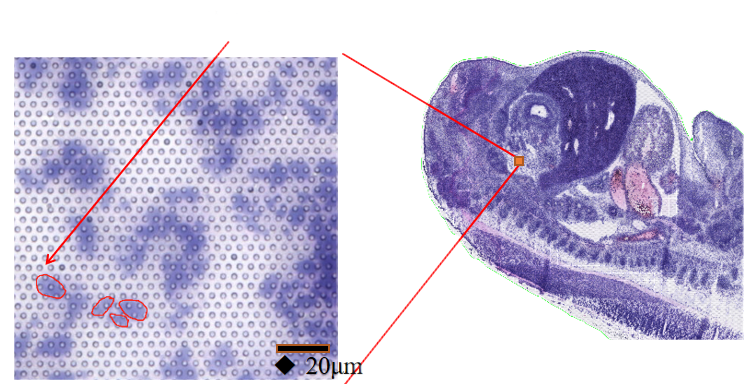
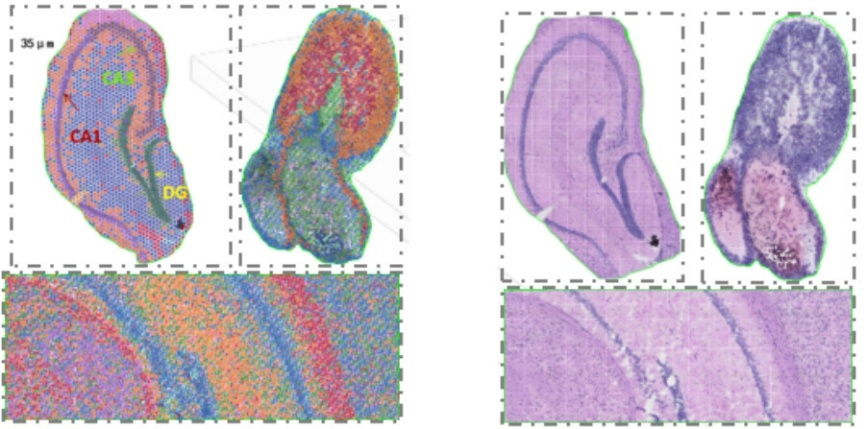
1) Subcellulær oppløsning: Hvert fangstområde inneholdt >2 millioner romlige strekkodede flekker med en diameter på 2,5 µm og en avstand på 5 µm mellom punktsentre, noe som muliggjør romlig transkriptomanalyse med subcellulær oppløsning (5 µm).
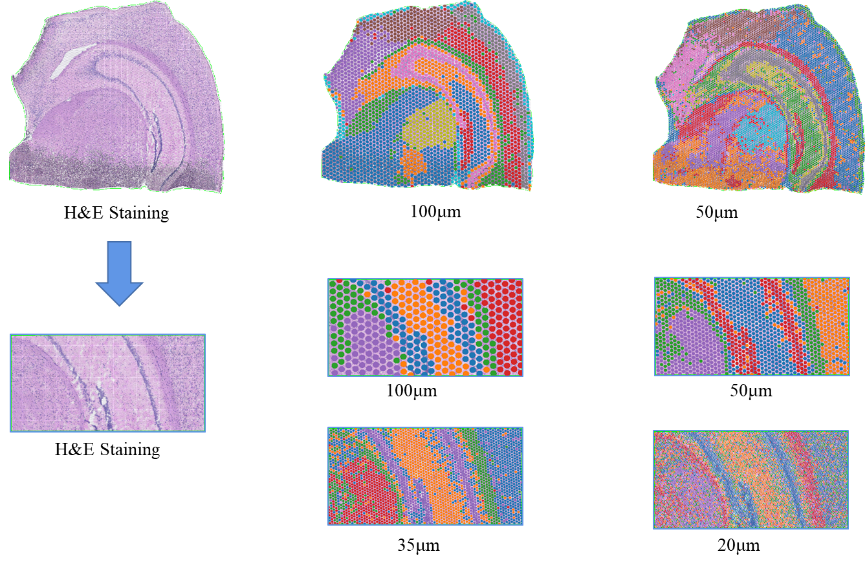
2) Multi-level oppløsningsanalyse: Fleksibel multi-level analyse fra 100 μm til 5 μm for å løse ulike vevsfunksjoner med optimal oppløsning.
3) Omfattende transkriptomprofilering: Transkripsjoner fanget fra hele vevsglasset kan analyseres, uten begrensning på antall målgener og målområde.
Tjenestespesifikasjoner
| Bibliotek | Sekvenseringsstrategi | Datautgang anbefales |
| S1000 cDNA-bibliotek | BMKMANU S1000-Illumina PE150 | 60 Gb/prøve |
Eksempelkrav
| Prøve | Antall | Størrelse | RNA kvalitet |
| OCT innebygd vevsblokk | 2-3 blokker/prøve | Ca.6,8x6,8x6,8 mm3 | RIN≥7 |
For mer informasjon om veiledning for prøveforberedelse og servicearbeidsflyt, kan du gjerne snakke med aBMKGENE-ekspert
Servicearbeidsflyt

Eksperimentdesign

Prøvelevering

Foreksperiment
Romlig strekkoding

Bygging av bibliotek

Sekvensering

Dataanalyse

Ettersalgstjenester
Dataene generert av BMKMANU S1000 analyseres ved hjelp av programvaren "BSTMatrix", som er uavhengig designet av BMKGENE, inkludert:
1) Generering av genekspresjonsmatrise
2) HE bildebehandling
3) Kompatibel med nedstrøms tredjepartsprogramvare for analyse
4) Online "BSTViewer" hjelper deg med å oppnå visualiseringsresultater ved forskjellige oppløsninger.
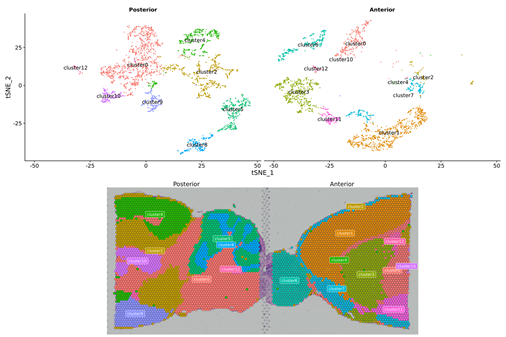
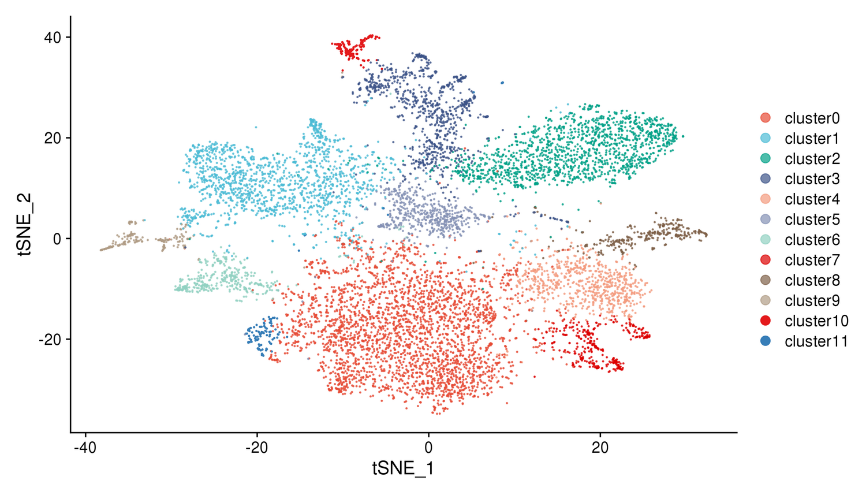
1. Spot clustering
2. Romlig fordeling
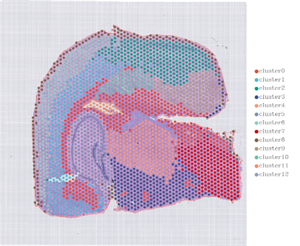
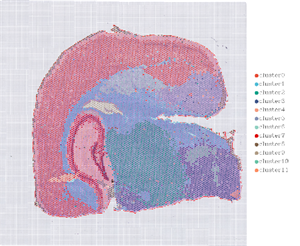
Nmerknad: Vedtaknivå=13 (100 µm, venstre); 7 (50 µm, Ikke sant)
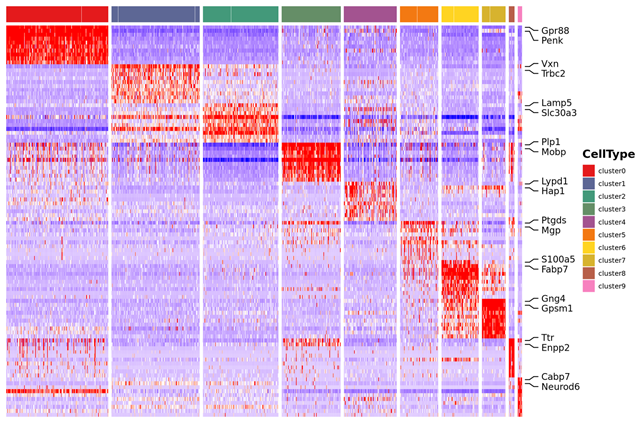
3. Markør uttrykk overflod clustering heatmap
4.Inter-sample dataanalyse