Soppgenom
Tjenestefordeler
● Flere sekvenseringsstrategier tilgjengelig for ulike forskningsmål
● Svært erfaren i soppgenomsamling med over 10 000 pene-komplette genom satt sammen.
● Profesjonelt ettersalgs teknisk supportteam som oppfyller mer spesifikke forskningsbehov.
Tjenestespesifikasjoner
| Service | Sekvenseringsstrategi | Kvalitet garantert | Beregnet behandlingstid |
| Soppfinkart | Illumina 50X+Nanopore 100X | Contig N50≥2 Mb | 35 virkedager |
| PacBio HiFi 30X | |||
| Fungal pene-komplett kart | Illumina 50X+Nanopore 100X(Pacbio HiFi 30X)+Hi-C 100X | Kromosom forankringsforhold >90 % | 45 virkedager |
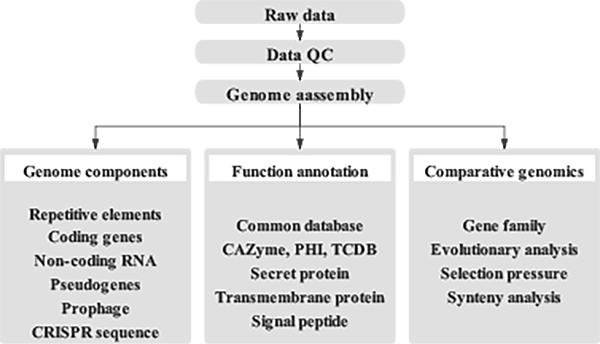
Bioinformatikkanalyser
● Kvalitetskontroll av rådata
● Genomsamling
● Genomkomponentanalyse
● Annotering av genfunksjon
● Komparativ genomisk analyse
Prøvekrav og levering
Eksempelkrav:
TilDNA-ekstrakter:
| Eksempeltype | Beløp | Konsentrasjon | Renhet |
| DNA-ekstrakter | > 1,2 μg | > 20 ng/μl | OD260/280= 1,6-2,5 |
For vevsprøver:
| Prøvetype | Anbefalt prøvebehandling | Prøvelagring og forsendelse |
| Encellet sopp | Observer gjær under mikroskop og samle dem i eksponentiell fase Overfør kulturen (som inneholder ca. 3-4.5e9 celler) til en 1,5 eller 2 ml eppendorf.(Hold deg på is) Sentrifuger røret i 1 min ved 14000 g for å samle bakterier og fjern supernatanten forsiktig Forsegl røret og frys bakteriene i flytende nitrogen i minst 1-3 timer.Oppbevar røret i -80 ℃ kjøleskap. | Frys prøvene i flytende nitrogen i 3-4 timer og oppbevar i flytende nitrogen eller -80 grader til langtidsreservasjon.Prøvefrakt med tørris er nødvendig. |
| Makrosopp | Vev i aktivt voksende fase anbefales. Skyll vevet med endotoksinfritt vann, deretter 70 % etonal. Lagre prøven i kryorør. |
Servicearbeidsflyt

Prøvelevering

Bygging av bibliotek

Sekvensering

Dataanalyse

Ettersalgstjenester
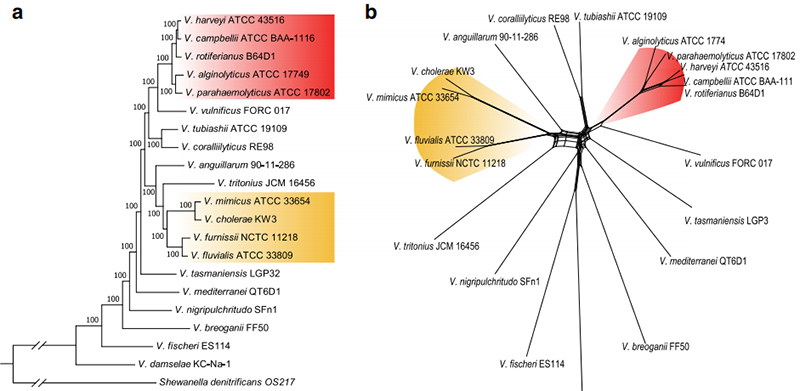
1. Cirkosdiagram over soppgenomiske komponenter
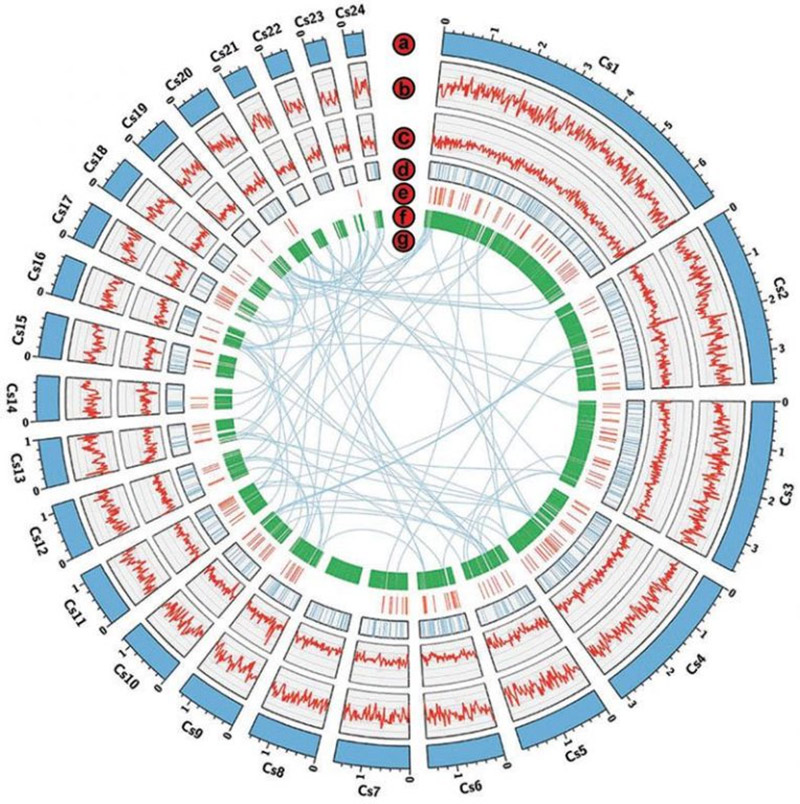
2.Komparativ genomikkanalyse: Fylogenetisk tre