
Plante/dyr De Novo Genome Sequencing
Tjenestefordeler

Utvikling av sekvenseringsplattformer og bioinformatikk ide novogenomsamling
(Amarasinghe SL et al.,Genombiologi, 2020)
● Konstruere nye genomer og forbedre eksisterende referansegenomer for arter av interesse.
● Høyere nøyaktighet, kontinuitet og fullstendighet i montering
● Konstruere grunnleggende ressurs for forskning innen sekvenspolymorfisme, QTL-er, genredigering, avl, etc.
● Utstyrt med hele spekteret av tredjegenerasjons sekvenseringsplattformer: ett-stopps genommonteringsløsning
● Fleksible sekvenserings- og sammenstillingsstrategier som oppfyller forskjellige genomer med forskjellige funksjoner
● Høyt dyktig bioinformatikerteam med stor erfaring i komplekse genomsammenstillinger, inkludert polyploider, gigantiske genomer, etc.
● Over 100 vellykkede saker med en akkumulert publisert effektfaktor på over 900
● Omløpstid så raskt som 3 måneder for kromosomnivåsamling av genom.
● Solid teknisk støtte med en rekke patenter og opphavsrettigheter til programvare innen både eksperimentell side og bioinformatikk.
Tjenestespesifikasjoner
|
Innhold
|
Plattform
|
Les lengde
|
Dekning
|
| Genomundersøkelse
| Illumina NovaSeq
| PE150
| ≥ 50X
|
| Genomsekvensering
| PacBio Revio
| 15 kb HiFi-leser
| ≥ 30X
|
| Hei-C
| Illumina NovaSeq
| PE150
| ≥100X
|
Arbeidsflyt

Prøvekrav og levering
Eksempelkrav:
| Arter | Vev | For PacBio | For Nanopore |
| Dyr | Viscerale organer (lever, milt, etc.) | ≥ 1,0 g | ≥ 3,5 g |
| Muskel | ≥ 1,5 g | ≥ 5,0 g | |
| Blod fra pattedyr | ≥ 1,5 ml | ≥ 5,0 ml | |
| Blod fra fisk eller fugler | ≥ 0,2 ml | ≥ 0,5 ml | |
| Planter | Friske blader | ≥ 1,5 g | ≥ 5,0 g |
| Kronblad eller stilk | ≥ 3,5 g | ≥ 10,0 g | |
| Røtter eller frø | ≥ 7,0 g | ≥ 20,0 g | |
| Celler | Cellekultur | ≥ 3×107 | ≥ 1×108 |
Anbefalt prøvelevering
Beholder: 2 ml sentrifugerør (tinnfolie anbefales ikke)
For de fleste prøvene anbefaler vi å ikke konservere i etanol.
Prøvemerking: Prøver må være tydelig merket og identiske med innsendt prøveinformasjonsskjema.
Forsendelse: Tørris: Prøver må først pakkes i poser og begraves i tørris.
Servicearbeidsflyt

Eksperimentdesign

Prøvelevering

DNA-ekstraksjon

Bygging av bibliotek

Sekvensering

Dataanalyse

Ettersalgstjenester
*Demoresultater vist her er alle fra genomer publisert med Biomarker Technologies
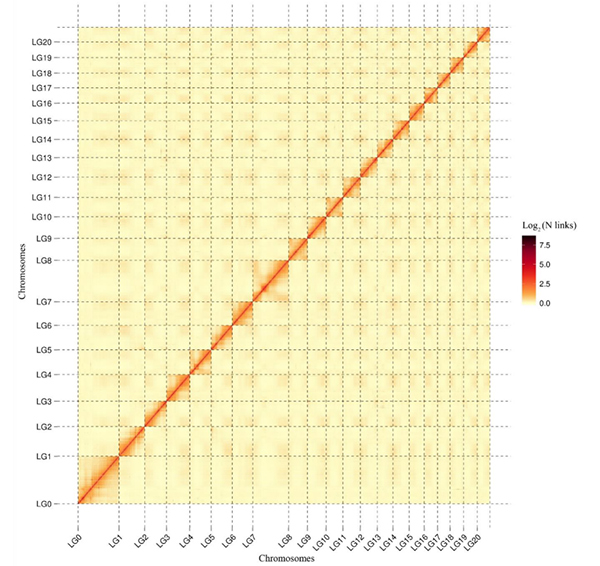
1.Circos på kromosom-nivå genom montering avG. rotundifoliumav Nanopore sekvenseringsplattform
Wang M et al.,Molekylærbiologi og evolusjon, 2021
2. Statistikk over Weining ruggenomsamling og merknad
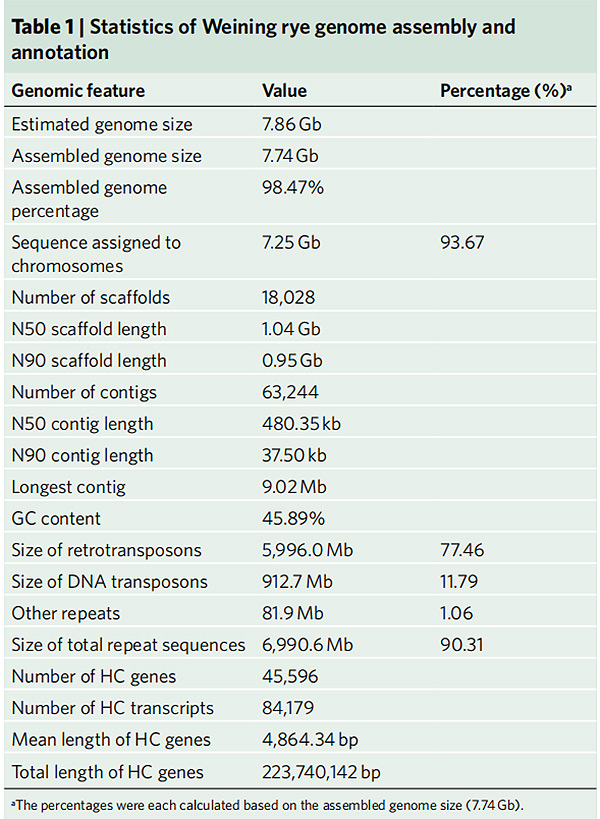
Li G et al.,Naturgenetikk, 2021
3.Genprediksjon avSechium edulegenom, avledet fra tre prediksjonsmetoder:De novoprediksjon, Homologi-basert prediksjon og RNA-Seq-databasert prediksjon
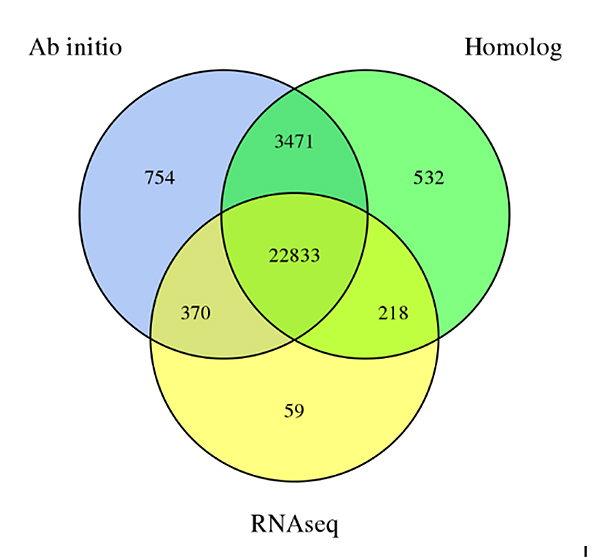
Fu A et al.,Hagebruksforskning, 2021
4. Identifikasjon av intakte lange terminale gjentakelser i tre bomullsgenomer
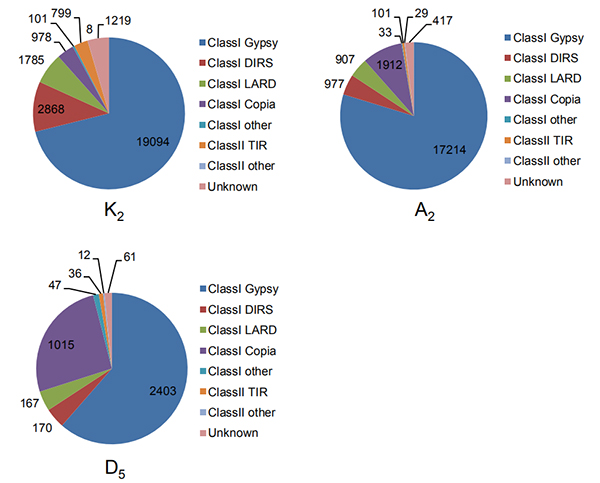
Wang M et al.,Molekylærbiologi og evolusjon, 2021
5.Hi-C varmekart overC. acuminatagenom som viser genom-omfattende alt-for-alle interaksjoner.Intensiteten til Hi-C-interaksjoner er proporsjonal med lineær avstand mellom contigs.En ren rett linje på dette varmekartet indikerer en svært nøyaktig forankring av contigs på kromosomer.(Contig forankringsforhold: 96,03 %)
kang M et al.,Naturkommunikasjon,2021
BMK sak
En genomsamling av høy kvalitet fremhever ruggenomiske egenskaper og agronomisk viktige gener
Publisert: Naturgenetikk, 2021
Sekvenseringsstrategi:
Genomsamling: PacBio CLR-modus med 20 kb bibliotek (497 Gb, ca. 63×)
Sekvenskorrigering: NGS med 270 bp DNA-bibliotek (430 Gb, ca. 54×) på Illumina-plattformen
Contigs forankring: Hi-C-bibliotek (560 Gb, ca. 71×) på Illumina-plattformen
Optisk kart: (779,55 Gb, ca. 99×) på Bionano Irys
Nøkkelresultater
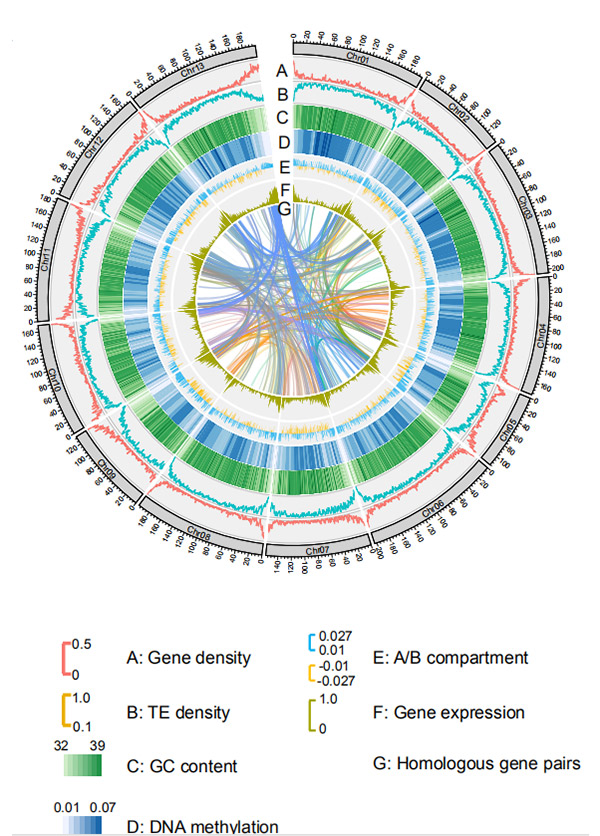
1. En samling av Weining ruggenom ble publisert med total genomstørrelse på 7,74 Gb (98,74 % av estimert genomstørrelse ved flowcytometri).Stillas N50 i denne enheten oppnådde 1,04 Gb.93,67% av contigs ble vellykket forankret på 7 pseudo-kromosomer.Denne forsamlingen ble evaluert av koblingskart, LAI og BUSCO, noe som resulterte i høye skårer i alle evalueringer.
2. Videre studier på komparativ genomikk, genetisk koblingskart, transkriptomiske studier ble utført på basis av dette genomet.En rekke egenskaper relaterte genomiske egenskaper ble avslørt, inkludert genomomfattende gendupliseringer og deres innvirkning på stivelsesbiosyntesegener;fysisk organisering av komplekse prolamin-loki, genuttrykkstrekk som ligger til grunn for tidlig overskriftstrekk og antatt domestiseringsassosierte kromosomale regioner og loki i rug.
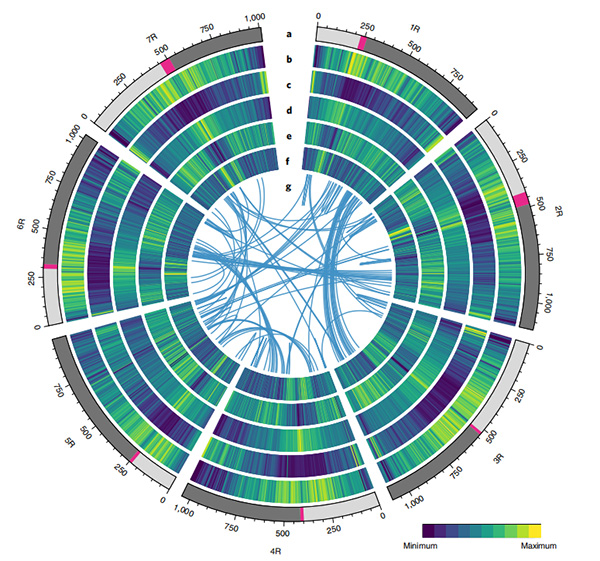 Circos-diagram over genomiske trekk ved Weining ruggenom | 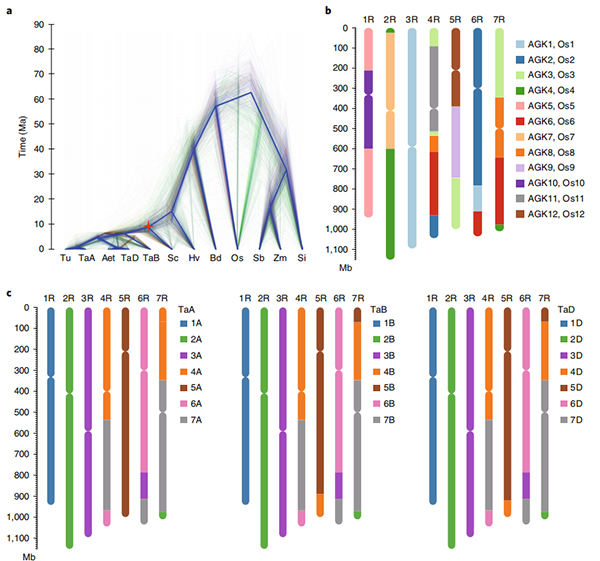 Evolusjons- og kromosomsyntenyanalyser av ruggenomet |
Li, G., Wang, L., Yang, J.et al.En genomsamling av høy kvalitet fremhever ruggenomiske egenskaper og agronomisk viktige gener.Nat Genet 53,574–584 (2021).
https://doi.org/10.1038/s41588-021-00808-z










