Komparativ genomikk
Tjenestefordeler
● Omfattende analysepakke, som inneholder de åtte vanligste analysene
● Høy pålitelighet i analyse med detaljert og lett forståelig tolkning av resultater
● Godt utformede publiseringsklare figurer
● Høyt dyktige bioinformatikkteam oppfyller ulike personaliserte analysekrav
● Kortere behandlingstid med høyere nøyaktighet i analysen
● Rikelig erfaring med over 90 vellykkede saker med akkumulert publisert effektfaktor på over 900
Tjenestespesifikasjoner
| Beregnet behandlingstid | Antall arter | Analyser |
| 30 virkedager | 6 - 12 | Genfamilieklynger Gene familie utvidelse og sammentrekning Fylogenetisk trekonstruksjon Divergenstidsestimering (fossilkalibrering kreves) LTR-innsettingstid (for planter) Helgenomduplisering (for planter) Selektivt trykk Synteny-analyse |
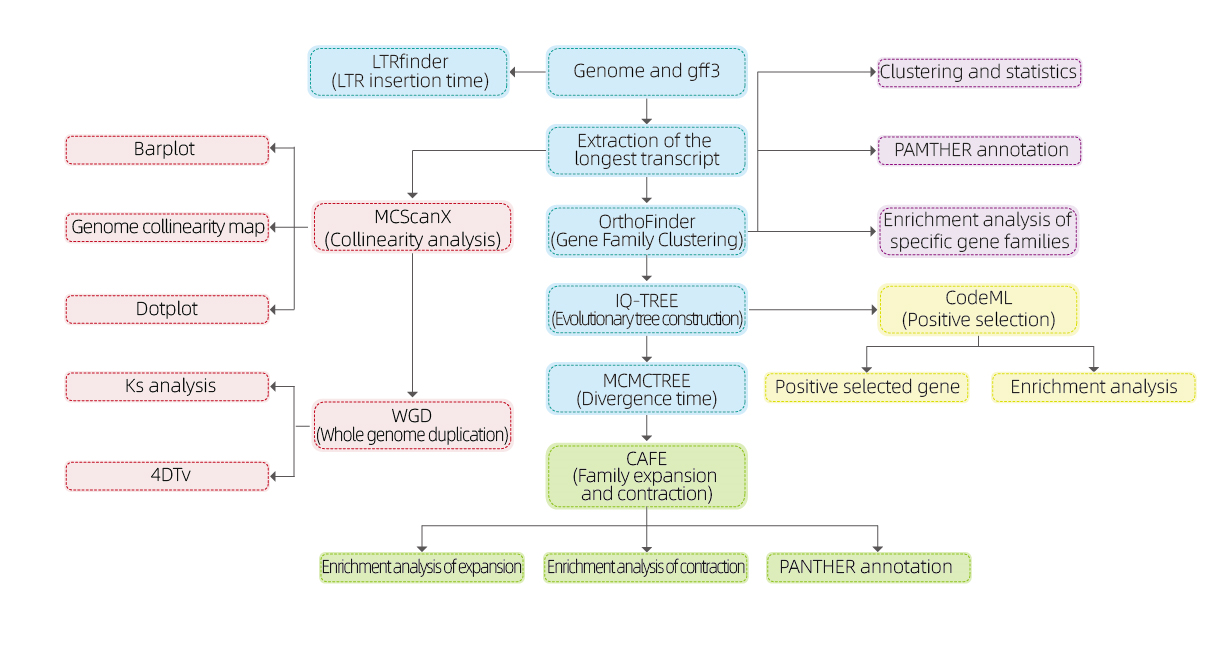
Bioinformatikkanalyser
● Genfamilie
● Fylogenetikk
● Divergenstid
● Selektivt trykk
● Synteny-analyse
Prøvekrav og levering
Eksempelkrav:
Vev eller DNA for genomsekvensering og montering
For vev
| Arter | Vev | undersøkelse | PacBio CCS |
| Dyr | Visceralt vev | 0,5 ~ 1 g | ≥ 3,5 g |
| Muskelvev | |||
| ≥ 5,0 g | |||
| ≥ 5,0 ml | |||
| Pattedyrblod | |||
| ≥ 0,5 ml | |||
| Fjærkre/fiskeblod | |||
| Anlegg | Ferskt blad | 1 ~ 2 g | ≥ 5,0 g |
| Kronblad/Stengel | 1 ~ 2 g | ≥ 10,0 g | |
| Rot/frø | 1 ~ 2 g | ≥ 20,0 g | |
| Celler | Dyrket celle | - | ≥ 1 x 108 |
Data
Genomsekvensfiler (.fasta) og merknadsfiler (.gff3) av nært beslektede arter
Servicearbeidsflyt

Eksperimentdesign

Prøvelevering

Bygging av bibliotek

Sekvensering

Dataanalyse

Ettersalgstjenester
*Demoresultatene vist her er alle fra genomer publisert med Biomarker Technologies
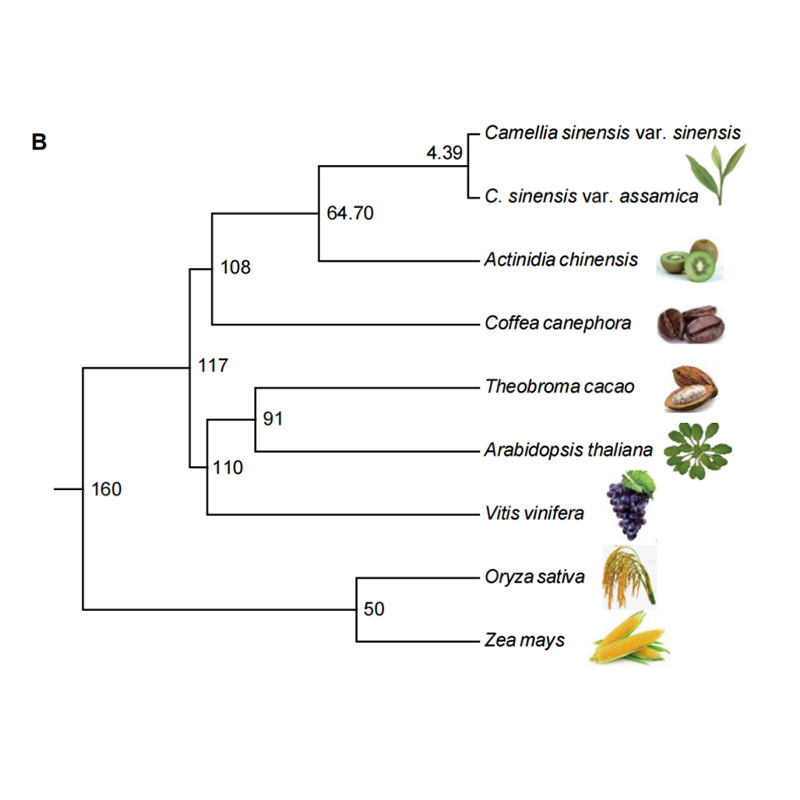
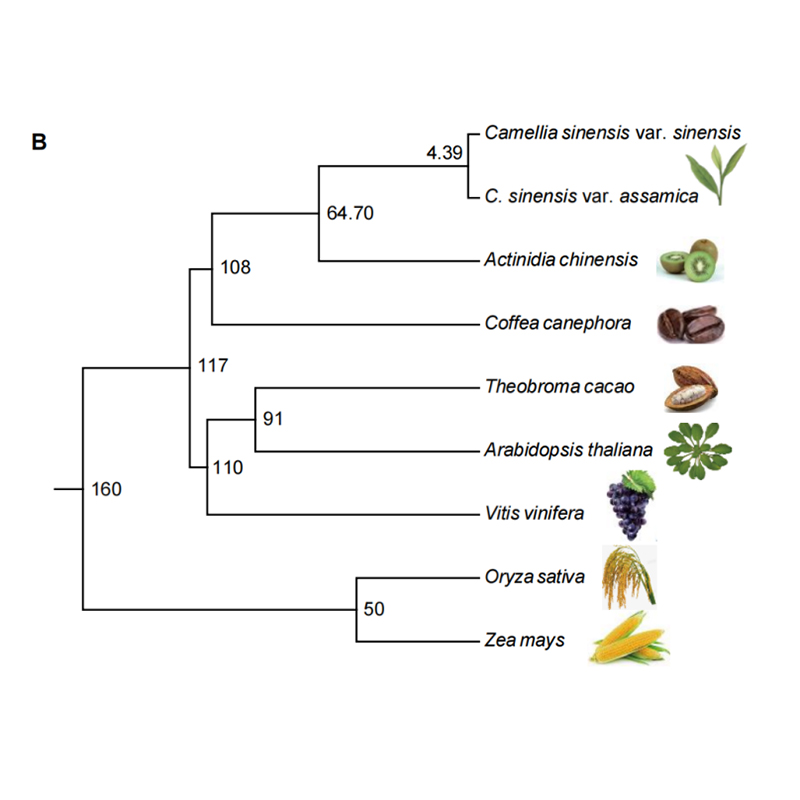
1.LTR-innsettingstidsestimering: Figuren viste en unik bimodal fordeling i LTR-RTs innsettingstider i Weining ruggenomet, sammenlignet med andre arter.Den siste toppen dukket opp for rundt 0,5 millioner år siden.
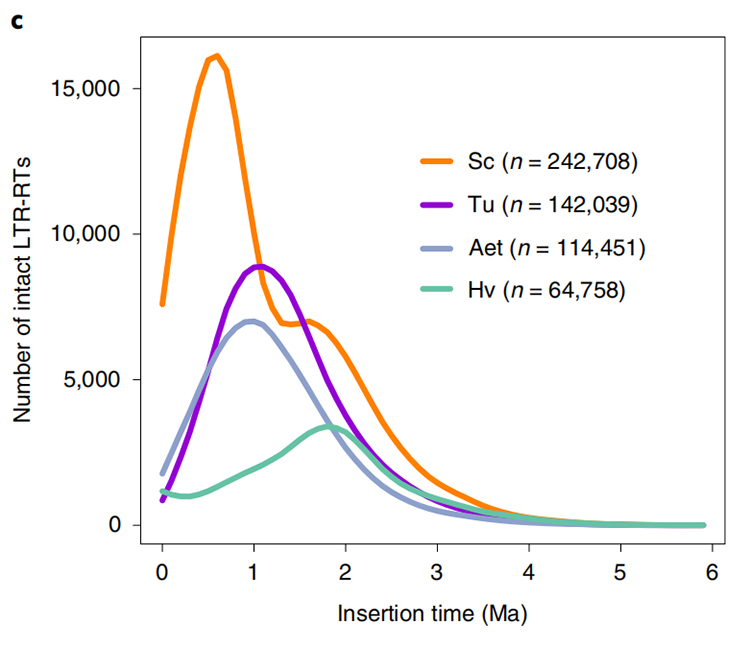
Li Guang et al.,Naturgenetikk, 2021
2.Fylogeni og genfamilieanalyse på chayote (Sechium edule): Ved å analysere chayote og de andre 13 beslektede artene i genfamilien, ble Chayote funnet å være nærmest beslektet med slangegresskar (Trichosanthes anguina).Chayote avledet fra slangegresskar i rundt 27-45 Mya og helgenomduplisering (WGD) ble observert i chayote i 25±4 Mya, som er den tredje WGD-hendelsen i cucuibitaceae.
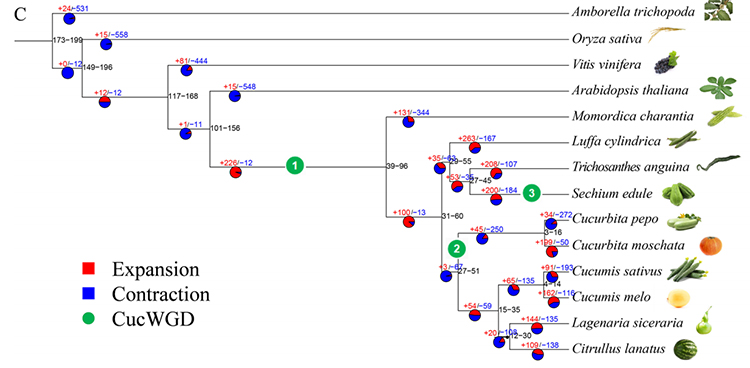
Fu A et al.,Hagebruksforskning, 2021
3. Synteny-analyse: Noen gener relatert til fytohormoner i fruktutvikling ble funnet i chayote, slangegresskar og squash.Korrelasjonen mellom chayote og squash er litt høyere enn mellom chayote og slangegresskar.
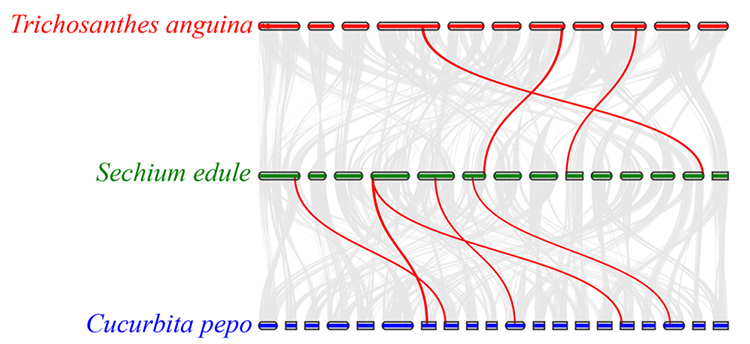
Fu A et al.,Hagebruksforskning, 2021
4.Genfamilieanalyse: KEGG-anriking på genfamilieutvidelse og sammentrekning i G.thurberi- og G.davidsonii-genomer viste at steroidbiosyntese og brassinosteroidbiosyntese-relaterte gener ble utvidet.
Yang Z et al.,BMC Biologi, 2021
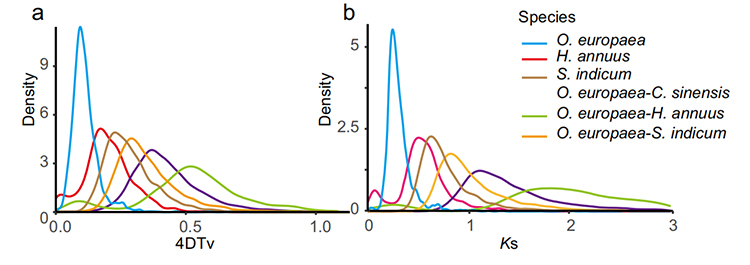
5. Helgenomduplikasjonsanalyse: 4DTV- og Ks-distribusjonsanalyse viste helgenomdupliseringshendelse.Topper av intraspecies vist dupliseringshendelser.Topper av interspecies vist artiasjonshendelser.Analysen indikerte at sammenlignet med de tre andre nært beslektede artene, gikk O. europaea gjennom en storskala genduplisering nylig.

Rao G et al.,Hagebruksforskning, 2021
BMK sak
Rose uten prickle: genomisk innsikt knyttet til fuktighetstilpasning
Publisert: National Science Review, 2021
Sekvenseringsstrategi:
'BasyesTornløs' (R.Wichurainan) genom:
Ca.93 X PacBio + ca.90 X Nanopore + 267 X Illumina
Nøkkelresultater
1. R.wichuraiana-genomet av høy kvalitet ble konstruert ved bruk av langleste sekvenseringsteknikker, som gir en samling på 530,07 Mb (estimert genomstørrelse var omtrent 525,9 Mb ved flowcytometri og 525,5 ved genomundersøkelse; Heterozygositeten var rundt 1,03%).BUSCOs estimerte poengsum var 93,9 %.Sammenlignet med "Old blush" (haploOB), ble kvaliteten og fullstendigheten til dette genomet bekreftet av base enkeltbasenøyaktighet og LTR-sammenstillingsindeks (LAI=20,03).R.wichuriana-genomet inneholder 32 674 proteinkodende gener.
2. Multi-omics felles analyse, bestående av komparativ genomikk, transkriptomikk, QTL-analyse av genetisk populasjon, avslørte den avgjørende spesifikasjonen mellom R. wichuraiana og Rosa chinensis.Også ekspresjonsvariasjon av beslektede gener i QTL var sannsynligvis assosiert med stamprickle-mønster.

Sammenlignende genomisk analyse mellom Basye's Thornless og Rosa chinensis, inkludert synteny-analyse, genfamilieklynge, ekspansjons- og sammentrekningsanalyse, avslørte et stort antall variasjoner, som var relatert til viktige egenskaper hos roser.Den unike utvidelsen i NAC- og FAR1/FRS-genfamilien var svært sannsynlig forbundet med resistens mot svart flekk.
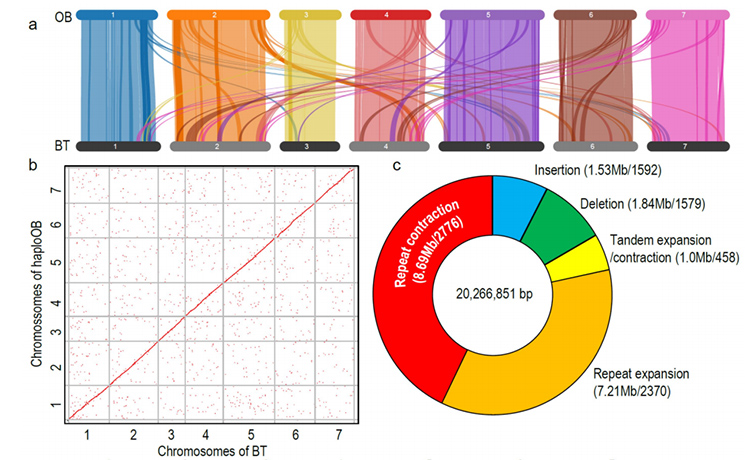

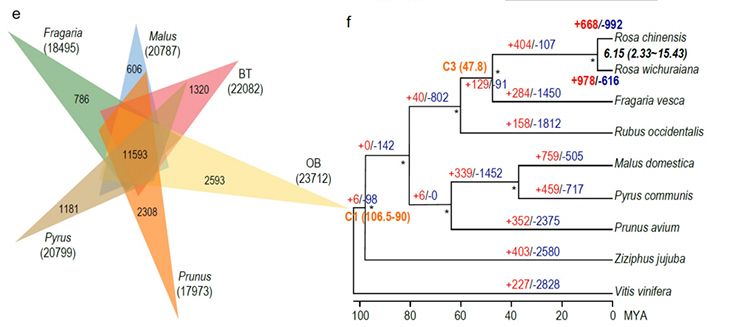
Komparativ genomikkanalyse mellom BT- og haploOB-genomer.
Zhong, M., et al."Rose uten prikkel: genomisk innsikt knyttet til fuktighetstilpasning"National Science Review, 2021;, nwab092.