NGS-mRNA (referanse)
Transkriptom er koblingen mellom genomisk genetisk informasjon og proteomet for biologisk funksjon.Transkripsjonsnivåregulering er den viktigste og mest studerte reguleringsmåten for organismer.Transkriptomsekvensering kan sekvensere transkriptomet på et hvilket som helst tidspunkt eller under alle forhold, med en oppløsning nøyaktig til et enkelt nukleotid. Den kan dynamisk gjenspeile nivået av gentrankripsjon, samtidig identifisere og kvantifisere sjeldne og normale transkripsjoner, og gi den strukturelle informasjonen til prøve spesifikke transkripsjoner.
For tiden har transkriptomsekvenseringsteknologi blitt mye brukt innen agronomi, medisin og andre forskningsfelt, inkludert dyre- og planteutviklingsregulering, miljøtilpasning, immuninteraksjon, genlokalisering, artsgenetisk evolusjon og tumor- og genetisk sykdomsdeteksjon.
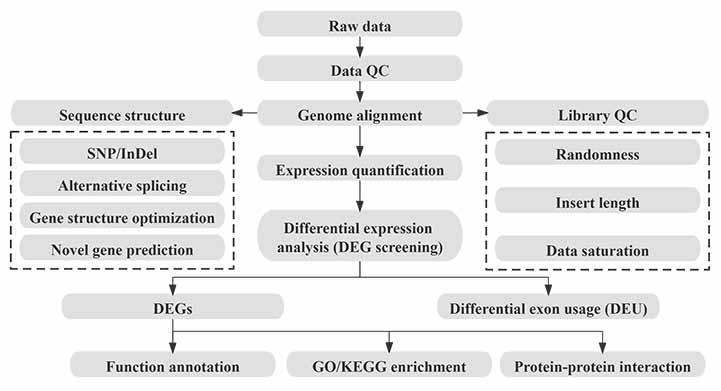
Bioinformatikk
Arbeidsflyt for bioinformatikk