Metagenomics (NGS)
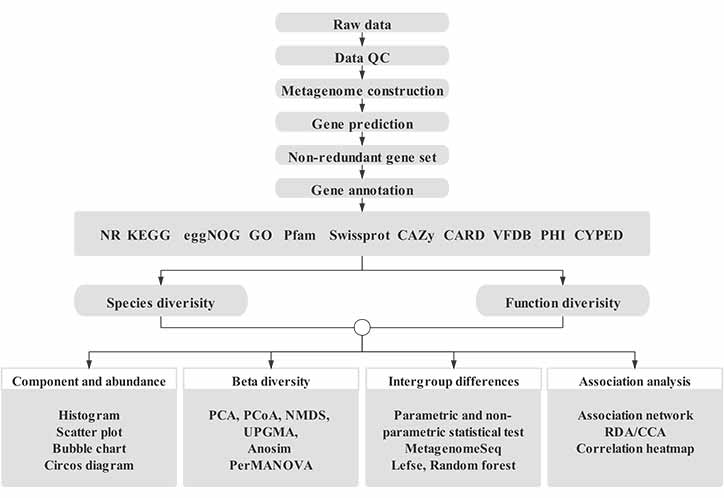
Denne analyseplattformen er designet for metagenomisk dataanalyse med hagle på grunnlag av mange års erfaring.Den består av integrert arbeidsflyt som inneholder forskjellige vanlige metagenomiske analyser, inkludert databehandling, studier på artsnivå, studier på genfunksjonsnivå, metagenombinning osv. I tillegg er tilpassede datautvinningsverktøy tilgjengelige på standard analysearbeidsflyt, inkludert gen- og artsspørring , parameterinnstilling, personlig figurgenerering, etc.
Transcipit identifikasjon
Arbeidsflyt for bioinformatikk