Hel transkriptomsekvensering – Illumina
Egenskaper
● Dobbelt bibliotek for å sekvensere det komplette transkriptomet: rRNA-uttømming etterfulgt av PE150 bibliotek forberedelse og størrelse valg etterfulgt av SE50 bibliotek forberedelse
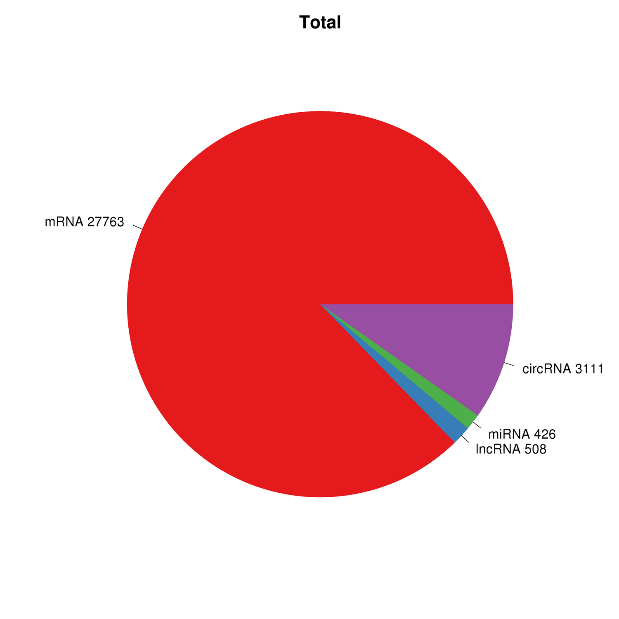
● Fullstendig bioinformatikkanalyse av mRNA, lncRNA, circRNA og miRNA i separate bioinformatikkrapporter
● Felles analyse av all RNA-ekspresjon i kombinert rapport, inkludert analyse av ceRNA-nettverk.
Tjenestefordeler
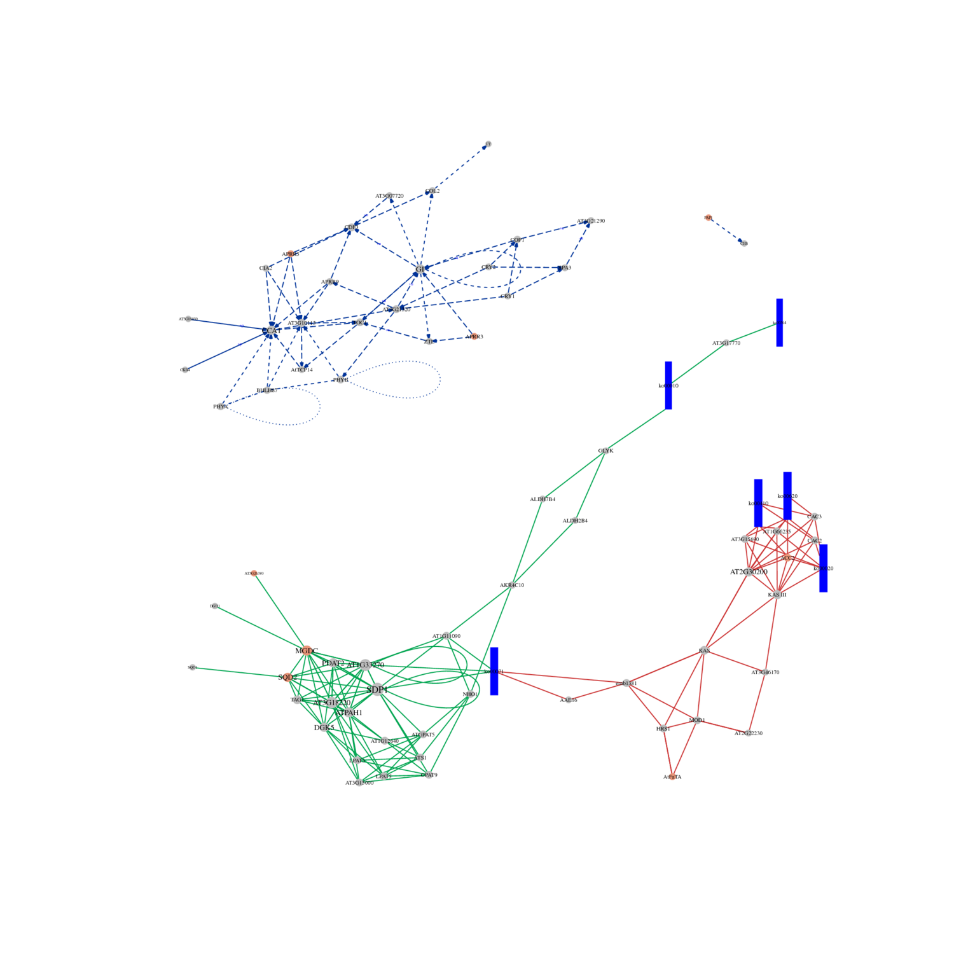
●Dybdeanalyse av regulatoriske nettverk: ceRNA-nettverksanalyse er aktivert ved felles sekvensering av mRNA, lncRNA, circRNA og miRNA og av en uttømmende bioinformatisk arbeidsflyt.
●Omfattende merknad: vi bruker flere databaser for funksjonelt å kommentere de differensielt uttrykte genene (DEGs) og utføre den tilsvarende berikelsesanalysen, og gir innsikt i de cellulære og molekylære prosessene som ligger til grunn for transkriptomresponsen.
●Omfattende kompetanse: Med en merittliste med vellykket lukking av over 2000 hele transkriptomprosjekter i ulike forskningsdomener, bringer teamet vårt et vell av erfaring til hvert prosjekt.
●Streng kvalitetskontroll: Vi implementerer kjernekontrollpunkter på tvers av alle stadier, fra prøve- og bibliotekforberedelse til sekvensering og bioinformatikk.Denne grundige overvåkingen sikrer levering av konsekvent høykvalitetsresultater.
● Omfattende merknad: vi bruker flere databaser for funksjonelt å kommentere de differensielt uttrykte genene (DEGs) og utføre den tilsvarende berikelsesanalysen, og gir innsikt i de cellulære og molekylære prosessene som ligger til grunn for transkriptomresponsen.
●Support etter salg: Vårt engasjement strekker seg utover prosjektgjennomføring med en 3-måneders ettersalgsserviceperiode.I løpet av denne tiden tilbyr vi prosjektoppfølging, feilsøkingshjelp og spørsmål og svar-økter for å løse eventuelle spørsmål knyttet til resultatene
Prøvekrav og levering
| Bibliotek | Sekvenseringsstrategi | Data anbefales | Kvalitetskontroll |
| rRNA utarmet | Illumina PE150 | 16 Gb | Q30≥85 % |
| Størrelse valgt | Illumina SE50 | 10-20M lesninger |
Eksempelkrav:
Nukleotider:
| Kons.(ng/μl) | Mengde (μg) | Renhet | Integritet |
| ≥ 100 | ≥ 1 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Begrenset eller ingen protein- eller DNA-kontaminering vist på gel. | Planter: RIN≥6,5 Dyr: RIN≥7,0 5,0≥28S/18S≥1,0; begrenset eller ingen grunnlinjehøyde |
Anbefalt prøvelevering
Container:
2 ml sentrifugerør (tinnfolie anbefales ikke)
Prøvemerking: Gruppe+replikat f.eks. A1, A2, A3;B1, B2, B3... ...
Forsendelse:
1.Tørris: Prøver må pakkes i poser og begraves i tørris.
2.RNAstabile rør: RNA-prøver kan tørkes i RNA-stabiliseringsrør (f.eks. RNAstable®) og sendes i romtemperatur.
Servicearbeidsflyt

Eksperimentdesign

Prøvelevering

RNA-ekstraksjon

Bygging av bibliotek

Sekvensering

Dataanalyse

Ettersalgstjenester
Bioinformatikk
Oversikt over RNA-uttrykk
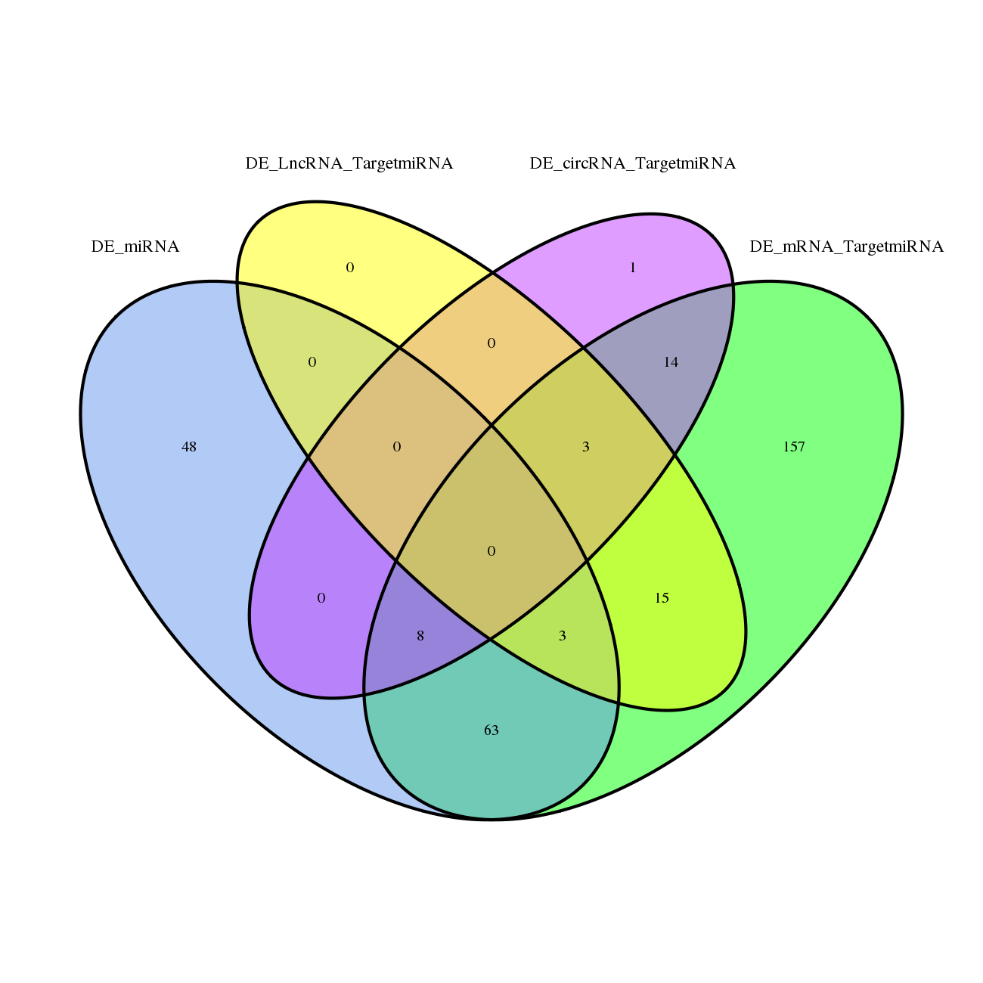
Differensielt uttrykte gener
ceRNA-analyse
Utforsk forskningsfremskritt som tilrettelegges av BMKGene 'hele transkriptomsekvenseringstjenester gjennom en kuratert samling av publikasjoner.
Dai, Y. et al.(2022) 'Omfattende ekspresjonsprofiler av mRNA, lncRNA og miRNA i Kashin-Beck sykdom identifisert ved RNA-sekvensering', Molecular Omics, 18(2), s. 154–166.doi: 10.1039/D1MO00370D.
Liu, N. nan et al.(2022) 'Fulllengde transkriptomer analyse av kuldemotstanden til Apis cerana i Changbai-fjellet under overvintringsperioden.', Gene, 830, s. 146503–146503.doi: 10.1016/J.GENE.2022.146503.
Wang, XJ et al.(2022) 'Multi-Omics integrasjonsbasert prioritering av konkurrerende endogene RNA-reguleringsnettverk i småcellet lungekreft: Molecular Characteristics and Drug Candidates', Frontiers in Oncology, 12, s.904865. doi: 10.3389/FONC.2022.904865/BIBTEX.
Xu, P. et al.(2022) 'Integrert analyse av lncRNA/circRNA-miRNA-mRNA-ekspresjonsprofilene avslører ny innsikt i potensielle mekanismer som respons på rotknute-nematoder i peanøtt', BMC Genomics, 23(1), s. 1–12.doi: 10.1186/S12864-022-08470-3/FIGURE/7.
Yan, Z. et al.(2022) "Hele-transkriptom-RNA-sekvensering fremhever de molekylære mekanismene assosiert med opprettholdelse av etterhøstkvalitet i brokkoli ved rød LED-bestråling", Postharvest Biology and Technology, 188, s.111878. doi: 10.1016/J.POSTHARVBIO.2022.111878.