Genomomfattende assosiasjonsanalyse
1. Tjenestefordeler
● Rikelig med prosjekttilfeller: siden etableringen i 2009 har BMKGENE fullført hundrevis av artsprosjekter i populasjons-GWAS-forskning, hjulpet forskere med å publisere mer enn 100 artikler, og den kumulative effektfaktoren nådde 500.
● Profesjonelle analytikere.
● Kort analysesyklus.
● Nøyaktig datautvinning.
2. Tjenestespesifikasjoner
| Type | Befolkningsskala | Sekvenseringsstrategi og dybde |
| SLAF-GWAS | Prøvenummer ≥200 | Genomstørrelse < 400M, med ref-genom, WGS anbefales |
| Genomstørrelse ≤ 1G, 100K Tags og 10X | ||
| 1G ≤ Genomstørrelse ≤ 2G, 200K Tags og 10X | ||
| Genomstørrelse > 2G, 300K Tags og 10X | ||
| WGS-GWAS | Prøvenummer ≥200 | 10X for hver prøve |
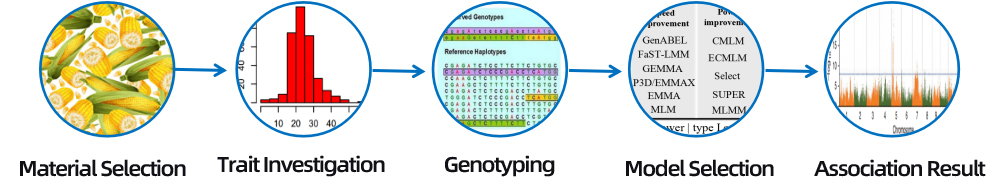
3. Materialvalg



Ulike varianter, underarter, landraser/genbanker/blandingsfamilier/ville ressurser
Ulike varianter, underarter, landraser
Halvsøskenfamilie/helsøskenfamilie/ville ressurser
4. Bioinformasjonsanalyse
● Genomomfattende assosiasjonsanalyse
● Analyse og screening av signifikant SNP
● Funksjonell annotering av kandidatgen
5. Servicearbeidsflyt

Eksperimentdesign

Prøvelevering

RNA-ekstraksjon

Bygging av bibliotek

Sekvensering

Dataanalyse

Ettersalgstjenester
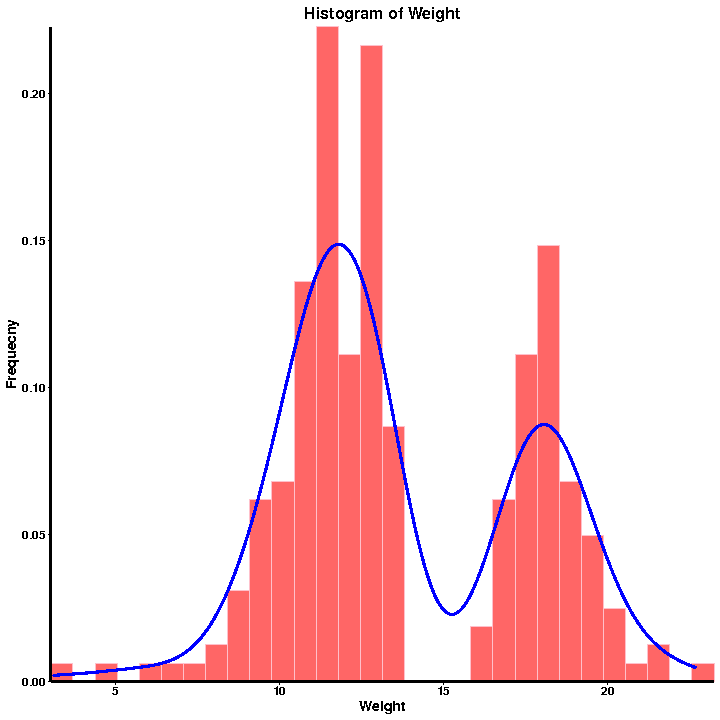
en.Fenotype QC
Frekvensfordelingshistogram
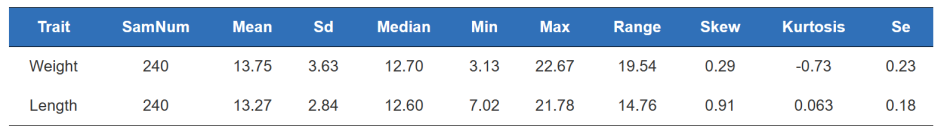
Fenotypestatistikk
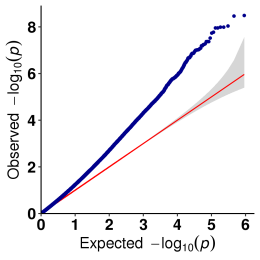
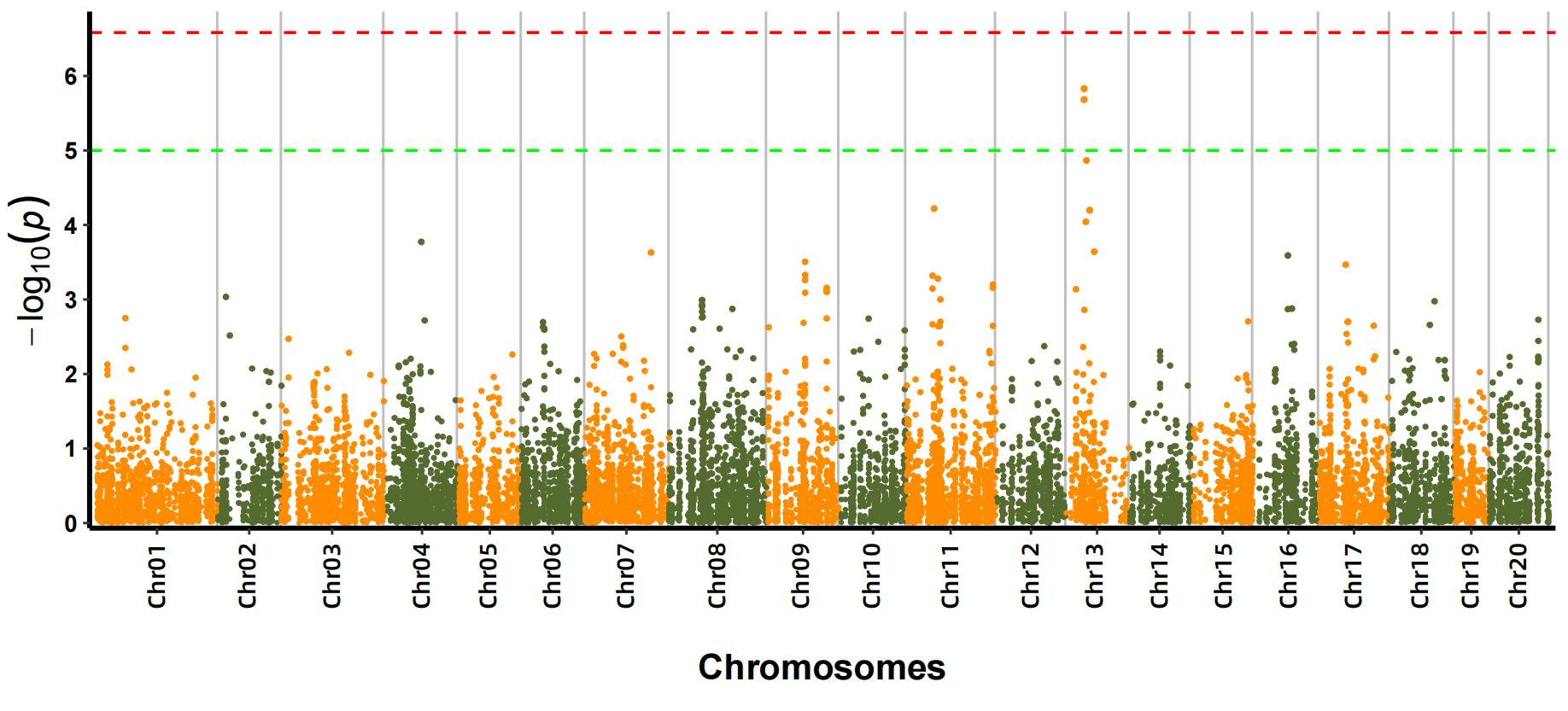
b.Assosiasjonsanalyse (modell: GEMMA, FaST-LMM, EMMAX)
QQ Plot
Manhattan Plot
| År | Tidsskrift | IF | Tittel |
| 2022 | NC | 17,69 | Genomisk grunnlag for giga-kromosomene og giga-genomet til trepionen Paeonia ostii |
| 2015 | NP | 7,43 | Domestiseringsfotavtrykk forankrer genomiske regioner av agronomisk betydning i soyabønner |
| 2018 | MP | 9.32 | Helgenom-resekvensering av en verdensomspennende samling av rapsfrø avslører genetisk grunnlag for deres økotype-divergens |
| 2022 | HR | 7,29 | Genomomfattende assosiasjonsanalyse gir molekylær innsikt i den naturlige variasjonen av vannmelonfrøstørrelse |