Human Whole Exome Sequencing
Tjenestefordeler
● Målrettet proteinkodende region: ved å fange og sekvensere proteinkodende region, brukes hWES til å avsløre varianter relatert til proteinstruktur;
● Høy nøyaktighet: med høy sekvenseringsdybde letter hWES deteksjon av vanlige varianter og sjeldne varianter med frekvenser lavere enn 1 %;
● Kostnadseffektivt: hWES gir omtrent 85 % av humane sykdomsmutasjoner fra 1 % av det menneskelige genomet;
● Fem strenge QC-prosedyrer som dekker hele prosessen med Q30>85 % garantert.
Eksempel spesifikasjoner
| Plattform
| Bibliotek
| Exon-fangststrategi
| Anbefal sekvenseringsstrategi
|
|
Illumina NovaSeq-plattformen
| PE150 | Agilent SureSelect Human All Exon V6 IDT xGen Exome Hyb Panel V2 | 5 Gb 10 Gb |
Eksempelkrav
| Eksempeltype
| Beløp(Qubit®)
| Volum
| Konsentrasjon
| Renhet (NanoDrop™) |
|
Genomisk DNA
| ≥ 300 ng | ≥ 15 μL | ≥ 20 ng/μL | OD260/280=1,8-2,0 ingen nedbrytning, ingen forurensning
|
Anbefalt sekvenseringsdybde
For Mendelske lidelser/sjeldne sykdommer: effektiv sekvenseringsdybde over 50×
For tumorprøver: effektiv sekvenseringsdybde over 100×
Bioinformatikk


Servicearbeidsflyt

Prøvelevering

DNA-ekstraksjon

Bygging av bibliotek

Sekvensering

Dataanalyse

Datalevering

Ettersalgstjenester
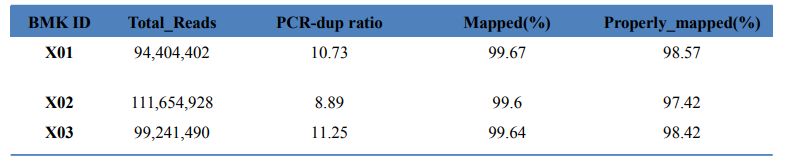
1. Justeringsstatistikk
Tabell 1 Statistikk over kartresultat
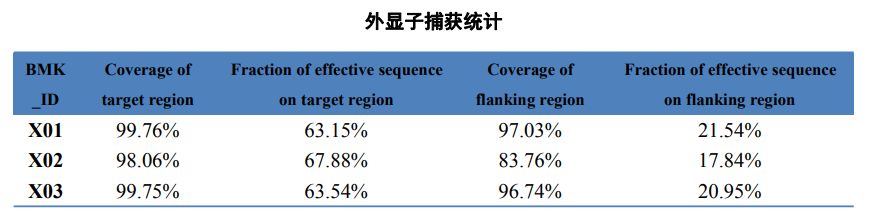
Tabell 2 Statistikk over eksomfangst
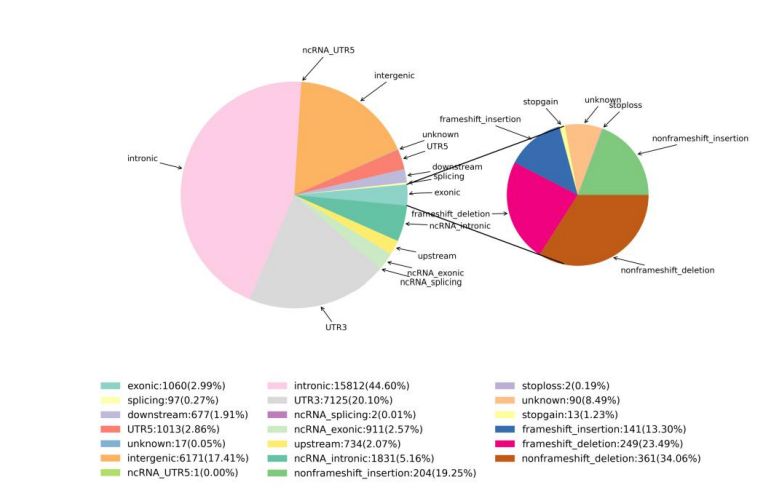
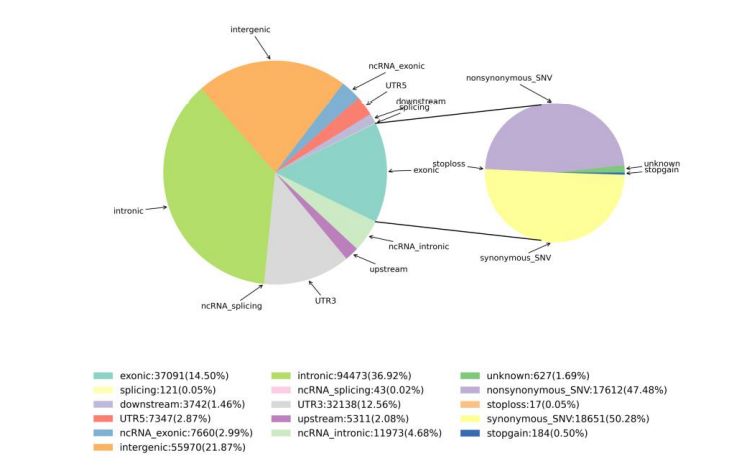
2. Variasjonsdeteksjon
Figur 1 Statistikk for SNV og InDel
3.Avansert analyse
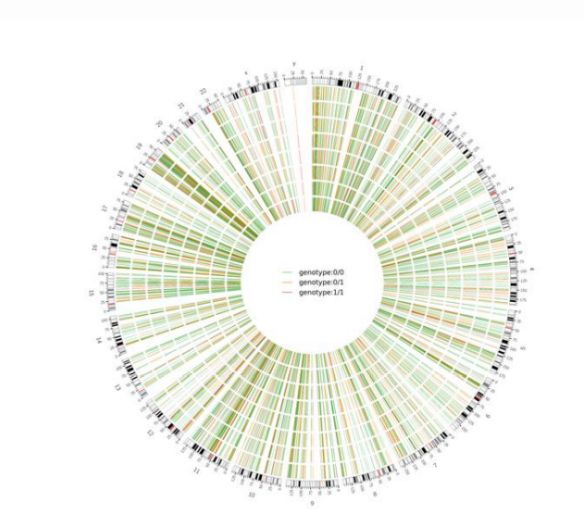
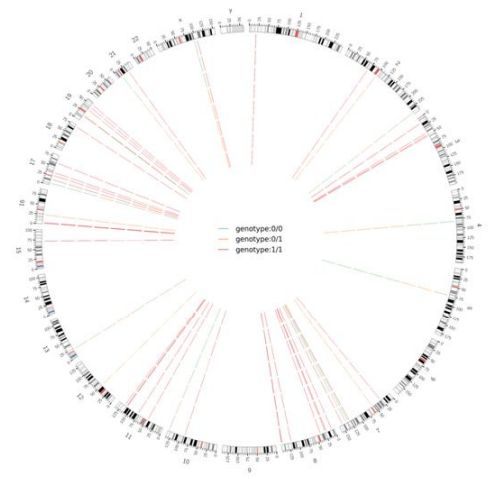
Figur 2 Circos-plott av genomomfattende skadelig SNV og InDel
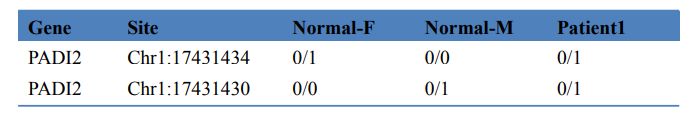
Tabell 3 Screening av sykdomsfremkallende gener