Métagénomique (NGS)
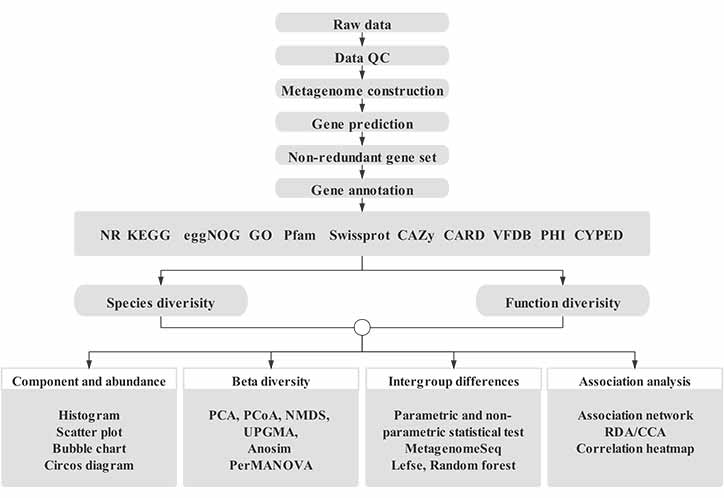
Cette plate-forme d'analyse est conçue pour l'analyse de données métagénomiques sur la base d'années d'expérience.Il consiste en un flux de travail intégré contenant diverses analyses métagénomiques couramment nécessaires, notamment le traitement des données, les études au niveau des espèces, les études au niveau des fonctions des gènes, le regroupement des métagénomes, etc. De plus, des outils d'exploration de données personnalisés sont disponibles sur le flux de travail d'analyse standard, y compris les requêtes sur les gènes et les espèces. , paramétrage, génération de figures personnalisées, etc.
Identification du relevé
Flux de travail en bioinformatique