Metagenomics (NGS)
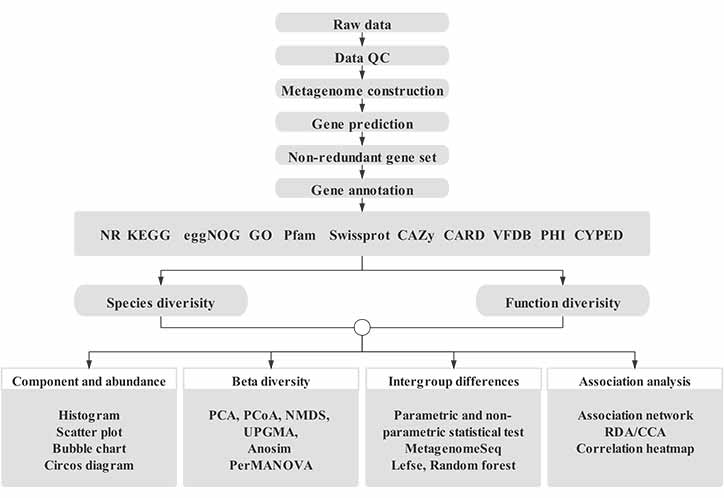
Denna analysplattform är designad för metagenomisk dataanalys med hagelgevär baserat på många års erfarenhet.Det består av ett integrerat arbetsflöde som innehåller olika vanliga metagenomiska analyser, inklusive databehandling, studier på artnivå, studier på genfunktionsnivå, metagenombinning, etc. Dessutom finns anpassade datautvinningsverktyg tillgängliga på standardanalysarbetsflöden, inklusive gen- och artförfrågan , parameterinställning, personlig figurgenerering, etc.
Transcipidentifiering
Arbetsflöde för bioinformatik