Metagenomika (NGS)
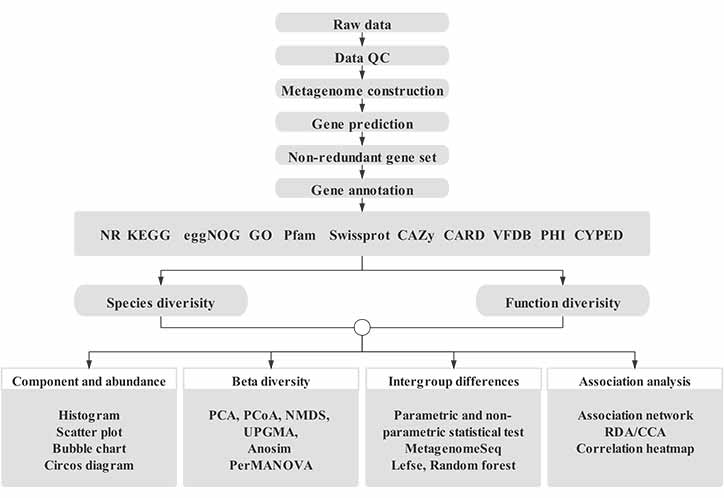
Hierdie ontledingsplatform is ontwerp vir haelgeweer metagenomiese data-analise op grond van jare se ondervinding.Dit bestaan uit geïntegreerde werkvloei wat verskeie algemeen benodigde metagenomika-ontledings bevat, insluitend dataverwerking, spesievlakstudies, geenfunksievlakstudies, metagenoombinning, ens. Daarbenewens is pasgemaakte data-ontginningsnutsmiddels beskikbaar op standaardanalise-werkvloei, insluitend geen- en spesienavraag , parameterinstelling, gepersonaliseerde figuurgenerering, ens.
Transsipeer identifikasie
Bioinformatika-werkvloei