NGS-مرنا (مرجع)
النسخ هو الرابط بين المعلومات الوراثية الجينومية وبروتين الوظيفة البيولوجية.يعد تنظيم المستوى النسخي هو أسلوب التنظيم الأكثر أهمية والأكثر دراسة على نطاق واسع للكائنات الحية.يمكن لتسلسل النسخ أن يقوم بتسلسل النسخة في أي وقت أو تحت أي ظرف، مع دقة دقيقة لنيوكليوتيد واحد. ويمكن أن يعكس ديناميكيًا مستوى نسخ الجينات، وتحديد وقياس النصوص النادرة والعادية في نفس الوقت، وتوفير المعلومات الهيكلية للنسخ الجيني. عينة النصوص المحددة.
في الوقت الحاضر، تُستخدم تكنولوجيا تسلسل النسخ على نطاق واسع في الهندسة الزراعية والطب ومجالات البحث الأخرى، بما في ذلك تنظيم تنمية الحيوانات والنباتات، والتكيف البيئي، والتفاعل المناعي، وتوطين الجينات، والتطور الوراثي للأنواع، والكشف عن الأورام والأمراض الوراثية.
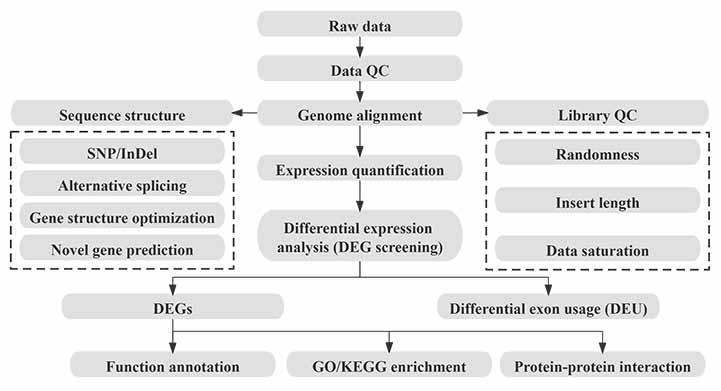
المعلوماتية الحيوية
تدفق عمل المعلوماتية الحيوية