NGS-mRNS (referencia)
A transzkriptom a kapcsolat a genomi genetikai információ és a biológiai funkció proteomja között.A transzkripciós szintű szabályozás az élőlények legfontosabb és legszélesebb körben vizsgált szabályozási módja.A transzkriptom szekvenálás bármely időpontban vagy bármilyen körülmények között meg tudja szekvenálni a transzkriptumot egyetlen nukleotid pontos felbontással. Dinamikusan tükrözheti a géntranszkripció szintjét, egyidejűleg azonosíthatja és mennyiségileg meghatározhatja a ritka és normál transzkriptumokat, és szerkezeti információkat szolgáltathat minta konkrét átirat.
Jelenleg a transzkriptom szekvenálási technológiát széles körben alkalmazzák az agronómiában, az orvostudományban és más kutatási területeken, beleértve az állatok és növények fejlődésének szabályozását, a környezeti alkalmazkodást, az immunkölcsönhatást, a gén lokalizációját, a fajok genetikai evolúcióját, valamint a daganatok és genetikai betegségek kimutatását.
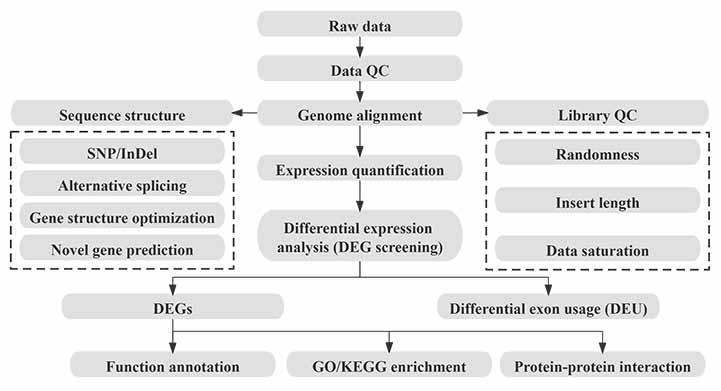
Bioinformatika
Bioinformatikai munkafolyamat