NGS-ARNm (referència)
El transcriptoma és l'enllaç entre la informació genètica genòmica i el proteoma de la funció biològica.La regulació del nivell transcripcional és el mode de regulació més important i més àmpliament estudiat dels organismes.La seqüenciació del transcriptoma pot seqüenciar el transcriptoma en qualsevol moment o en qualsevol condició, amb una resolució precisa d'un únic nucleòtid. Pot reflectir dinàmicament el nivell de transcripció gènica, identificar i quantificar simultàniament transcripcions rares i normals i proporcionar la informació estructural de transcripcions específiques de mostra.
Actualment, la tecnologia de seqüenciació del transcriptoma s'ha utilitzat àmpliament en agronomia, medicina i altres camps d'investigació, com ara la regulació del desenvolupament animal i vegetal, l'adaptació ambiental, la interacció immune, la localització de gens, l'evolució genètica de les espècies i la detecció de tumors i malalties genètiques.
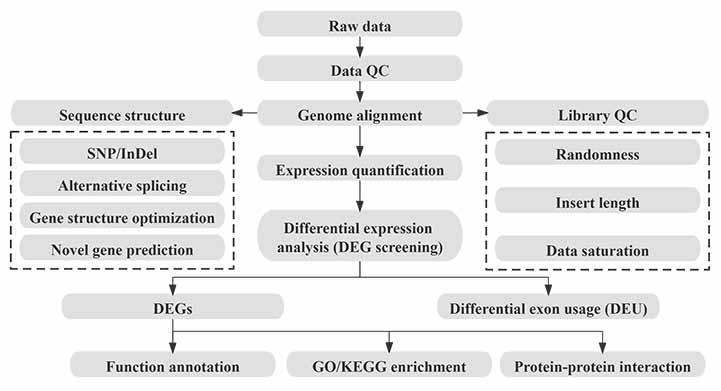
Bioinformàtica
Flux de treball en bioinformàtica