NGS-mRNA (справка)
Транскриптомът е връзката между геномната генетична информация и протеома на биологичната функция.Регулирането на нивото на транскрипция е най-важният и най-широко изучаваният начин на регулиране на организмите.Секвенирането на транскриптома може да секвенира транскриптома по всяко време или при всякакви условия, с разделителна способност, точна до един нуклеотид. То може динамично да отразява нивото на генна транскрипция, едновременно да идентифицира и количествено определя редки и нормални транскрипти и да предоставя структурна информация за примерни специфични преписи.
Понастоящем технологията за секвениране на транскриптоми е широко използвана в агрономията, медицината и други изследователски области, включително регулиране на развитието на животни и растения, адаптация към околната среда, имунно взаимодействие, локализация на гени, генетична еволюция на видове и откриване на тумори и генетични заболявания.
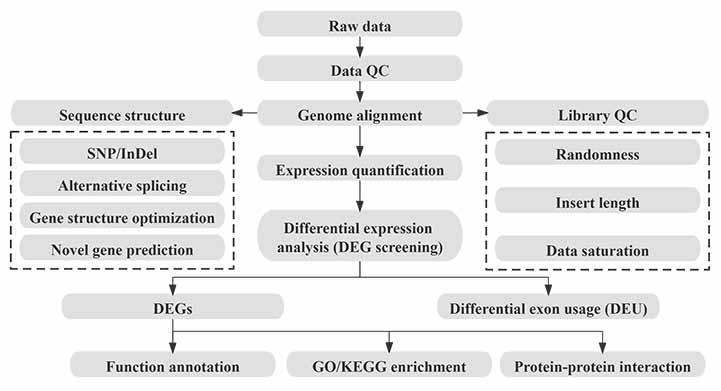
Биоинформатика
Работен поток по биоинформатика