NGS-mRNA(ອ້າງອີງ)
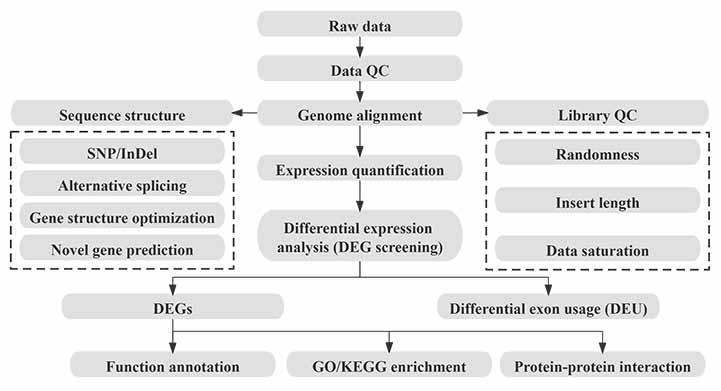
Transcriptome ແມ່ນການເຊື່ອມໂຍງລະຫວ່າງຂໍ້ມູນທາງພັນທຸກໍາຂອງ genomic ແລະ proteome ຂອງການເຮັດວຽກທາງຊີວະພາບ.ກົດລະບຽບລະດັບການຖອດຂໍ້ຄວາມແມ່ນສໍາຄັນທີ່ສຸດແລະເປັນວິທີການສຶກສາຢ່າງກວ້າງຂວາງທີ່ສຸດຂອງອົງການຈັດຕັ້ງ.ການຈັດລໍາດັບການຖອດຂໍ້ຄວາມສາມາດຈັດລໍາດັບ transcriptome ໃນຈຸດໃດຫນຶ່ງໃນເວລາຫຼືພາຍໃຕ້ເງື່ອນໄຂໃດກໍ່ຕາມ, ດ້ວຍຄວາມລະອຽດທີ່ຖືກຕ້ອງກັບ nucleotide ດຽວ. ມັນສາມາດສະທ້ອນເຖິງລະດັບຂອງ transcription gene, ພ້ອມກັນກໍານົດແລະປະລິມານການຖອດຂໍ້ຄວາມທີ່ຫາຍາກແລະປົກກະຕິ, ແລະໃຫ້ຂໍ້ມູນໂຄງສ້າງຂອງ. ຕົວຢ່າງການຖອດຂໍ້ຄວາມສະເພາະ.
ໃນປັດຈຸບັນ, ເທກໂນໂລຍີການຈັດລໍາດັບ transcriptome ໄດ້ຖືກນໍາໃຊ້ຢ່າງກວ້າງຂວາງໃນການປູກຝັງ, ຢາປົວພະຍາດແລະຂົງເຂດການຄົ້ນຄວ້າອື່ນໆ, ລວມທັງກົດລະບຽບການພັດທະນາສັດແລະພືດ, ການປັບຕົວຂອງສິ່ງແວດລ້ອມ, ປະຕິສໍາພັນຂອງພູມຕ້ານທານ, ທ້ອງຖິ່ນຂອງພັນທຸກໍາ, ການວິວັດທະນາທາງພັນທຸກໍາຂອງຊະນິດພັນແລະການກວດພົບໂຣກ tumor ແລະພັນທຸກໍາ.
ຊີວະວິທະຍາ
ກະແສການເຮັດວຽກຂອງ Bioinformatics