
Tam transkriptom dizilimi - Illumina
Hizmet Avantajları
● Sayımlar, ekspresyon ve kromozom bazlı göreceli ekspresyon açısından tüm RNA türlerine ilişkin tahmin
● Diferansiyel olarak ifade edilen RNA'ların ve karşılık gelen ifadenin tanımlanması
● Gen ortak ifade analizi
● ceRNA ağ analizi
● Yol analizini içeren anahtar genler
● BMKCloud tabanlı sonuç sunumu: Platformda özelleştirilmiş veri madenciliği mevcuttur
● Satış sonrası hizmetler projenin tamamlanmasından sonra 3 ay süreyle geçerlidir
Numune Gereksinimleri ve Teslimat
| Kütüphane | platformu | Önerilen veriler | Veri Kalite Kontrolü |
| Toplam RNA | Illumina PE150&SE50 | Circ/lnc/mRNA: 16G;miRNA: 10 milyon okuma | Q30≥85% |
Örnek Gereksinimler:
Nükleotidler:
| Saflık | Bütünlük | Bulaşma | Miktar |
| OD260/280≥1,7-2,5; OD260/230≥0,5-2,5; | Bitkiler için: RIN≥6,5;Hayvanlar için: RIN≥7;28S/18S≥1,0;sınırlı veya taban çizgisi yüksekliği yok | Jel üzerinde sınırlı miktarda protein veya DNA kontaminasyonu gösteriliyor veya hiç gösterilmiyor. | Kons.≥100 ng/μl;Hacim ≥ 10 ul;Toplam ≥ 2 μg |
Doku: Ağırlık(kuru): ≥1 g
*5 mg'dan küçük dokular için flaş dondurulmuş (sıvı nitrojen içinde) doku örneğinin gönderilmesini öneririz.
Hücre süspansiyonu: Hücre sayısı = 3×107
*Dondurulmuş hücre lizatını göndermenizi öneririz.Hücre sayısının 5×105'ten küçük olması durumunda sıvı nitrojende flaş dondurma yapılması önerilir.
Kan örnekleri:
PA×genKanRNATube;
6 mLLTRIzol ve 2mL kan(TRIzol:Kan=3:1)
Önerilen Numune Teslimatı
Konteyner:
2 ml santrifüj tüpü (Kalay folyo tavsiye edilmez)
Örnek etiketleme: Grup+kopya örneğin A1, A2, A3;B1, B2, B3... ...
Gönderi:
1.Kuru buz: Numunelerin torbalara konulması ve kuru buza gömülmesi gerekir.
2.RNAstabil tüpler: RNA örnekleri, RNA stabilizasyon tüpünde (örn. RNAstable®) kurutulabilir ve oda sıcaklığında gönderilebilir.
Hizmet İş Akışı

Deney tasarımı

Numune teslimatı

RNA ekstraksiyonu

Kütüphane inşaatı

Sıralama

Veri analizi

Satış sonrası hizmetler
Biyoenformatik
1. Her türlü RNA'ya dayalı göreceli ifadeye ilişkin tahmin
RNA ifadesindeki farklılıkların önemi üzerine sirkler
2.Entegre KEGG yolu ağı
Entegre KEGG yolu ağı
3.ceRNA ağ analizi
ceRNA Ağı tabanlı DE-circRNA-miRNA-mRNA Etkileşimleri
BMK Davası
Brassica campestris'in CMS Hattında Anter Gelişiminde Yer Alan CircRNA İfade Modeli ve ceRNA ve miRNA – mRNA Ağları
Yayınlanan:Uluslararası Moleküler Bilimler Dergisi,2019
NONMMUT015812-yıkma KP (shRNA-2) hücreleri ve negatif kontrol (sh-Scr) hücreleri, spesifik bir viral enfeksiyonun 6. gününde elde edildi.
Temel sonuçlar
Bu çalışma, Brassica campestris L. ssp.'de Polima sitoplazma erkek kısırlığı (CMS) hattı “Bcpol97-05A” ve verimli hat olan “Bcajh97-01B”yi tespit etmiştir.chinensis Makino, syn.B. rapa ssp.chinensis ve tam transkriptom dizilimi ile steril hattın çiçek tomurcukları ile verimli hat arasında RNA ekspresyon profili karşılaştırmaları gerçekleştirdi.
1. Toplam 31 diferansiyel olarak eksprese edilmiş (DE) circRNA, 47 DE miRNA ve 4779 DE mRNA tanımlandı.
2. Gen ontolojisi (GO) analizi, DE genlerinin çoğunun polen duvarı gelişiminde rol oynadığını gösterdi
3.DE mRNA'larının KEGG yolu zenginleştirme analizi (fertil soy ile karşılaştırıldığında steril soydaki yukarı regüle edilmiş ve aşağı regüle edilmiş mRNA'lar dahil) "nişasta ve sükroz metabolizması", "fenilpropanoid biyosentezi" ve "pentoz ve glukuronat karşılıklı dönüşümleri" gösterdi. ”en zengin metabolik yollardı.
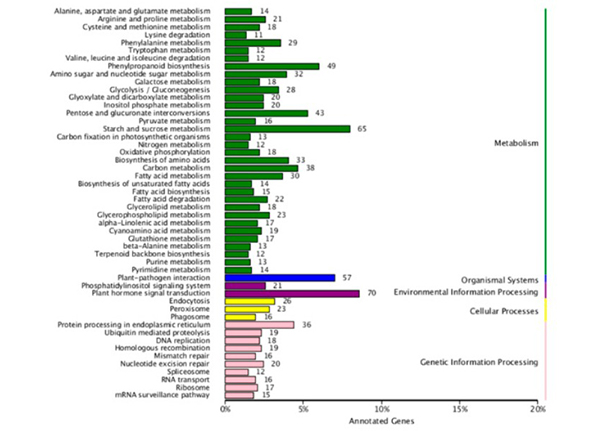 Diferansiyel olarak ifade edilen mRNA'ların KEGG yolu zenginleştirme analizi | 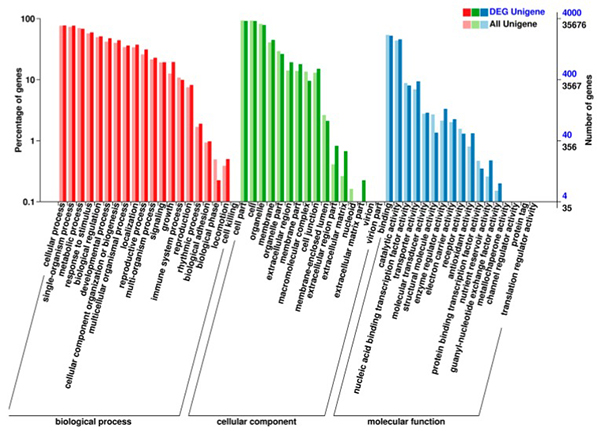 Diferansiyel olarak ifade edilen mRNA'ların gen ontolojisi (GO) sınıflandırması | 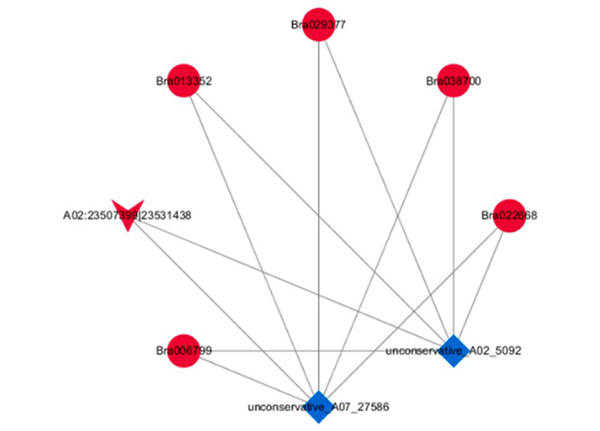 DEcircRNA – DEmiRNA – DEmRNA üçlü ağının görünümü |
Referans
Liang Y, Zhang Y, Xu L ve diğerleri.Brassica campestris'in[J] CMS Hattında Anter Gelişiminde Yer Alan CircRNA İfade Modeli ve ceRNA ve miRNA-mRNA Ağları.Uluslararası Moleküler Bilimler Dergisi, 2019, 20(19):4808-.DOI: 10.3390/ijms20194808














