Sammenlignende genomik
Service fordele
● Omfattende analysepakke, der indeholder de otte mest almindeligt nødvendige analyser
● Høj pålidelighed i analyse med detaljeret og let forståelig fortolkning af resultater
● Veldesignede udgivelsesklare figurer
● Højt kvalificeret bioinformatik-team opfylder forskellige personaliserede analysekrav
● Kortere gennemløbstid med højere nøjagtighed i analysen
● Rigelig erfaring med over 90 vellykkede sager med en akkumuleret publiceret effektfaktor på over 900
Service specifikationer
| Estimeret ekspeditionstid | Antal arter | Analyser |
| 30 arbejdsdage | 6 - 12 | Genfamilieklynger Genfamilieudvidelse og sammentrækning Fylogenetisk trækonstruktion Estimering af divergenstid (fossil kalibrering påkrævet) LTR indsættelsestid (for planter) Helgenomduplikation (for planter) Selektivt tryk Synteny analyse |
Bioinformatiske analyser
● Genfamilie
● Fylogenetik
● Divergens tid
● Selektivt tryk
● Synteny-analyse
Prøvekrav og levering
Prøvekrav:
Væv eller DNA til genomsekventering og samling
Til væv
| Arter | Væv | Undersøgelse | PacBio CCS |
| Dyr | Visceralt væv | 0,5 ~ 1 g | ≥ 3,5 g |
| Muskelvæv | |||
| ≥ 5,0 g | |||
| ≥ 5,0 ml | |||
| Pattedyrs blod | |||
| ≥ 0,5 ml | |||
| Fjerkræ/Fiskeblod | |||
| Plante | Friske blade | 1 ~ 2 g | ≥ 5,0 g |
| Kronblad/Stængel | 1 ~ 2 g | ≥ 10,0 g | |
| Rod/frø | 1 ~ 2 g | ≥ 20,0 g | |
| Celler | dyrket celle | - | ≥ 1 x 108 |
Data
Genomsekvensfiler (.fasta) og annotationsfiler (.gff3) af nært beslægtede arter
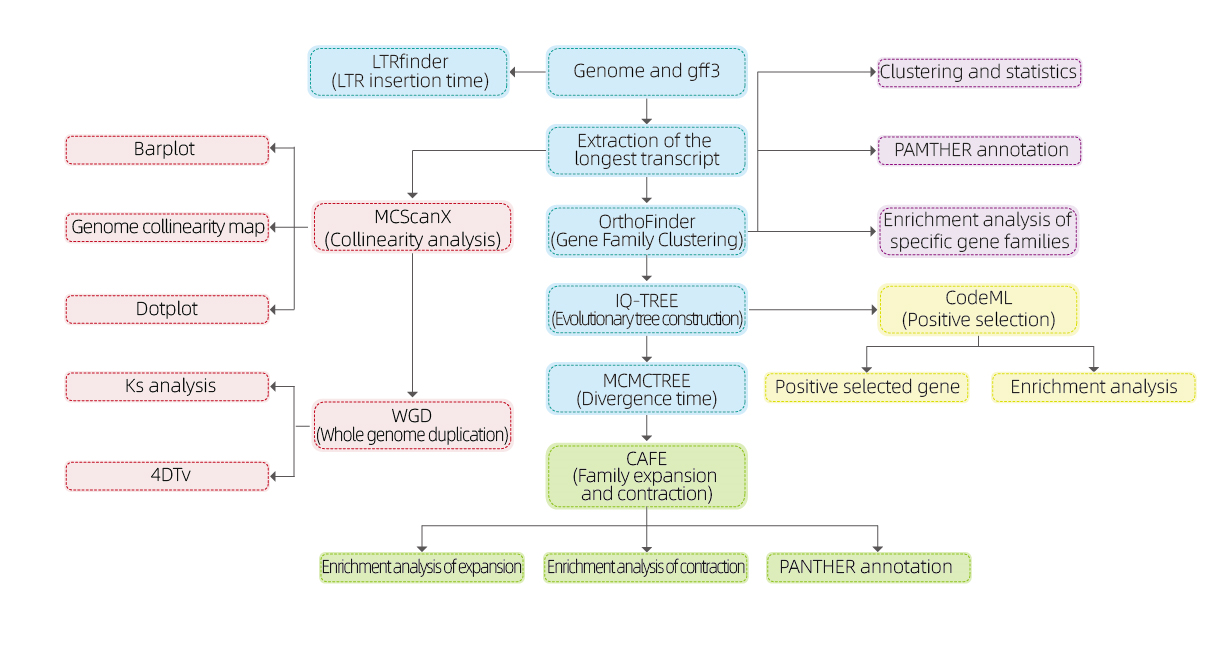
Service Work Flow

Eksperimentdesign

Prøve levering

Biblioteksbyggeri

Sekvensering

Dataanalyse

Eftersalgsservice
*Demoresultater vist her er alle fra genomer offentliggjort med Biomarker Technologies
1.LTR inserttidsestimering: Figuren viste en unik bimodal fordeling i LTR-RTs indsættelsestider i Weining ruggenomet sammenlignet med andre arter.Den seneste top dukkede op for omkring 0,5 millioner år siden.
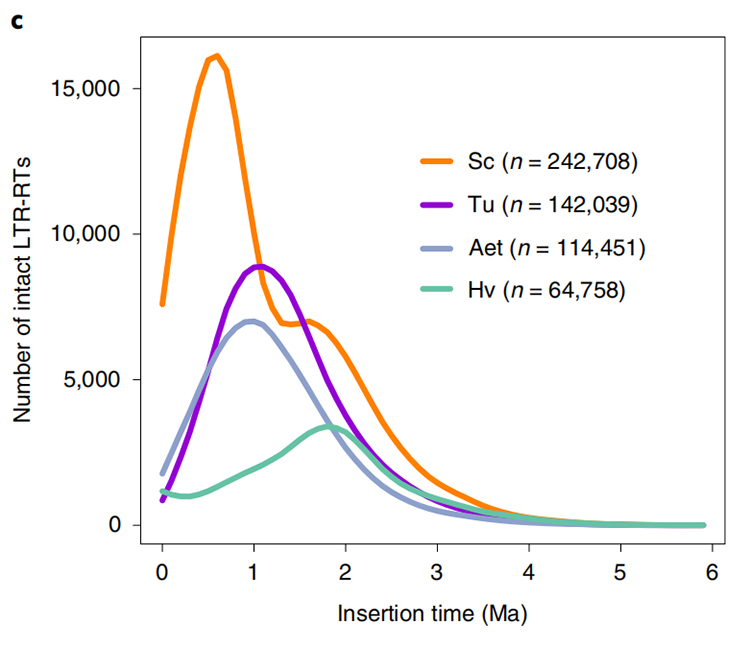
Li Guang et al.,Naturgenetik, 2021
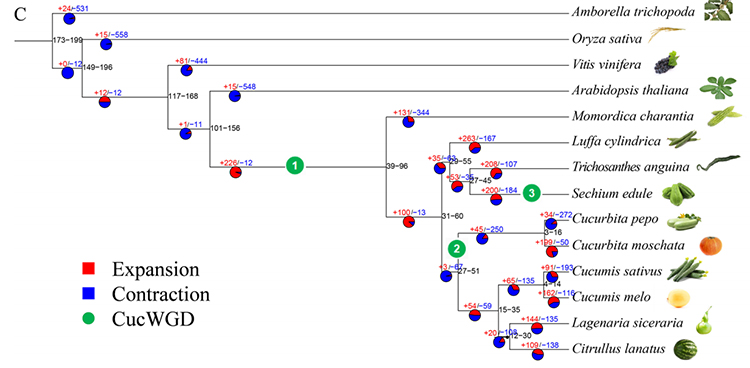
2. Fylogeni og genfamilieanalyse på chayote (Sechium edule): Ved at analysere chayote og de andre 13 beslægtede arter i genfamilien viste det sig, at Chayote var tættest beslægtet med slangegræskar (Trichosanthes anguina).Chayote afledt af slange-græskar i omkring 27-45 Mya og helgenomduplikation (WGD) blev observeret i chayote i 25±4 Mya, hvilket er den tredje WGD-hændelse i cucuibitaceae.
Fu A et al.,Havebrugsforskning, 2021
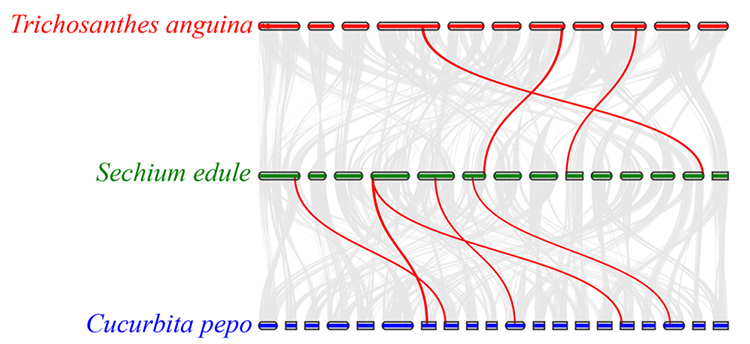
3. Synteny-analyse: Nogle gener relateret til phytohormoner i frugtudvikling blev fundet i chayote, slange-græskar og squash.Korrelationen mellem chayote og squash er lidt højere end den mellem chayote og slange-græskar.
Fu A et al.,Havebrugsforskning, 2021
4.Genfamilieanalyse: KEGG-berigelse på genfamilieudvidelse og sammentrækning i G.thurberi- og G.davidsonii-genomer viste, at steroidbiosyntese og brassinosteroidbiosynteserelaterede gener blev udvidet.
Yang Z et al.,BMC Biologi, 2021
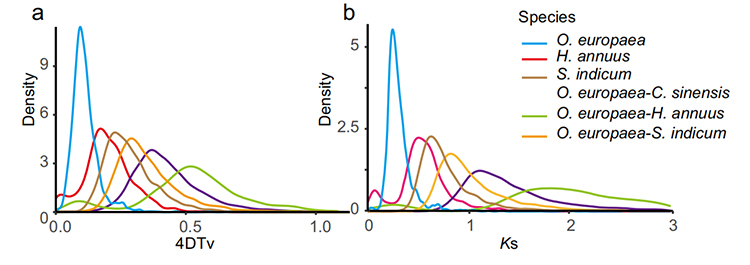
5. Helgenomduplikationsanalyse: 4DTV og Ks distributionsanalyse viste hele genomduplikationshændelse.Toppe af intraarts viste duplikationsbegivenheder.Toppe af interspecies vist artsdannelsesbegivenheder.Analysen indikerede, at sammenlignet med de tre andre nært beslægtede arter, gik O. europaea igennem en genduplikation i stor skala for nylig.

Rao G et al.,Havebrugsforskning, 2021
BMK sag
Rose uden prikle: genomisk indsigt forbundet med fugttilpasning
Udgivet: National Science Review, 2021
Sekvenseringsstrategi:
'BasyesTornfri' (R.Wichurainan) genom:
Ca.93 X PacBio + ca.90 X Nanopore + 267 X Illumina
Nøgleresultater
1. R.wichuraiana-genomet af høj kvalitet blev konstrueret ved hjælp af langlæste sekventeringsteknikker, som giver en samling på 530,07 Mb (estimeret genomstørrelse var ca. 525,9 Mb ved flowcytometri og 525,5 ved genomundersøgelse; Heterozygositet var omkring 1,03%).BUSCO estimerede score var 93,9%.Sammenligning med "Old blush" (haploOB) blev kvaliteten og fuldstændigheden af dette genom bekræftet af base-enkeltbase-nøjagtighed og LTR-samlingsindeks (LAI=20,03).R.wichuriana-genomet indeholder 32.674 proteinkodende gener.
2.Multi-omics fælles analyse, bestående af komparativ genomik, transkriptomics, QTL-analyse af genetisk population, afslørede den afgørende artsdannelse mellem R. wichuraiana og Rosa chinensis.Også ekspressionsvariation af beslægtede gener i QTL var sandsynligvis forbundet med stamprickle-mønster.

Komparativ genomisk analyse mellem Basye's Thornless og Rosa chinensis, herunder synteny-analyse, genfamilieklynge, ekspansions- og kontraktionsanalyse, afslørede et stort antal variationer, som var relateret til afgørende træk hos roser.Den unikke ekspansion i NAC- og FAR1/FRS-genfamilien var meget sandsynligt forbundet med resistens mod sort plet.
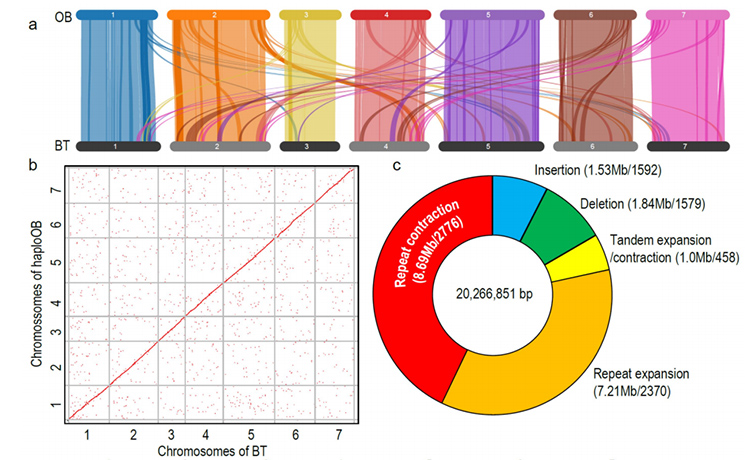

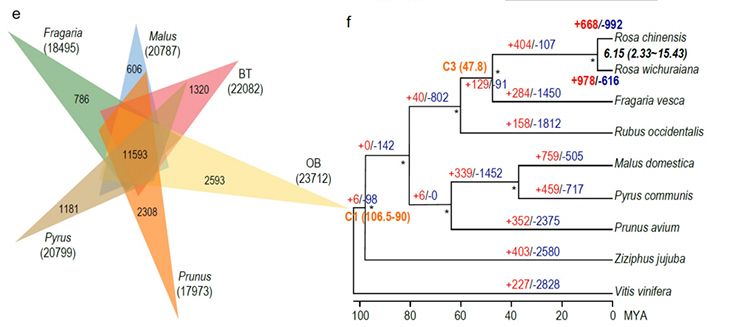
Komparativ genomisk analyse mellem BT- og haploOB-genomer.
Zhong, M., et al."Rose uden prikle: genomisk indsigt forbundet med fugttilpasning"National Science Review, 2021;, nwab092.