DNA/RNA-Sequenzierung – Nanoporen-Sequenzer
Servicedetails-Funktionen
| Plattform | Bibliotheksgröße | Theoretischer Datenertrag (pro Zelle) | Einzelbasis-Genauigkeit | Anwendungen |
| Nanopore | 8 Kb, 10 kb, 20 kb, Ultralong, cDNA-PCR | 70–90 GB/Zelle | 85-92 % | SV-Calling, De-novo, Sequenzierung in voller Länge, Iso-Seq, Genannotation, Nachweis der DNA-Methylierung |
Servicevorteile
● Über 5 Jahre Erfahrung mit der PacBio-Sequenzierungsplattform mit Tausenden abgeschlossenen Projekten mit verschiedenen Arten.
● BMKGENE ist offizieller Partner von Oxford Nanopore mit Dual-Plattform-RNA/DNA-Zertifizierung.
● Es gibt Mainstream-Modelle von Sequenzern mit kompletter Ausstattung und ausreichendem Sequenzierungsdurchsatz.
● Basierend auf der Nanopore-Plattform wurden mehr als 10 Tier- und Pflanzenforschungen von Denovo in international renommierten Fachzeitschriften veröffentlicht.
Beispielanforderungen
| Beispielstyp | Menge | Konzentration (Qubit ®) | Volumen | Reinheit | Andere |
| Genomische DNA | Abhängig von den Datenanforderungen | ≥20ng/μl | ≥15μl | OD260/280=1,7-2,2; OD260/230≥1,5; Klarer Peak bei 260 nm, keine Verunreinigungen | Die Konzentration muss mit Qubit und Qubit/Nanopore ≤ 2 gemessen werden |
| Gesamt-RNA | ≥1,2μg | ≥100μg/μl | ≥15μl | OD260/280=1,7-2,5; OD260/230=0,5-2,5;keine Verunreinigungen | RIN-Wert ≥7,5 |
Service-Workflow
Probenvorbereitung

Bibliotheksbau

Sequenzierung

Datenanalyse

Projektabwicklung
Datenqualitätsbewertung von DNA-Proben
Tabelle 1. Statistiken zu sauberen Daten.
| BMKID | rawSeqNum | rawSumBase | cleanSeqNum | cleanSumBase | cleanN50Len | cleanN90Len | cleanMeanLen | cleanMaxLen | cleanMeanQual |
| DNA_BMK01 | 1.218.239 | 26.37 | 1.121.736 | 25,90 | 28.014 | 15.764 | 23.090 | 143.181 | 9 |
Datenqualitätsbewertung der RNA-Probe
Tabelle 1. Statistiken zu sauberen Daten.
| Dateiname | Kunden ID | ReadNum | Basisnummer | N50 | Mittlere Länge | Maximale Länge | Mittlerer Qscore |
| RNA_BMK001 | C2 | 8.947.708 | 4.047.230.083 | 398 | 452 | 129.227 | F12 |
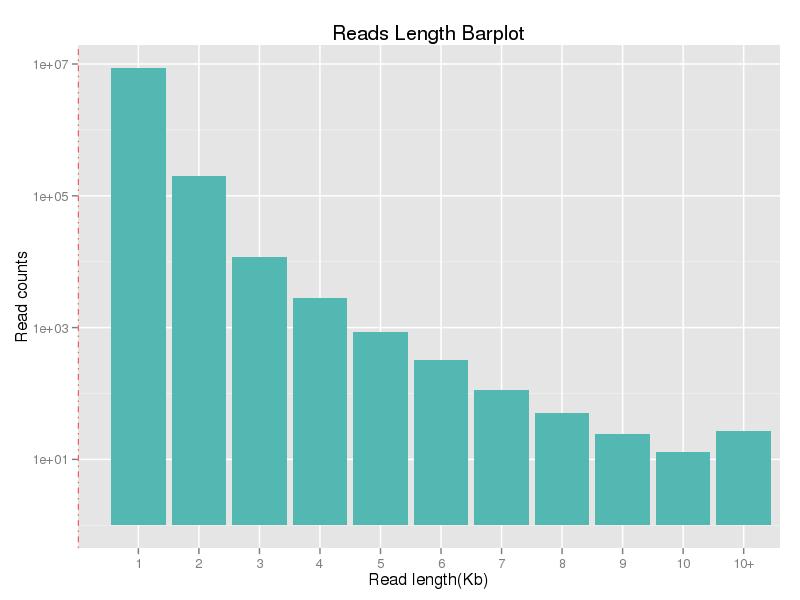
Abbildung 1. Leselängenverteilung
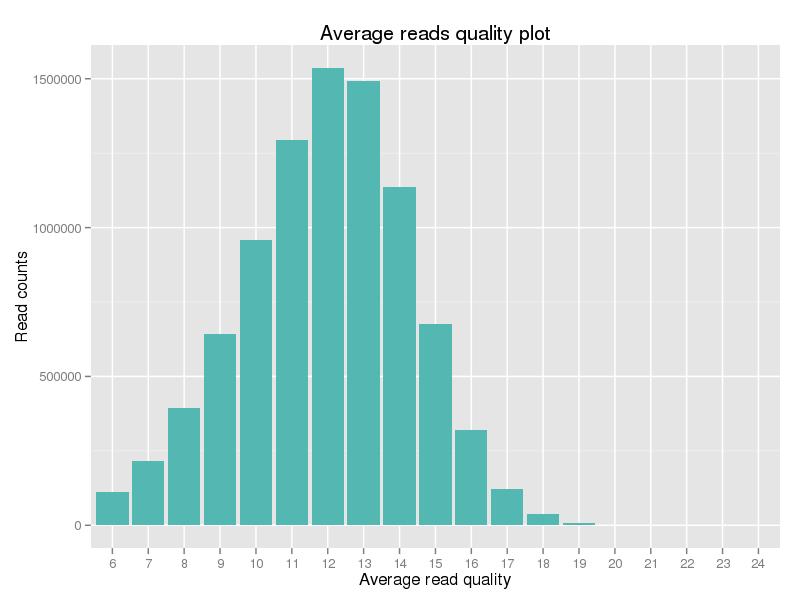
Abbildung 2. Qualitätsfaktorverteilung sauberer Daten
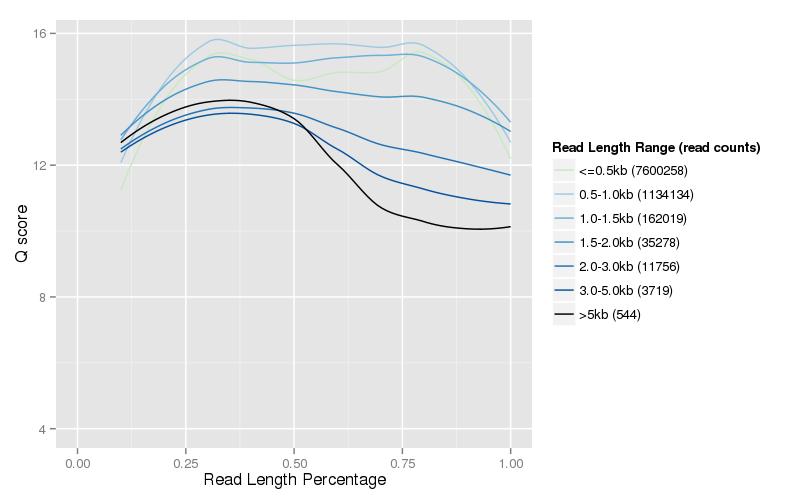
Abbildung 3. Längen- und Qualitätsbewertungsverteilung sauberer Daten