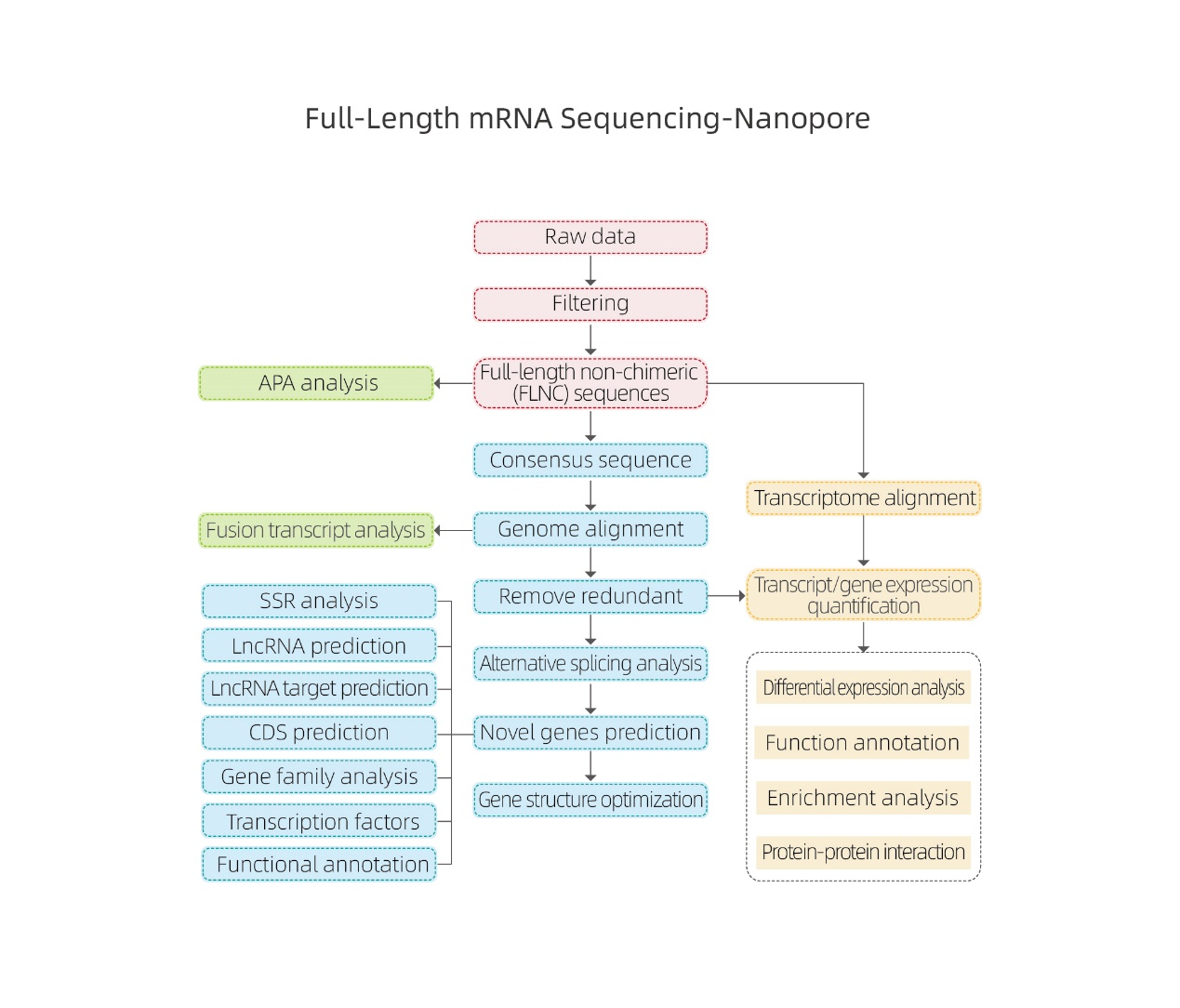
Sekuenca e mARN-së me gjatësi të plotë-Nanopore
Avantazhet e Shërbimit
● Paragjykim me sekuencë të ulët
● Zbulimi i molekulave të cDNA me gjatësi të plotë
● Kërkohen më pak të dhëna për të mbuluar të njëjtin numër transkriptesh
● Identifikimi i izoformave të shumta për gjen
● Kuantifikimi i shprehjes në nivel izoformë
Specifikimet e Shërbimit
| Librari | Platforma | Rendimenti i rekomanduar i të dhënave (Gb) | Kontrolli i cilësisë |
| cDNA-PCR (e pasuruar me Poly-A) | Nanopore PromethION P48 | 6 Gb/kampion (në varësi të specieve) | Raporti i gjatësisë së plotë (70% Nota mesatare e cilësisë: Q10
|
Analizat bioinformatike
●Përpunimi i të dhënave të papërpunuara
● Identifikimi i transkriptit
● Ndarja alternative
● Kuantifikimi i shprehjes në nivelin e gjeneve dhe nivelin e izoformës
● Analiza e shprehjes diferenciale
● Shënimi dhe pasurimi i funksionit (DEG dhe DET)
Kërkesat e mostrës dhe dorëzimi
Kërkesat e mostrës:
Nukleotidet:
| Konk.(ng/μl) | Sasia (μg) | Pastërti | Integriteti |
| ≥ 100 | ≥ 0.6 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Ndotje e kufizuar ose pa proteina ose ADN e treguar në xhel. | Për bimët: RIN≥7.0; Për kafshët: RIN≥7.5; 5.0≥28S/18S≥1.0; lartësi bazë e kufizuar ose aspak |
Indi: Pesha (e thatë): ≥1 g
*Për indet më të vogla se 5 mg, rekomandojmë dërgimin e mostrës së indit të ngrirë (në azot të lëngshëm).
Pezullimi i qelizave: Numri i qelizave = 3×106- 1×107
*Ne rekomandojmë dërgimin e lizatit të qelizave të ngrira.Në rast se ajo qelizë numërohet më e vogël se 5×105Rekomandohet i ngrirë në azot të lëngshëm, i cili preferohet për mikroekstraktim.
Mostrat e gjakut: Vëllimi≥1 ml
Dorëzimi i mostrës së rekomanduar
Enë: tub centrifuge 2 ml (Folja e kallajit nuk rekomandohet)
Etiketimi i mostrës: Grup+përsëritje p.sh. A1, A2, A3;B1, B2, B3 ... ...
Dërgesa: 2, Akull i thatë: Mostrat duhet të paketohen në thasë dhe të varrosen në akull të thatë.
- Tubat e qëndrueshëm të RNA: Mostrat e ARN-së mund të thahen në tubin e stabilizimit të ARN-së (p.sh. RNAstable®) dhe të dërgohen në temperaturën e dhomës.
Rrjedha e punës së shërbimit
Nukleotidet:

Dorëzimi i mostrës

Ndërtimi i bibliotekës

Sekuenca

Analiza e të dhënave

Shërbimet pas shitjes
Rrjedha e punës së shërbimit
Indi:

Dizajni i eksperimentit

Dorëzimi i mostrës

Ekstraktimi i ARN-së

Ndërtimi i bibliotekës

Sekuenca

Analiza e të dhënave

Shërbimet pas shitjes
1.Analiza e shprehjes diferenciale - Grafiku i vullkanit
Analiza e shprehjes diferenciale mund të përpunohet në të dy nivelet e gjeneve për të identifikuar gjenet e shprehura në mënyrë diferenciale (DEGs) dhe në nivelin izoformë për të identifikuar në mënyrë diferenciale
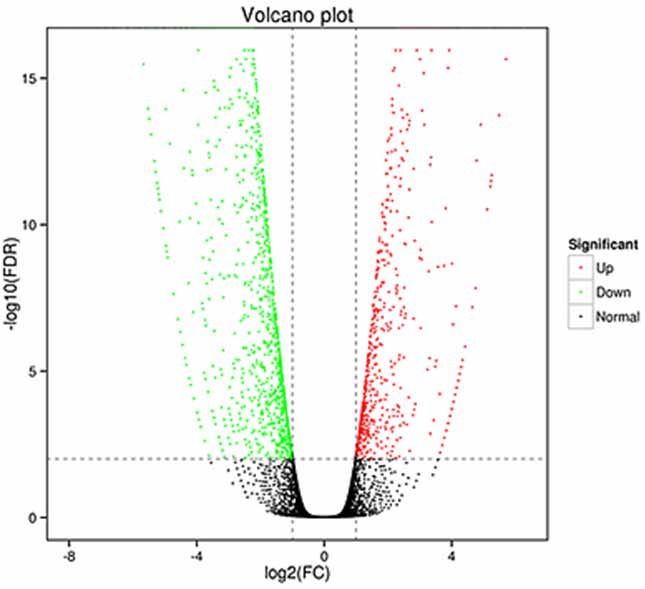
transkriptet e shprehura (DET)
2.Harta e nxehtësisë e grupimit hierarkik
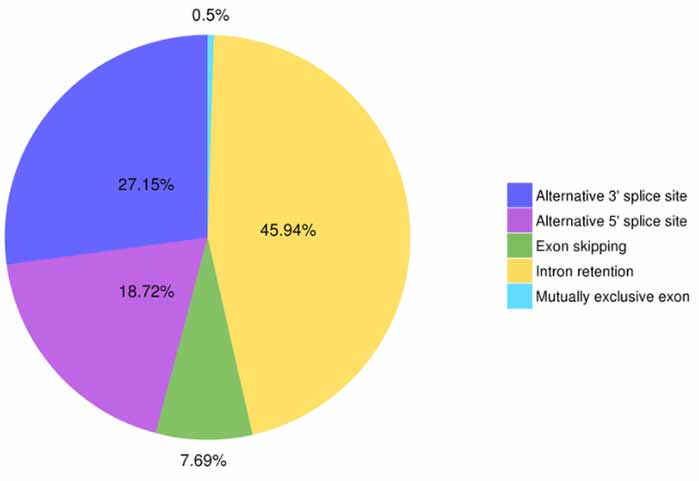
3.Identifikimi dhe klasifikimi alternativ i bashkimit
Pesë lloje të ngjarjeve alternative të bashkimit mund të parashikohen nga Astalavista.
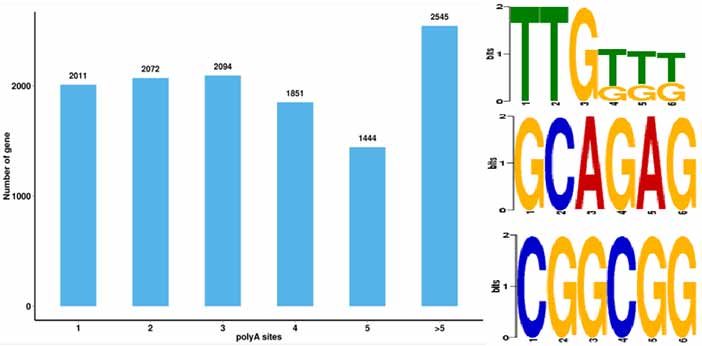
4.Identifikimi i ngjarjeve alternative të poliadenilimit (APA) dhe motivi në 50 bp në rrjedhën e sipërme të poli-A
Rasti BMK
Identifikimi alternativ i bashkimit dhe sasia e nivelit të izoformës me sekuencë të transkriptomeve me gjatësi të plotë nanopore
Publikuar:Nature Communications, 2020
Strategjia e renditjes:
Grupimi: 1. CLL-SF3B1(WT);2. CLL-SF3B1 (mutacion K700E);3. Qelizat B normale
Strategjia e renditjes: Sekuenca e bibliotekës MinION 2D, renditja e bibliotekës PromethION 1D;të dhëna të leximit të shkurtër nga të njëjtat mostra
Platforma e renditjes: Nanopore MinION;Nanopore PromethION;
Rezultatet kryesore
1.Identifikimi alternativ i bashkimit në nivel izoformë
Sekuencat e lexuara gjatë fuqizojnë identifikimin e mutantit SF3B1K700E-vendosjet e ndryshuara të bashkimit në nivelin e izoformës.35 alternativa 3'SS dhe 10 alternativa 5'SS u zbuluan se ishin të lidhura në mënyrë të konsiderueshme diferenciale midis SF3B1K700Edhe SF3B1WT.33 nga 35 ndryshimet u zbuluan rishtazi nga sekuenca të lexuara gjatë.
2. Kuantifikimi Alternativ i Splicing në nivel izoformë
Shprehja e izoformave të mbajtjes së intronit (IR) në SF3B1K700Edhe SF3B1WTu përcaktuan në sasi bazuar në sekuencat nanopore, duke zbuluar një rregullim të ulët global të izoformave IR në SF3B1K700E.
Referenca
Tang AD, Soulette CM, Baren MJV, etj.Karakterizimi i transkriptit të plotë të mutacionit SF3B1 në leuçeminë limfocitare kronike zbulon rregullimin e poshtëm të introneve të mbajtura [J].Nature Communications.