Нанапоры для секвенирования мРНК поўнай даўжыні
Перавагі абслугоўвання
● Нізкая зрушэнне паслядоўнасці
● Выяўленне поўнаметражных малекул кДНК
● Менш дадзеных, неабходных для ахопу аднолькавай колькасці стэнаграм
● Ідэнтыфікацыя некалькіх ізаформ на ген
● Колькасная ацэнка экспрэсіі на ўзроўні ізаформы
Спецыфікацыі абслугоўвання
| Бібліятэка | Платформа | Рэкамендаваны аб'ём дадзеных (Гб) | Кантроль якасці |
| кДНК-ПЦР (узбагачаны Poly-A) | Нанапор PromethION P48 | 6 Гб/узор (у залежнасці ад выгляду) | Суадносіны поўнай даўжыні >70% Сярэдні бал якасці: Q10
|
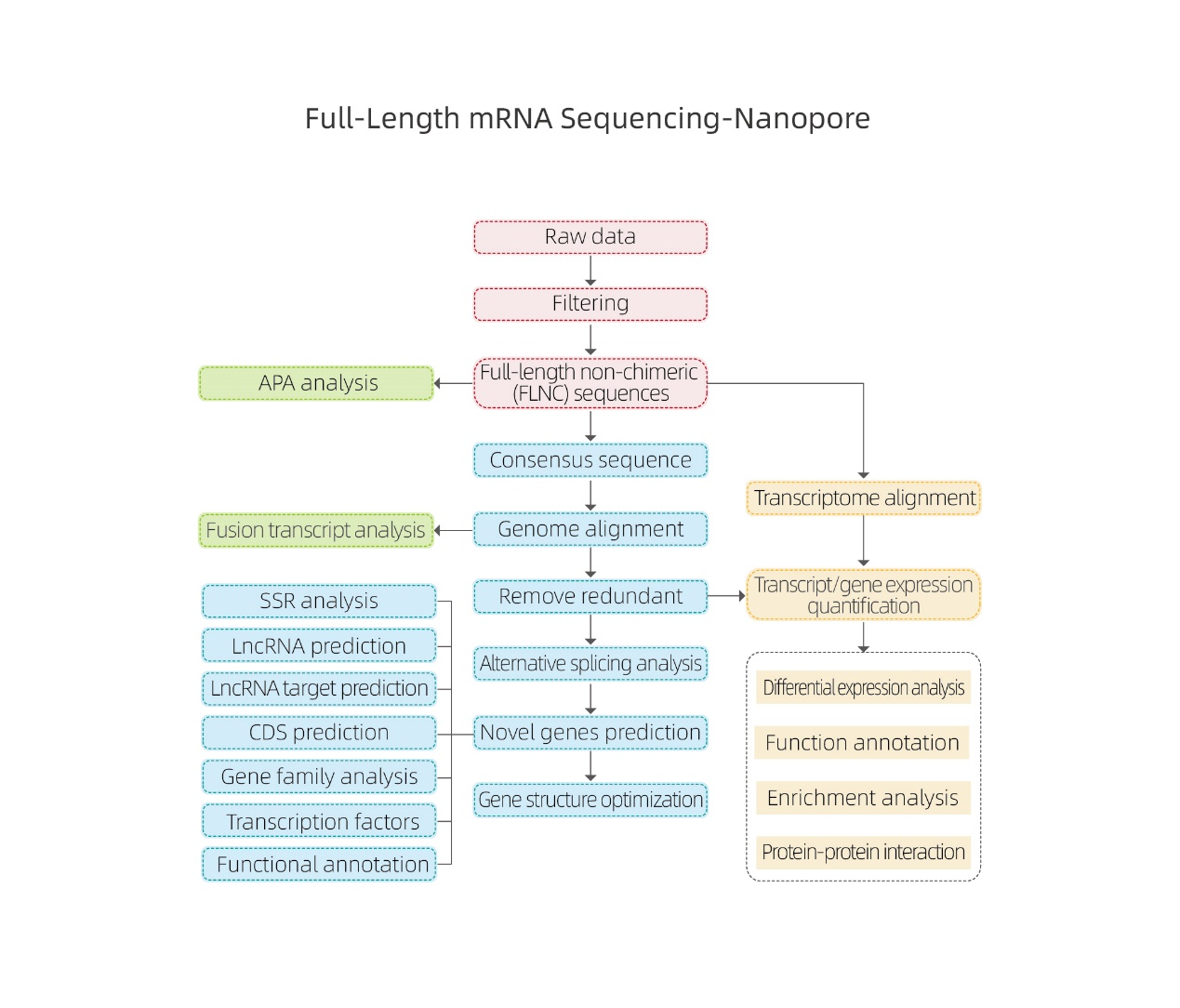
Біяінфарматычныя аналізы
●Апрацоўка неапрацаваных дадзеных
● Ідэнтыфікацыя стэнаграмы
● Альтэрнатыўнае зрошчванне
● Колькасная ацэнка экспрэсіі на ўзроўні гена і ўзроўню ізаформы
● Дыферэнцыяльны аналіз выразаў
● Анатацыя функцый і ўзбагачэнне (DEG і DET)
Узоры патрабаванняў і пастаўка
Узоры патрабаванняў:
Нуклеатыды:
| Канц. (нг/мкл) | Колькасць (мкг) | Чысціня | Сумленнасць |
| ≥ 100 | ≥ 0,6 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Забруджванне бялком або ДНК на гелі абмежавана або адсутнічае. | Для раслін: RIN≥7,0; Для жывёл: RIN≥7,5; 5,0≥28S/18S≥1,0; абмежаванае павышэнне зыходнага ўзроўню або яго адсутнасць |
Тканіна: Вага (сухі): ≥1 г
*Для тканіны, меншай за 5 мг, мы рэкамендуем адправіць хутка замарожаны (у вадкім азоце) узор тканіны.
Завісь клетак: колькасць клетак = 3×106- 1×107
*Мы рэкамендуем адпраўляць замарожаны лізат клетак.У выпадку, калі колькасць клетак менш 5×105, рэкамендуецца хуткая замарозка ў вадкім азоце, якая з'яўляецца пераважнай для мікраэкстракцыі.
Узоры крыві: Аб'ём≥1 мл
Рэкамендаваны ўзор дастаўкі
Кантэйнер: цэнтрыфужная прабірка аб'ёмам 2 мл (фальга не рэкамендуецца)
Прыклад маркіроўкі: група+рэплікацыя, напрыклад, A1, A2, A3;B1, B2, B3... ...
Адгрузка: 2、Сухі лёд: Узоры трэба спакаваць у мяшкі і пахаваць у сухім лёдзе.
- Прабіркі для стабілізацыі РНК: узоры РНК можна высушыць у прабірцы для стабілізацыі РНК (напрыклад, RNAstable®) і адправіць пры пакаёвай тэмпературы.
Працэдура абслугоўвання
Нуклеатыды:

Дастаўка ўзору

Будаўніцтва бібліятэкі

Секвеніраванне

Аналіз дадзеных

Пасляпродажнае абслугоўванне
Працэдура абслугоўвання
Тканіна:

Дызайн эксперыменту

Дастаўка ўзору

Вылучэнне РНК

Будаўніцтва бібліятэкі

Секвеніраванне

Аналіз дадзеных

Пасляпродажнае абслугоўванне
1.Аналіз дыферэнцыяльнага выразу - графік вулкана
Дыферэнцыяльны аналіз экспрэсіі можа быць апрацаваны як на генным узроўні для ідэнтыфікацыі генаў з дыферэнцыяльнай экспрэсіяй (DEG), так і на ўзроўні ізаформы для дыферэнцыяльнай ідэнтыфікацыі
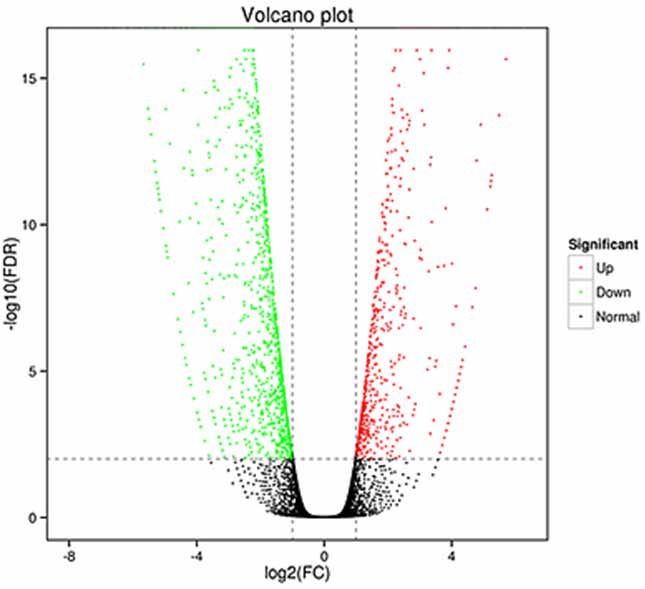
экспрэсіўныя стэнаграмы (DET)
2.Цеплавая карта іерархічнай кластарызацыі
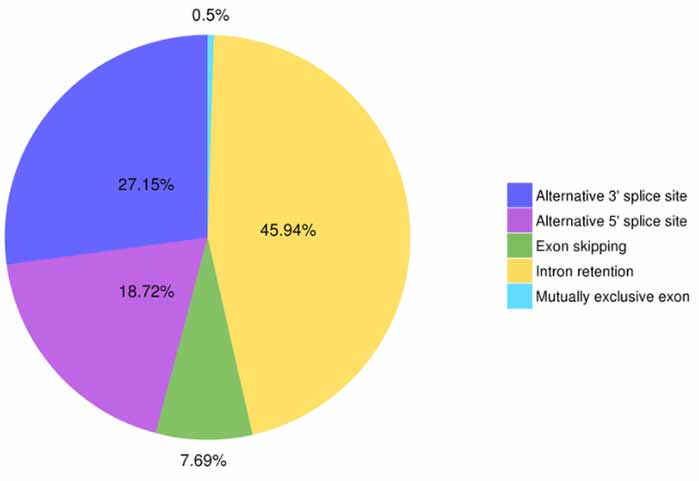
3.Альтэрнатыўная ідэнтыфікацыя і класіфікацыя сплайсінгу
Astalavista можа прадказаць пяць тыпаў альтэрнатыўных падзей сплайсінгу.
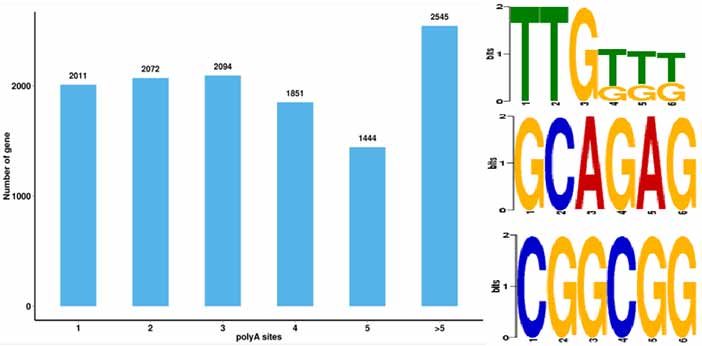
4.Ідэнтыфікацыя падзей альтэрнатыўнага поліадэнілавання (APA) і Motif на 50 bp перад полі-A
Справа БМК
Альтэрнатыўная ідэнтыфікацыя сплайсінгу і колькасная ацэнка на ўзроўні ізаформы шляхам секвеніравання поўнаразмернага транскрыптому нанапор
Апублікавана:Nature Communications, 2020
Стратэгія паслядоўнасці:
Групоўка: 1. CLL-SF3B1(WT);2. CLL-SF3B1(мутацыя K700E);3. Нармальныя В-клеткі
Стратэгія секвенирования: секвенирование бібліятэкі MinION 2D, секвенирование бібліятэкі PromethION 1D;кароткае чытанне дадзеных з тых жа ўзораў
Платформа секвенирования: Nanopore MinION;Нанапор PromethION;
Асноўныя вынікі
1. Альтэрнатыўная ідэнтыфікацыя сплайсінгу на ўзроўні ізаформы
Паслядоўнасці доўгага чытання дазваляюць ідэнтыфікаваць мутантны SF3B1K700E-змененыя месцы зрошчвання на ўзроўні ізаформы.Было выяўлена, што 35 альтэрнатыўных 3′SS і 10 альтэрнатыўных 5′SS значна адрозніваюцца паміж SF3B1K700Eі SF3B1WT.33 з 35 змяненняў былі нядаўна выяўленыя паслядоўнасцямі доўгага чытання.
2.Колькасная ацэнка альтэрнатыўнага сплайсінгу на ўзроўні ізаформы
Экспрэсія ізаформ затрымкі інтрона (IR) у SF3B1K700Eі SF3B1WTбылі колькасна ацэненыя на аснове паслядоўнасцей нанапор, выяўляючы глабальнае зніжэнне ВК-ізаформ у SF3B1K700E.
Даведка
Tang AD, Soulette CM, Baren MJV і інш.Характарыстыка поўнаметражнага стэнаграмы мутацыі SF3B1 пры хранічным лімфацытарным лейкозе выяўляе зніжэнне рэгуляцыі захаваных інтронаў [J].Камунікацыі прыроды.