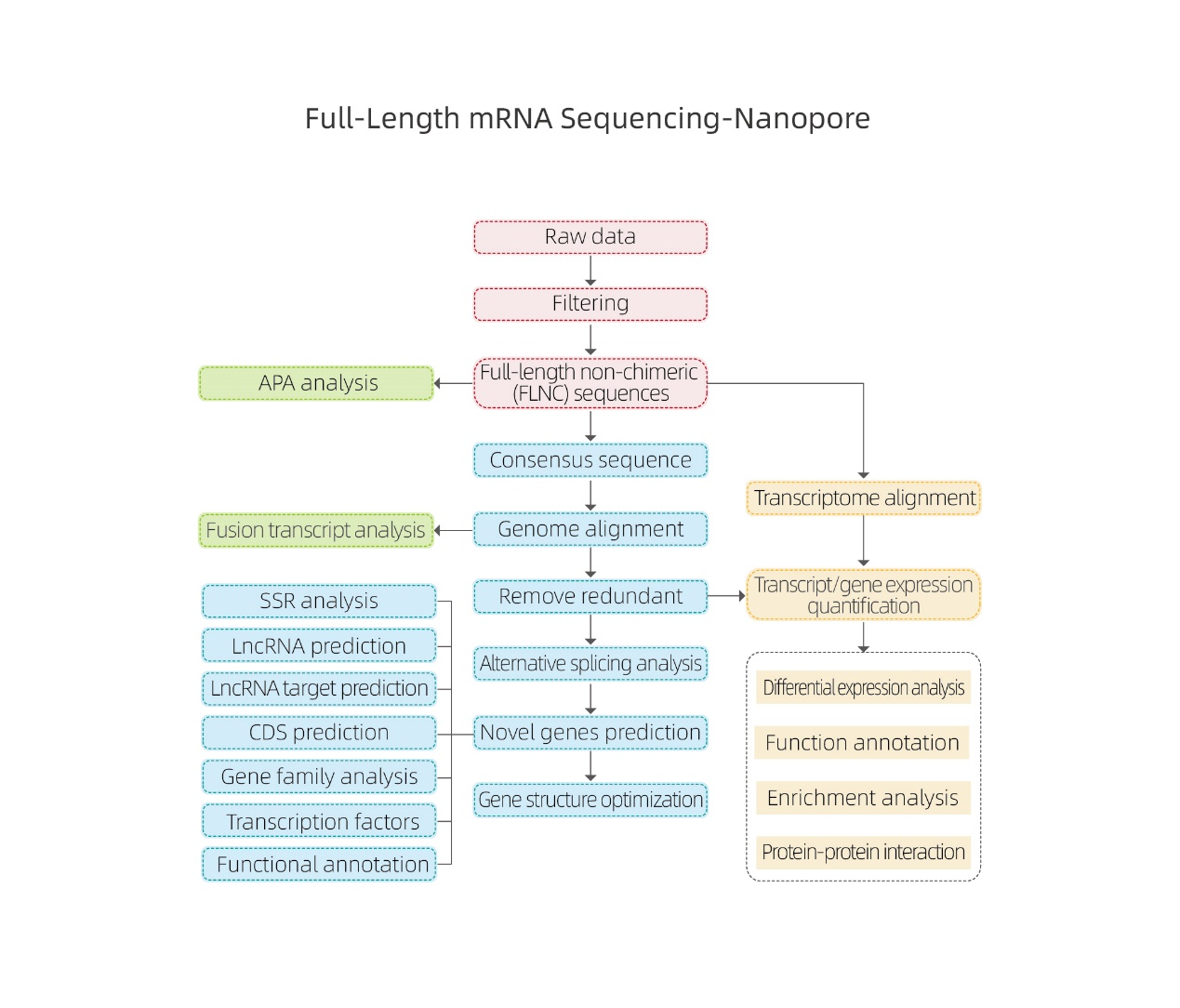
Secuenciación de ARNm de longitud completa: nanoporos
Ventajas del servicio
● Sesgo de secuencia bajo
● Revelar moléculas de ADNc de longitud completa
● Se requieren menos datos para cubrir la misma cantidad de transcripciones
● Identificación de múltiples isoformas por gen.
● Cuantificación de expresiones a nivel de isoformas.
Especificaciones de servicio
| Biblioteca | Plataforma | Rendimiento de datos recomendado (Gb) | Control de calidad |
| ADNc-PCR (enriquecido con poli-A) | Nanoporo PromethION P48 | 6 Gb/muestra (Según especie) | Proporción de longitud completa>70% Puntuación de calidad media: Q10
|
Análisis bioinformáticos.
●Procesamiento de datos sin procesar
● Identificación de transcripción
● Empalme alternativo
● Cuantificación de expresión a nivel de gen y nivel de isoforma.
● Análisis de expresión diferencial
● Anotación y enriquecimiento de funciones (DEG y DET)
Requisitos de muestra y entrega
Requisitos de muestra:
Nucleótidos:
| Conc.(ng/μl) | Cantidad (μg) | Pureza | Integridad |
| ≥ 100 | ≥ 0,6 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 En el gel se muestra contaminación limitada o nula de proteínas o ADN. | Para plantas: RIN≥7,0; Para animales: RIN≥7,5; 5,0≥28S/18S≥1,0; elevación basal limitada o nula |
Tejido: Peso (seco): ≥1 g
*Para tejido de menos de 5 mg, recomendamos enviar una muestra de tejido congelada instantáneamente (en nitrógeno líquido).
Suspensión celular: Recuento de células = 3×106- 1×107
*Recomendamos enviar lisado celular congelado.En caso de que la celda cuente menos de 5×105, se recomienda la congelación instantánea en nitrógeno líquido, que es preferible para la microextracción.
Muestras de sangre: Volumen≥1 ml
Entrega de muestra recomendada
Envase: tubo de centrífuga de 2 ml (no se recomienda papel de aluminio)
Etiquetado de muestras: Grupo+réplica, por ejemplo, A1, A2, A3;B1, B2, B3... ...
Envío: 2. Hielo seco: las muestras deben empaquetarse en bolsas y enterrarse en hielo seco.
- Tubos RNAstable: las muestras de ARN se pueden secar en un tubo de estabilización de ARN (por ejemplo, RNAstable®) y enviarse a temperatura ambiente.
Flujo de trabajo del servicio
Nucleótidos:

Entrega de muestra

construcción de biblioteca

Secuenciación

Análisis de los datos

Servicios postventa
Flujo de trabajo del servicio
Tejido:

Diseño de experimentos

Entrega de muestra

extracción de ARN

construcción de biblioteca

Secuenciación

Análisis de los datos

Servicios postventa
1.Análisis de expresión diferencial -Gráfico de volcán
El análisis de expresión diferencial se puede procesar tanto a nivel genético para identificar genes expresados diferencialmente (DEG) como a nivel de isoformas para identificar diferencialmente
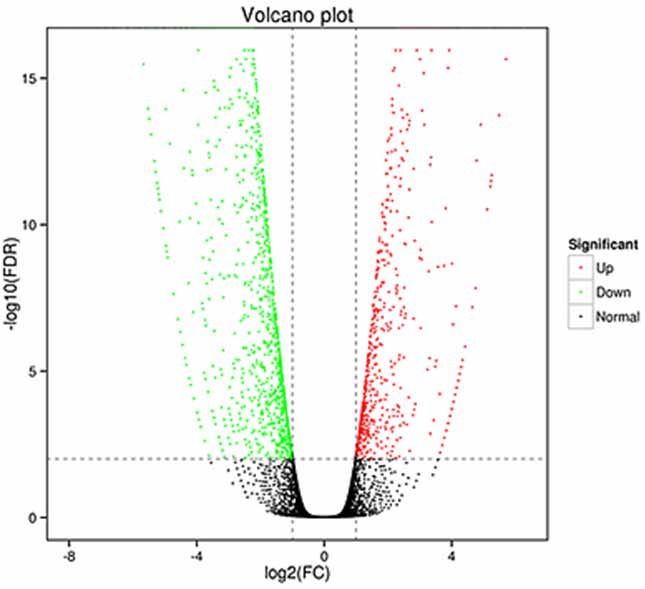
transcripciones expresadas (DET)
2.Mapa de calor de agrupación jerárquica
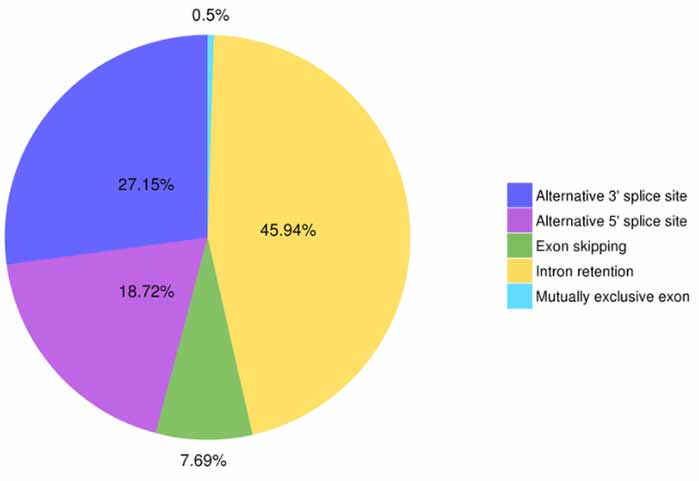
3.Identificación y clasificación de empalmes alternativos.
Astalavista puede predecir cinco tipos de eventos de empalme alternativos.
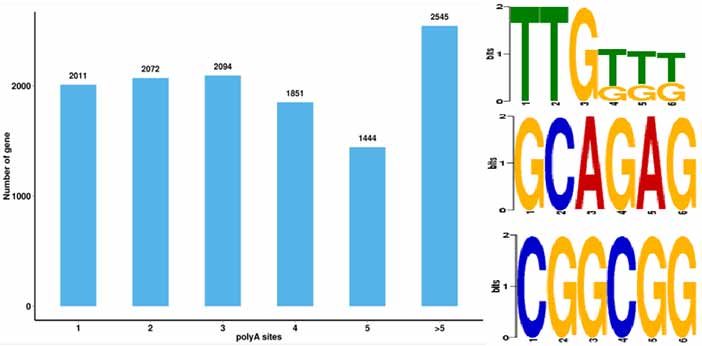
4.Identificación de eventos de poliadenilación alternativa (APA) y motivo a 50 pb aguas arriba de poli-A
Caso BMK
Identificación de empalme alternativo y cuantificación a nivel de isoformas mediante secuenciación del transcriptoma de longitud completa de nanoporos
Publicado:Comunicaciones de la naturaleza, 2020
Estrategia de secuenciación:
Agrupación: 1. CLL-SF3B1(WT);2. CLL-SF3B1 (mutación K700E);3. Células B normales
Estrategia de secuenciación: secuenciación de la biblioteca MinION 2D, secuenciación de la biblioteca PromethION 1D;datos de lectura corta de las mismas muestras
Plataforma de secuenciación: Nanopore MinION;PromethION de nanoporos;
Resultados clave
1.Identificación de empalme alternativo a nivel de isoforma
Las secuencias de lectura larga permiten la identificación del mutante SF3B1K700E-sitios de empalme alterados a nivel de isoforma.Se encontró que 35 3′SS alternativos y 10 5′SS alternativos estaban empalmados de manera significativamente diferencial entre SF3B1K700Ey SF3B1WT.33 de las 35 alteraciones fueron descubiertas recientemente mediante secuencias de lectura larga.
2. Cuantificación del empalme alternativo a nivel de isoforma
Expresión de isoformas de retención de intrones (IR) en SF3B1K700Ey SF3B1WTse cuantificaron en función de secuencias de nanoporos, revelando una regulación negativa global de las isoformas de IR en SF3B1K700E.
Referencia
Tang AD, Soulette CM, Baren MJV, et al.La caracterización completa de la transcripción de la mutación SF3B1 en la leucemia linfocítica crónica revela una regulación negativa de los intrones retenidos [J].Comunicaciones de la naturaleza.