
16S/18S/ITS Амплікон секвенування-PacBio
Переваги сервісу

● Довгі читання, що розкривають повну послідовність 16S/18S/ITS
● Високоточний базовий виклик із секвенуванням режиму PacBio CCS
● Роздільна здатність на рівні виду в анотації OTU/ASV
● Найновіший потік аналізу QIIME2 із різними аналізами з точки зору бази даних, анотацій, OTU/ASV.
● Застосовується для різноманітних досліджень мікробної спільноти
● BMK володіє великим досвідом із понад 100 000 проб на рік,охоплюючи ґрунт, воду, газ, мул, фекалії, кишечник, шкіру, ферментаційний бульйон, комах, рослини тощо.
● BMKCloud сприяє інтерпретації даних, що містить 45 персоналізованих інструментів аналізу
Специфікації послуги
| СеквенуванняПлатформа | Бібліотека | Рекомендовані дані | Час обороту |
| PacBio Продовження II | СМРТ-дзвінок | Теги 5K/10K/20K | 44 робочих дні |
Біоінформаційні аналізи
● Контроль якості необроблених даних
● Кластеризація OTU/De-Noise (ASV)
● Анотація OTU
● Альфа-різноманіття
● Бета-різноманітність
● Міжгруповий аналіз
● Аналіз асоціацій проти експериментальних факторів
● Передбачення функції гена
Вимоги до зразків і доставка
Вимоги до зразків:
дляекстракти ДНК:
| Тип зразка | Сума | Концентрація | Чистота |
| екстракти ДНК | > 1 мкг | > 20 нг/мкл | OD260/280= 1,6-2,5 |
Для екологічних зразків:
| Тип зразка | Рекомендована процедура відбору проб |
| грунт | Кількість проби: прибл.5 г;Залишки засохлого речовини необхідно видалити з поверхні;Великі шматки подрібнити і пропустити через фільтр 2 мм;Аліквоти зразків у стерильній ЕР-пробірці або циропробірці для резервування. |
| Фекалії | Кількість проби: прибл.5 г;Зберіть та аліквотуйте зразки в стерильну EP-пробірку або кріопробірку для резервування. |
| Кишковий вміст | Зразки необхідно обробляти в асептичних умовах.Промийте зібрану тканину PBS;Центрифугуйте PBS і зберіть осад в EP-пробірки. |
| Шлам | Кількість проби: прибл.5 г;Зберіть і аліквотуйте зразок мулу в стерильну EP-пробірку або кріопробірку для резервування |
| Водний об'єкт | Для зразка з обмеженою кількістю мікроорганізмів, наприклад водопровідної води, колодязної води тощо, наберіть принаймні 1 л води та пропустіть її через фільтр 0,22 мкм, щоб збагатити мікроорганізми на мембрані.Зберігайте мембрану в стерильній пробірці. |
| Шкіра | Обережно поскрібайте поверхню шкіри стерильним ватним тампоном або хірургічним лезом і помістіть його в стерильну пробірку. |
Рекомендована доставка зразків
Заморожуйте зразки в рідкому азоті протягом 3-4 годин і зберігайте в рідкому азоті або -80 градусів для тривалого зберігання.Потрібна доставка зразків із сухим льодом.
Потік роботи служби

Доставка зразків

Будівництво бібліотеки

Секвенування

Аналіз даних

Післяпродажне обслуговування
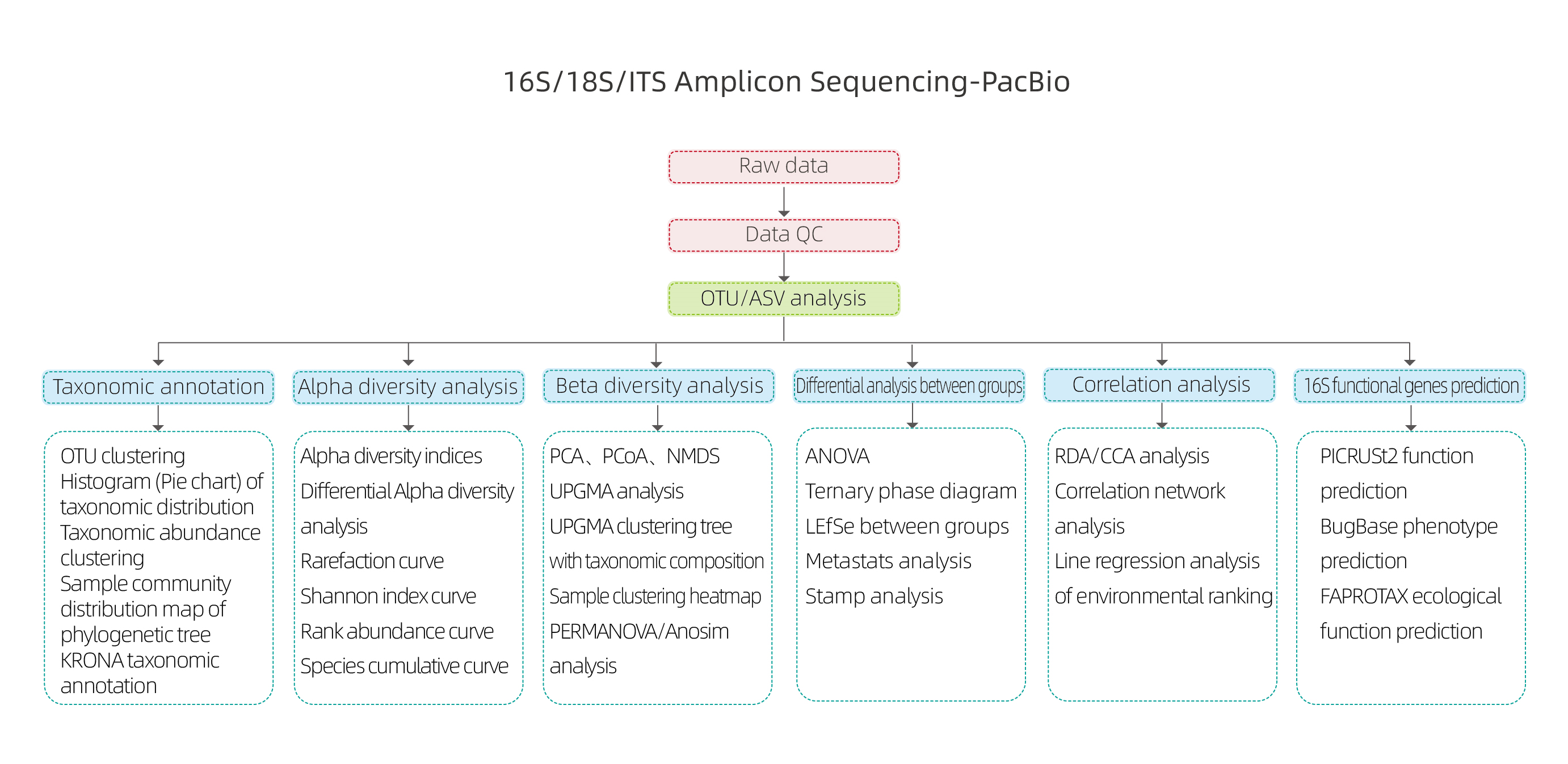
1. Коефіцієнт анотації профілювання мікробної спільноти на основі V3+V4 (Illumina) проти профілювання на основі повної довжини (PacBio).
(Для статистики застосовано дані 30 випадково вибраних проектів)
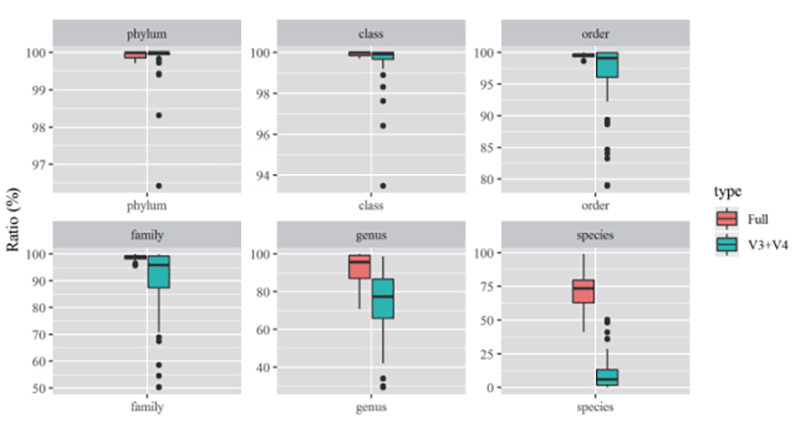
2. Швидкість анотації повнорозмірного секвенування ампліконів на видовому рівні в різних типах зразків
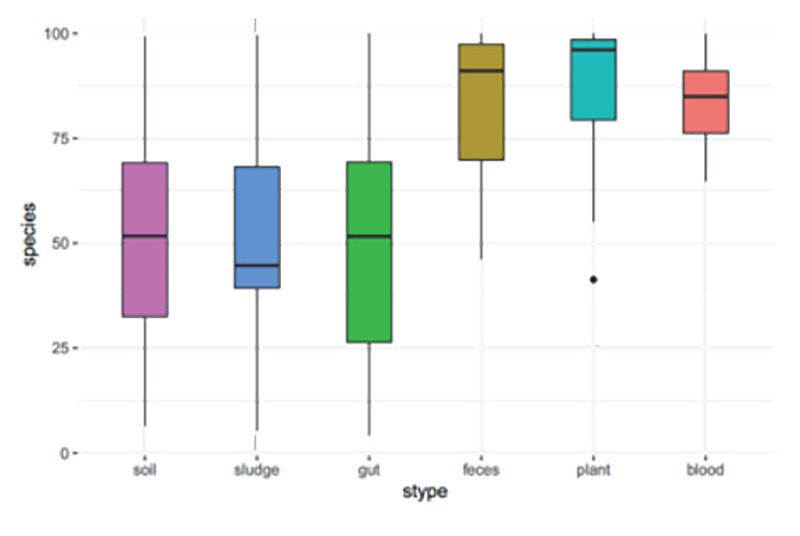
3.Поширення виду
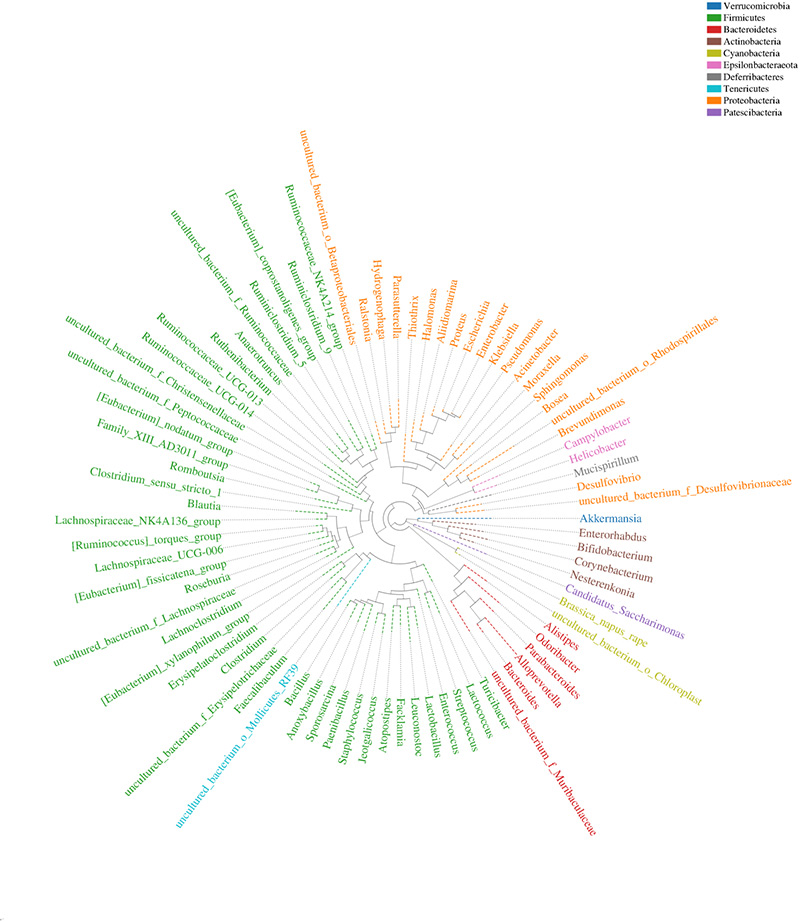
4.Філогенетичне дерево
Корпус БМК
Вплив миш’яку викликає пошкодження кишкового бар’єру та подальшу активацію осі кишківник-печінка, що призводить до запалення та піроптозу печінки у качок
Опубліковано:Наука про загальне середовище,2021 рік
Стратегія послідовності:
Зразки: контроль проти 8 мг/кг ATO групи, що піддавалася впливу
Вихід даних секвенування: загалом 102 583 необроблених послідовностей CCS
Контроль: 54 518 ± 747 ефективних CCS
Під впливом АТО: 45 050 ± 1675 ефективних CCS
Ключові результати
Альфа-різноманіття:Вплив АТО суттєво змінив насиченість і різноманітність кишкових мікробів у качок.
Аналіз метастатів:
На рівні типу: 2 типи бактерій виявлені лише в контрольних групах
На рівні роду: було знайдено 6 родів, що значно відрізняються відносною чисельністю
На видовому рівні: загалом ідентифіковано 36 видів, 6 з яких значно відрізняються відносною чисельністю
довідка
Thingholm, LB, et al.«Особи з ожирінням із діабетом 2 типу та без нього демонструють різну функціональну здатність і склад кишкових мікробів».Клітинний господар і мікроб26.2 (2019).











