
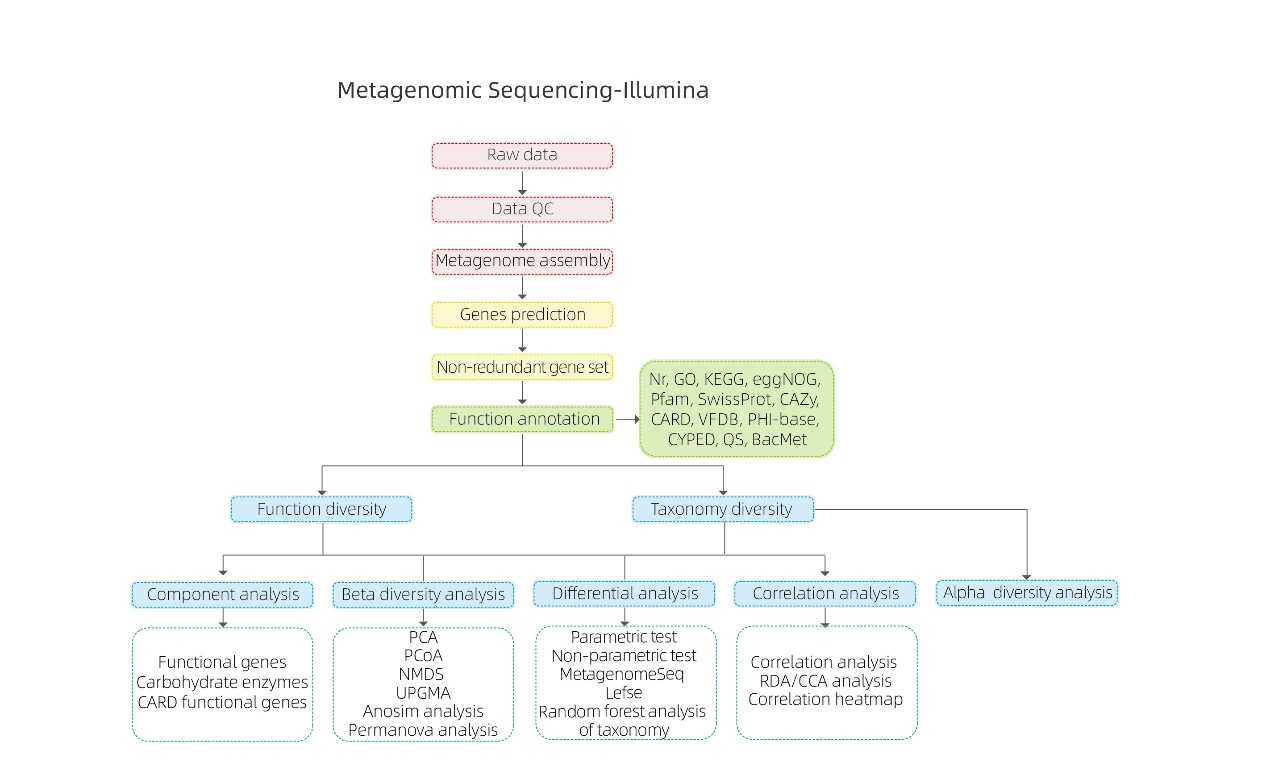
Sequenciamento Metagenômico -NGS
Vantagens do serviço
● Isolamento e livre de cultivo para perfilamento de comunidades microbianas
● Alta resolução na detecção de espécies de baixa abundância em amostras ambientais
● A ideia de “meta” integra todas as características biológicas ao nível funcional, ao nível da espécie e ao nível do gene, o que reflecte uma visão dinâmica e mais próxima da realidade.
● A BMK acumula enorme experiência em diversos tipos de amostras, com mais de 10.000 amostras processadas.
Especificações de serviço
| Plataforma | Sequenciamento | Dados recomendados | Tempo de resposta |
| Plataforma Illumina NovaSeq | PE150 | 6g/10g/20g | 45 dias úteis |
Análises de bioinformática
● Controle de qualidade de dados brutos
● Montagem do metagenoma
● Conjunto de genes e anotação não redundantes
● Análise da diversidade de espécies
● Análise de diversidade de funções genéticas
● Análise intergrupo
● Análise de associação contra fatores experimentais
Requisitos de amostra e entrega
Requisitos de amostra:
ParaExtratos de DNA:
| Tipo de amostra | Quantia | Concentração | Pureza |
| Extratos de DNA | > 30 ng | > 1 ng/μl | DO260/280 = 1,6-2,5 |
Para amostras ambientais:
| Tipo de amostra | Procedimento de amostragem recomendado |
| Solo | Quantidade de amostragem: aprox.5g;A substância murcha restante precisa ser removida da superfície;Triture pedaços grandes e passe por filtro de 2 mm;Alíquotas de amostras em tubo EP estéril ou tubo ciro para reserva. |
| Fezes | Quantidade de amostragem: aprox.5g;Coletar e alíquotar amostras em tubo EP estéril ou criotubo para reserva. |
| Conteúdo intestinal | As amostras precisam ser processadas sob condições assépticas.Lave o tecido coletado com PBS;Centrifugue o PBS e colete o precipitante em tubos EP. |
| Lodo | Quantidade de amostragem: aprox.5g;Coletar e alíquotar amostra de lodo em tubo EP estéril ou criotubo para reserva |
| Corpo d'água | Para amostras com quantidade limitada de micróbios, como água de torneira, água de poço, etc., colete pelo menos 1 L de água e passe por um filtro de 0,22 μm para enriquecer micróbios na membrana.Armazene a membrana em tubo estéril. |
| Pele | Raspe cuidadosamente a superfície da pele com um cotonete estéril ou lâmina cirúrgica e coloque-a em um tubo estéril. |
Entrega de amostra recomendada
Congele as amostras em nitrogênio líquido por 3-4 horas e armazene em nitrogênio líquido ou -80 graus para reserva de longo prazo.É necessário enviar amostras com gelo seco.
Fluxo de trabalho de serviço

Entrega de amostra

Construção de biblioteca

Sequenciamento

Análise de dados

Serviços pós-venda
1.Histograma: Distribuição de espécies
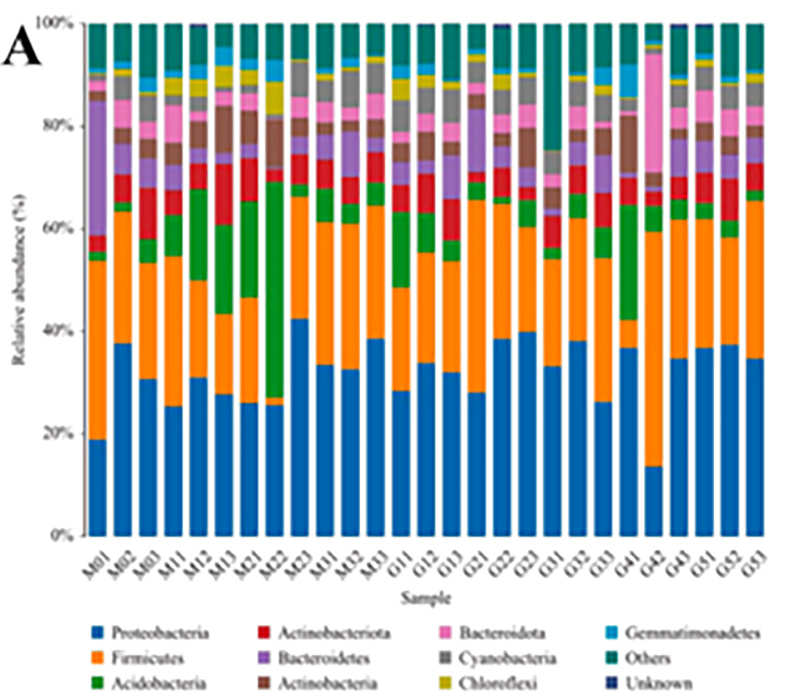
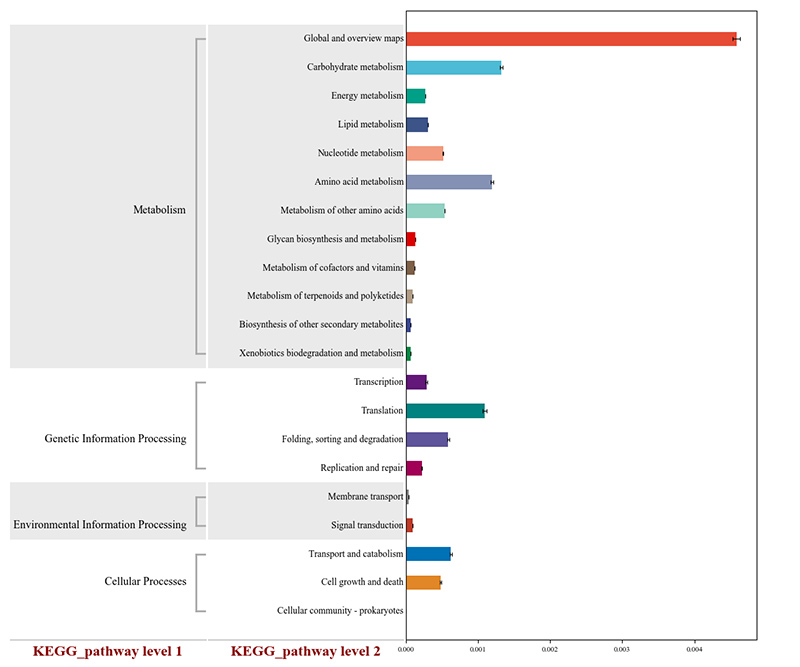
2.Genes funcionais anotados para vias metabólicas KEGG
3.Mapa de calor: Funções diferenciais baseadas na abundância relativa de genes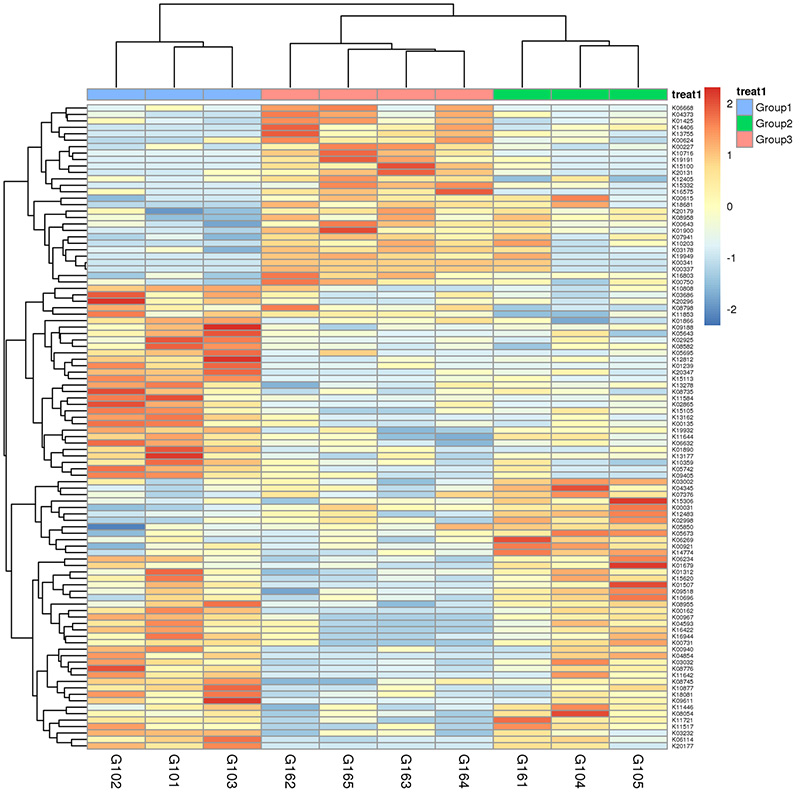 4.Circos de genes de resistência a antibióticos CARD
4.Circos de genes de resistência a antibióticos CARD
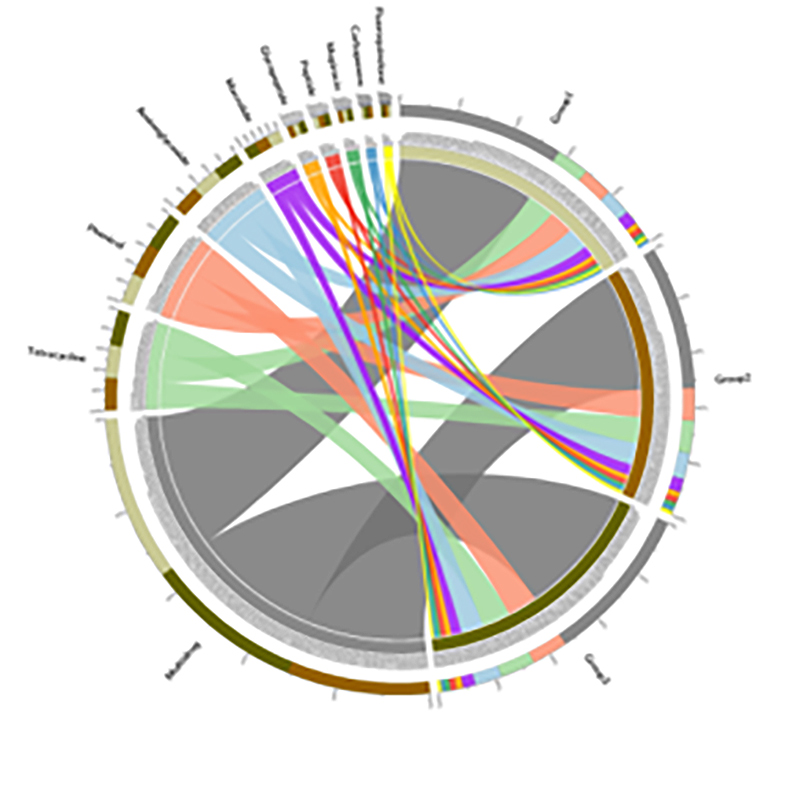
Caso BMK
Prevalência de genes de resistência a antibióticos e patógenos bacterianos ao longo do continuum solo-raiz de mangue
Publicados:Diário de Materiais Perigosos, 2021
Estratégia de sequenciamento:
Materiais: Extratos de DNA de quatro fragmentos de amostras associadas a raízes de mangue: solo não plantado, compartimentos de rizosfera, epísfera e endosfera
Plataforma: Illumina HiSeq 2500
Alvos: Metagenoma
Região V3-V4 do gene 16S rRNA
Principais resultados
O sequenciamento metagenômico e o perfil de metabarcoding no continuum solo-raiz de mudas de mangue foram processados para estudar a disseminação de genes de resistência a antibióticos (ARGs) do solo para as plantas.Dados metagenômicos revelaram que 91,4% dos genes de resistência a antibióticos foram comumente identificados em todos os quatro compartimentos do solo mencionados acima, o que mostrou uma tendência contínua.O sequenciamento do amplicon 16S rRNA gerou 29.285 sequências, representando 346 espécies.Combinando com o perfil de espécies por sequenciação de amplicon, esta disseminação foi considerada independente da microbiota associada à raiz, no entanto, poderia ser facilitada pela mobilidade de elementos genéticos.Este estudo identificou o fluxo de ARGs e patógenos do solo para as plantas através de um continuum solo-raiz interconectado.
Referência
Wang, C., Hu, R., Strong, PJ, Zhuang, W. e Shu, L..(2020).Prevalência de genes de resistência a antibióticos e patógenos bacterianos ao longo do continuum solo-raiz de mangue.Jornal de Materiais Perigosos, 408, 124985.











