
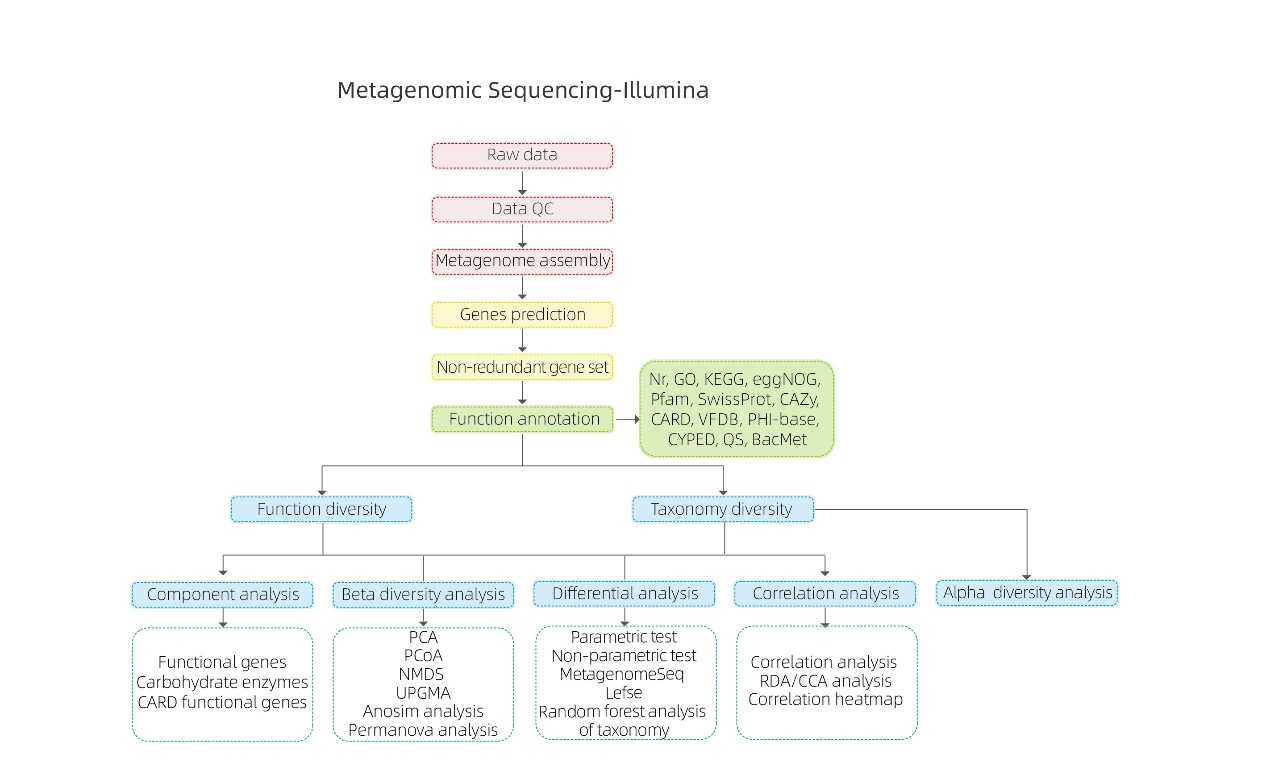
Secuenciación metaxenómica -NGS
Vantaxes do servizo
● Libre de illamento e cultivo para o perfil de comunidades microbianas
● Alta resolución na detección de especies de pouca abundancia en mostras ambientais
● A idea de “meta-” integra todas as características biolóxicas a nivel funcional, de especie e de xene, o que reflicte unha visión dinámica máis próxima á realidade.
● BMK acumula unha enorme experiencia en diversos tipos de mostras con máis de 10.000 mostras procesadas.
Especificacións do servizo
| Plataforma | Secuenciación | Datos recomendados | Tempo de resposta |
| Plataforma Illumina NovaSeq | PE150 | 6 G/10 G/20 G | 45 días laborables |
Análise bioinformática
● Control de calidade dos datos brutos
● Montaxe do metaxenoma
● Conxunto de xenes non redundantes e anotación
● Análise da diversidade de especies
● Análise da diversidade de funcións xenéticas
● Análise intergrupal
● Análise de asociación fronte a factores experimentais
Requisitos de mostra e entrega
Requisitos de mostra:
ParaExtractos de ADN:
| Tipo de mostra | Cantidade | Concentración | Pureza |
| Extractos de ADN | > 30 ng | > 1 ng/μl | OD260/280= 1,6-2,5 |
Para mostras ambientais:
| Tipo de mostra | Procedemento de mostraxe recomendado |
| Solo | Cantidade da mostra: aprox.5 g;A substancia murcha restante debe ser eliminada da superficie;Moer pezas grandes e pasar por un filtro de 2 mm;Mostras alícuotas en tubo EP estéril ou cirotubo para reserva. |
| Feces | Cantidade da mostra: aprox.5 g;Recoller e alícuotar mostras en tubo EP estéril ou criotubo para reserva. |
| Contidos intestinais | As mostras deben procesarse en condicións asépticas.Lavar o tecido recollido con PBS;Centrifugar o PBS e recoller o precipitante en tubos EP. |
| Lodos | Cantidade da mostra: aprox.5 g;Recollida e alícuota de mostra de lodo en tubo EP estéril ou criotubo para reserva |
| Corpo de auga | Para mostras con cantidade limitada de microbios, como auga da billa, auga de pozo, etc., Recolla polo menos 1 L de auga e pásase por un filtro de 0,22 μm para enriquecer os microbios na membrana.Almacene a membrana nun tubo estéril. |
| Pel | Raspe coidadosamente a superficie da pel cun cotonete estéril ou unha lámina cirúrxica e colócaa nun tubo estéril. |
Entrega de mostras recomendada
Conxelar as mostras en nitróxeno líquido durante 3-4 horas e almacenar en nitróxeno líquido ou -80 graos para reservar a longo prazo.É necesario o envío de mostras con xeo seco.
Fluxo de traballo do servizo

Entrega da mostra

Construción da biblioteca

Secuenciación

Análise de datos

Servizos posvenda
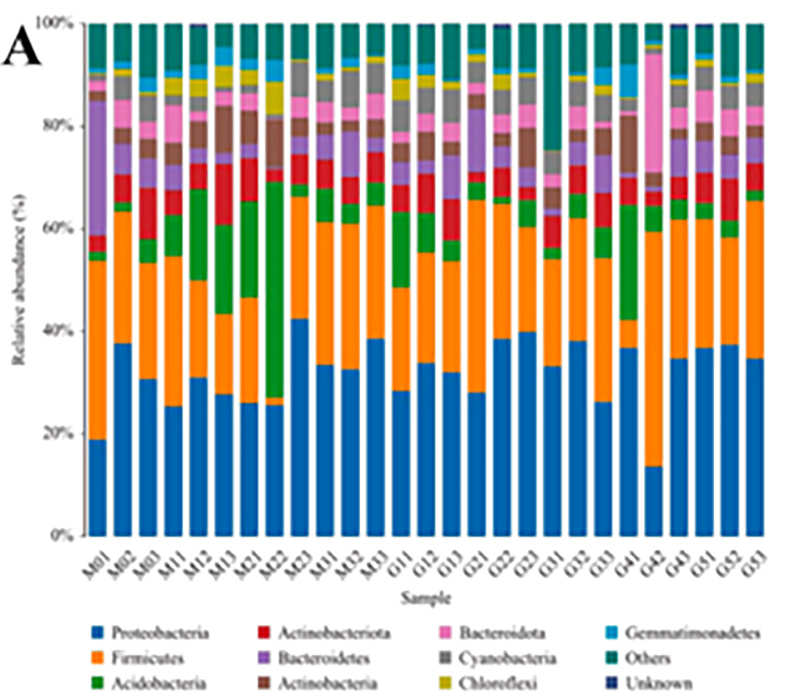
1.Histograma: distribución das especies
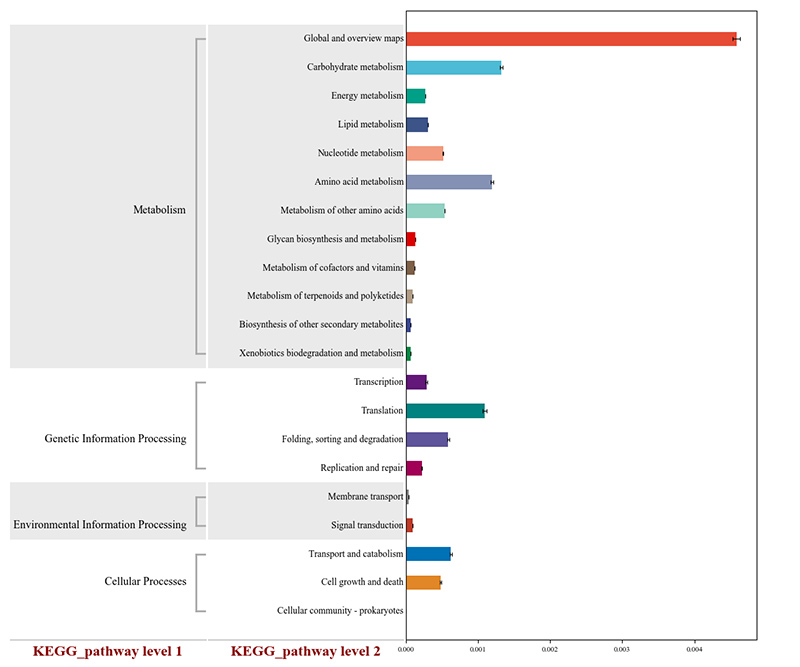
2.Xenes funcionais anotados nas vías metabólicas KEGG
3.Mapa térmico: funcións diferenciais baseadas na abundancia relativa dos xenes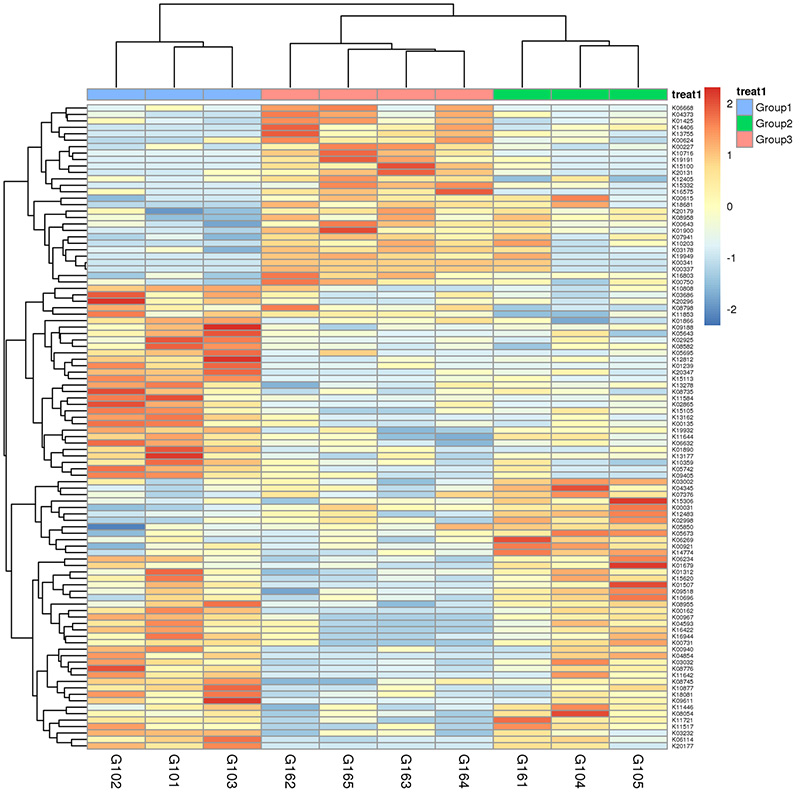 4.Circos de xenes de resistencia a antibióticos CARD
4.Circos de xenes de resistencia a antibióticos CARD
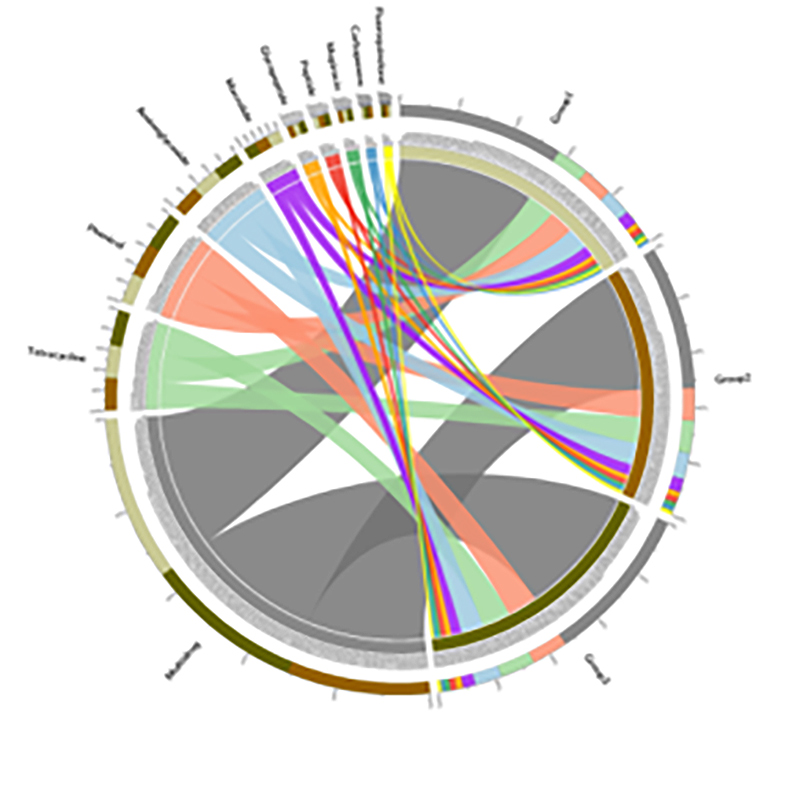
Caso BMK
Prevalencia de xenes de resistencia a antibióticos e patóxenos bacterianos ao longo do continuo chan-raíz de mangle
Publicado:Journal of Hazardous Materials, 2021
Estratexia de secuenciación:
Materiais: extractos de ADN de catro fragmentos de mostras asociadas á raíz de mangle: solo non plantado, rizosfera, episfera e endosfera.
Plataforma: Illumina HiSeq 2500
Obxectivos: Metaxenoma
Rexión V3-V4 do xene do ARNr 16S
Resultados clave
Procesouse a secuenciación metaxenómica e o perfil de metabarcodificación no continuo solo-raíz de mudas de mangle para estudar a diseminación de xenes de resistencia a antibióticos (ARG) do solo ás plantas.Os datos metaxenómicos revelaron que o 91,4% dos xenes de resistencia a antibióticos identificáronse habitualmente nos catro compartimentos do solo mencionados anteriormente, o que mostrou unha moda continua.A secuenciación do amplicón do ARNr 16S xerou 29.285 secuencias, que representan 346 especies.Ao combinarse co perfís de especies mediante a secuenciación de amplicóns, descubriuse que esta diseminación era independente da microbiota asociada á raíz, pero podería ser facilitada por elementos xenéticos móbiles.Este estudo identificou o fluxo de ARG e patóxenos do solo ás plantas a través do continuo interconectado solo-raíz.
Referencia
Wang, C., Hu, R., Strong, PJ, Zhuang, W. e Shu, L. .(2020).Prevalencia de xenes de resistencia a antibióticos e patóxenos bacterianos ao longo do continuo chan-raíz de mangle.Revista de materiais perigosos, 408, 124985.











