
Secuenciación Metagenómica -NGS
Ventajas del servicio
● Aislamiento y libre de cultivo para la elaboración de perfiles de comunidades microbianas.
● Alta resolución en la detección de especies de baja abundancia en muestras ambientales.
● La idea de “meta” integra todas las características biológicas a nivel funcional, de especie y de gen, lo que refleja una visión dinámica que se acerca más a la realidad.
● BMK acumula una enorme experiencia en diversos tipos de muestras con más de 10.000 muestras procesadas.
Especificaciones de servicio
| Plataforma | Secuenciación | Datos recomendados | tiempo de respuesta |
| Plataforma Illumina NovaSeq | PE150 | 6G/10G/20G | 45 días laborables |
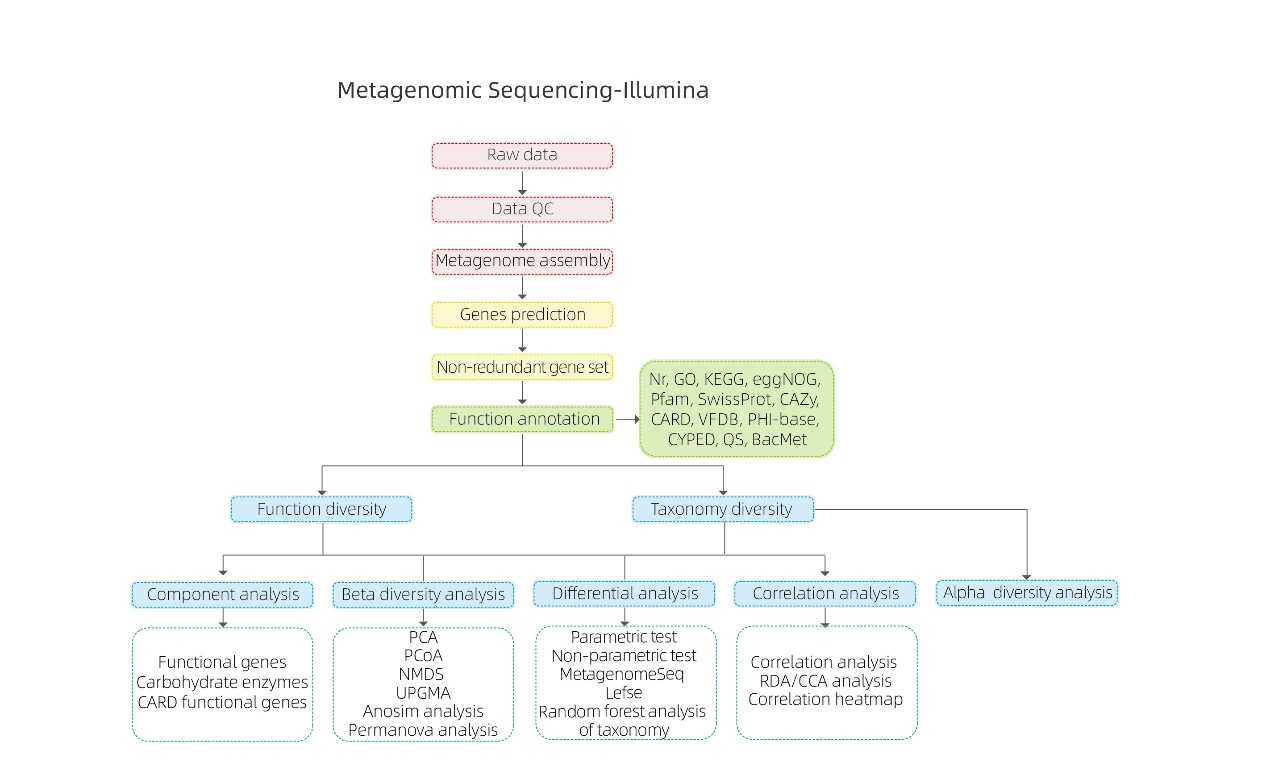
Análisis bioinformáticos.
● Control de calidad de los datos sin procesar
● Ensamblaje del metagenoma
● Conjunto de genes y anotaciones no redundantes
● Análisis de diversidad de especies.
● Análisis de diversidad de funciones genéticas.
● Análisis intergrupal
● Análisis de asociación frente a factores experimentales.
Requisitos de muestra y entrega
Requisitos de muestra:
Paraextractos de ADN:
| Tipo de ejemplo | Cantidad | Concentración | Pureza |
| extractos de ADN | > 30 ng | > 1 ng/μl | DO260/280= 1,6-2,5 |
Para muestras ambientales:
| Tipo de ejemplo | Procedimiento de muestreo recomendado |
| Suelo | Cantidad de muestra: aprox.5 gramos;Es necesario eliminar de la superficie los restos de sustancia marchita;Moler trozos grandes y pasar por filtro de 2 mm;Muestras alícuotas en tubo EP o criotubo estéril para reserva. |
| Heces | Cantidad de muestra: aprox.5 gramos;Recoja y haga alícuotas de muestras en un tubo EP o criotubo estéril para reservarlas. |
| Contenido intestinal | Las muestras deben procesarse en condiciones asépticas.Lavar el tejido recogido con PBS;Centrifugar el PBS y recoger el precipitante en tubos EP. |
| Lodo | Cantidad de muestra: aprox.5 gramos;Recoger y tomar alícuotas de la muestra de lodo en un tubo EP o criotubo estéril para reservar |
| Agua corporal | Para muestras con una cantidad limitada de microbios, como agua del grifo, agua de pozo, etc., recolecte al menos 1 litro de agua y páselo a través de un filtro de 0,22 μm para enriquecer los microbios en la membrana.Guarde la membrana en un tubo estéril. |
| Piel | Raspe con cuidado la superficie de la piel con un hisopo de algodón esterilizado o un bisturí quirúrgico y colóquelo en un tubo esterilizado. |
Entrega de muestra recomendada
Congele las muestras en nitrógeno líquido durante 3 a 4 horas y guárdelas en nitrógeno líquido o a -80 grados para una reserva a largo plazo.Se requiere envío de muestras con hielo seco.
Flujo de trabajo del servicio

Entrega de muestra

construcción de biblioteca

Secuenciación

Análisis de los datos

Servicios postventa
1.Histograma: Distribución de especies
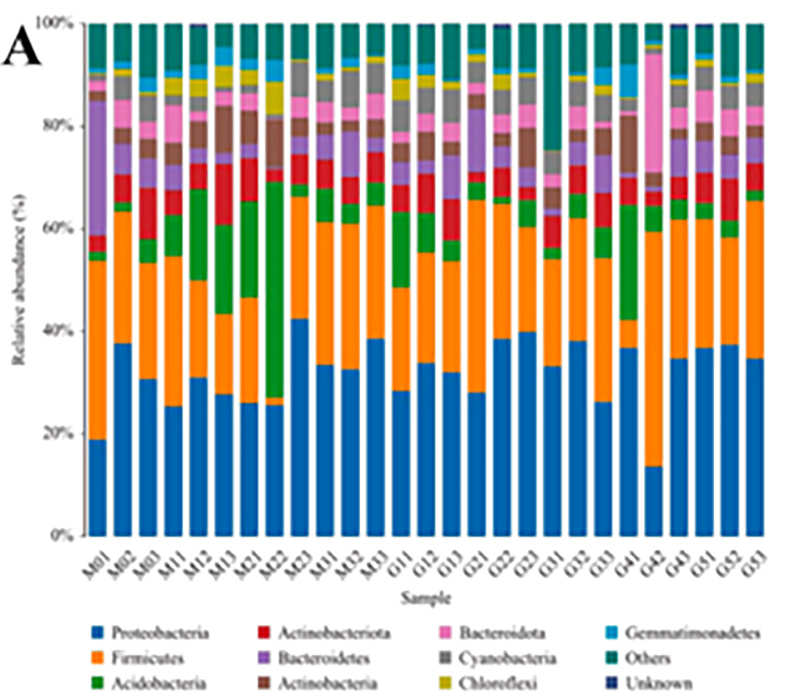
2.Genes funcionales anotados en las vías metabólicas de KEGG.
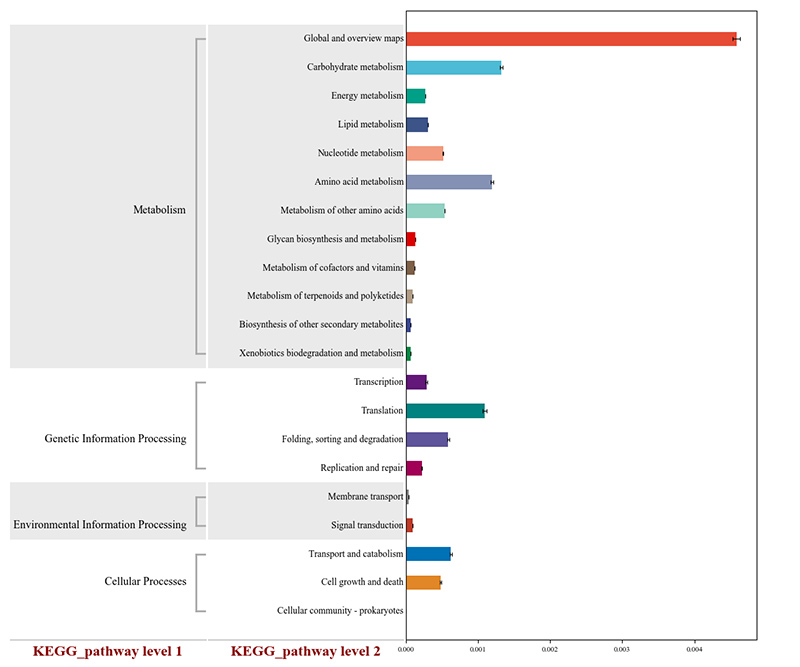
3.Mapa de calor: funciones diferenciales basadas en la abundancia relativa de genes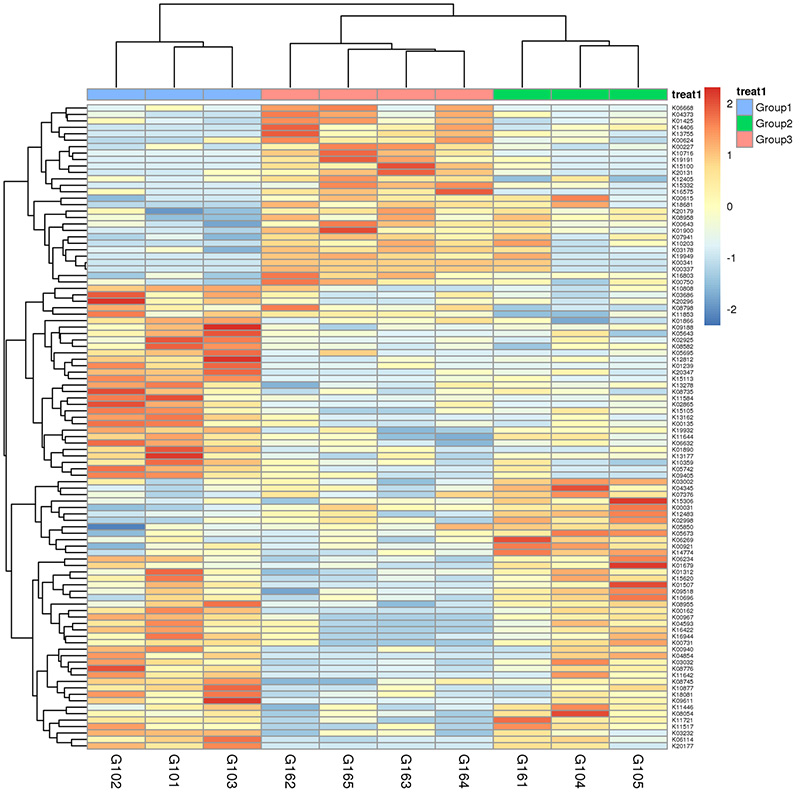 4.Circos de genes de resistencia a antibióticos CARD
4.Circos de genes de resistencia a antibióticos CARD
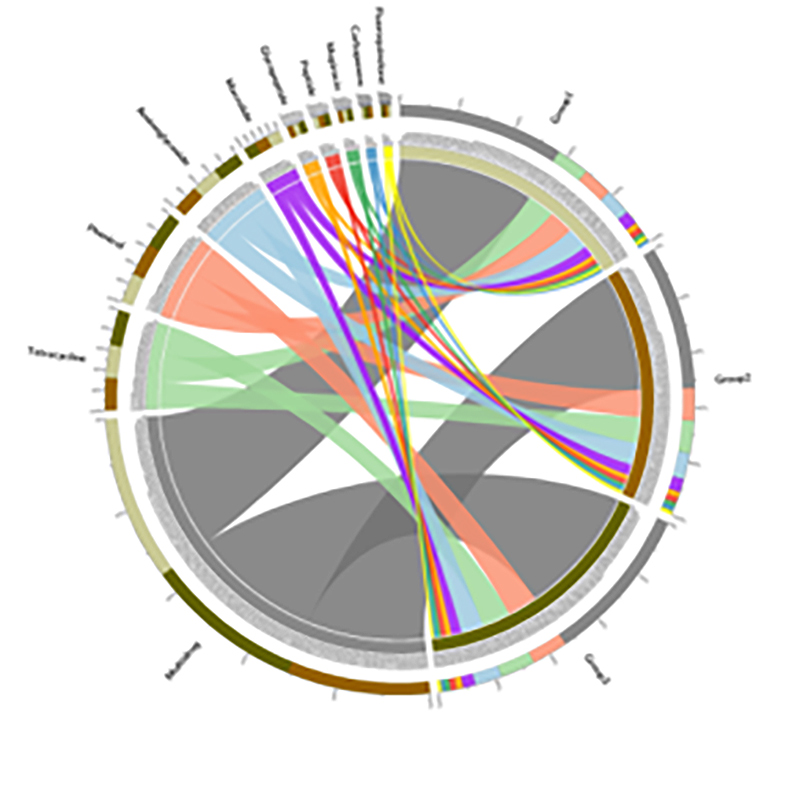
Caso BMK
Prevalencia de genes de resistencia a antibióticos y patógenos bacterianos a lo largo del continuo suelo-raíz de manglar
Publicado:Revista de materiales peligrosos, 2021
Estrategia de secuenciación:
Materiales: Extractos de ADN de cuatro fragmentos de muestras asociadas a raíces de mangle: suelo no plantado, compartimentos de rizosfera, episfera y endosfera.
Plataforma: Illumina HiSeq 2500
Objetivos: Metagenoma
Región V3-V4 del gen 16S rRNA
Resultados clave
Se procesaron secuenciaciones metagenómicas y perfiles de códigos de barras en el continuo suelo-raíz de árboles jóvenes de manglares para estudiar la diseminación de genes de resistencia a antibióticos (ARG) del suelo a las plantas.Los datos metagenómicos revelaron que el 91,4% de los genes de resistencia a los antibióticos se identificaron comúnmente en los cuatro compartimentos del suelo mencionados anteriormente, lo que mostró una moda continua.La secuenciación del amplicón de ARNr 16S generó 29.285 secuencias, que representan 346 especies.Al combinarla con el perfil de especies mediante la secuenciación de amplicones, se descubrió que esta diseminación era independiente de la microbiota asociada a las raíces; sin embargo, podría verse facilitada por la movilidad de elementos genéticos.Este estudio identificó el flujo de ARG y patógenos desde el suelo hacia las plantas a través de un continuo interconectado suelo-raíz.
Referencia
Wang, C., Hu, R., Strong, PJ, Zhuang, W. y Shu, L.(2020).Prevalencia de genes de resistencia a antibióticos y patógenos bacterianos a lo largo del continuo suelo-raíces de manglar.Diario de materiales peligrosos, 408, 124985.











